| 登録情報 | データベース: PDB / ID: 5l74
|
|---|
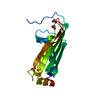
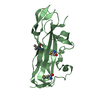
| タイトル | Plexin A2 extracellular segment domains 4-5 (PSI2-IPT2), resolution 1.36 Angstrom |
|---|
 要素 要素 | Plexin-A2 |
|---|
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  receptor (受容体) / receptor (受容体) /  axon guidance (軸索誘導) / PSI-IPT domains axon guidance (軸索誘導) / PSI-IPT domains |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cerebellar granule cell precursor tangential migration / CRMPs in Sema3A signaling / Sema3A PAK dependent Axon repulsion / SEMA3A-Plexin repulsion signaling by inhibiting Integrin adhesion / semaphorin-plexin signaling pathway involved in axon guidance / limb bud formation / semaphorin receptor complex / pharyngeal system development / semaphorin receptor activity / negative regulation of cell adhesion ...cerebellar granule cell precursor tangential migration / CRMPs in Sema3A signaling / Sema3A PAK dependent Axon repulsion / SEMA3A-Plexin repulsion signaling by inhibiting Integrin adhesion / semaphorin-plexin signaling pathway involved in axon guidance / limb bud formation / semaphorin receptor complex / pharyngeal system development / semaphorin receptor activity / negative regulation of cell adhesion / neural tube development / centrosome localization / positive regulation of axonogenesis / semaphorin-plexin signaling pathway /  somitogenesis / somitogenesis /  regulation of cell migration / regulation of cell shape / collagen-containing extracellular matrix / cell surface receptor signaling pathway / identical protein binding / regulation of cell migration / regulation of cell shape / collagen-containing extracellular matrix / cell surface receptor signaling pathway / identical protein binding /  細胞膜類似検索 - 分子機能 細胞膜類似検索 - 分子機能 Plexin-A2, sema domain /  Plexin, TIG domain 2 / TIG domain found in plexin / Plexin, TIG domain 2 / TIG domain found in plexin /  Plexin, TIG domain 1 / TIG domain / Plexin, TIG domain 1 / TIG domain /  Plexin, cytoplasmic RhoGTPase-binding domain / Plexin cytoplasmic RhoGTPase-binding domain / Plexin, cytoplasmic RhoGTPase-binding domain / Plexin cytoplasmic RhoGTPase-binding domain /  Plexin, cytoplasmic RasGAP domain / Plexin cytoplasmic RasGAP domain / Plexin, cytoplasmic RasGAP domain / Plexin cytoplasmic RasGAP domain /  Plexin family ...Plexin-A2, sema domain / Plexin family ...Plexin-A2, sema domain /  Plexin, TIG domain 2 / TIG domain found in plexin / Plexin, TIG domain 2 / TIG domain found in plexin /  Plexin, TIG domain 1 / TIG domain / Plexin, TIG domain 1 / TIG domain /  Plexin, cytoplasmic RhoGTPase-binding domain / Plexin cytoplasmic RhoGTPase-binding domain / Plexin, cytoplasmic RhoGTPase-binding domain / Plexin cytoplasmic RhoGTPase-binding domain /  Plexin, cytoplasmic RasGAP domain / Plexin cytoplasmic RasGAP domain / Plexin, cytoplasmic RasGAP domain / Plexin cytoplasmic RasGAP domain /  Plexin family / Plexin repeat / Plexin repeat / Plexin family / Plexin repeat / Plexin repeat /  Sema domain / Sema domain /  semaphorin domain / semaphorin domain /  Sema domain / Sema domain superfamily / Sema domain profile. / IPT/TIG domain / ig-like, plexins, transcription factors / Rho GTPase activation protein / PSI domain / domain found in Plexins, Semaphorins and Integrins / IPT domain / Immunoglobulin E-set / WD40/YVTN repeat-like-containing domain superfamily / Immunoglobulin-like fold類似検索 - ドメイン・相同性 Sema domain / Sema domain superfamily / Sema domain profile. / IPT/TIG domain / ig-like, plexins, transcription factors / Rho GTPase activation protein / PSI domain / domain found in Plexins, Semaphorins and Integrins / IPT domain / Immunoglobulin E-set / WD40/YVTN repeat-like-containing domain superfamily / Immunoglobulin-like fold類似検索 - ドメイン・相同性 |
|---|
| 生物種 |    Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.36 Å 分子置換 / 解像度: 1.36 Å |
|---|
 データ登録者 データ登録者 | Kong, Y. / Janssen, B.J.C. / Malinauskas, T. / Vangoor, V.R. / Coles, C.H. / Kaufmann, R. / Ni, T. / Gilbert, R.J.C. / Padilla-Parra, S. / Pasterkamp, R.J. / Jones, E.Y. |
|---|
| 資金援助 |  英国, 3件 英国, 3件 | 組織 | 認可番号 | 国 |
|---|
| Cancer Research UK | A10976 |  英国 英国 | | Medical Research Council (United Kingdom) | G9900061 |  英国 英国 | | Wellcome Trust | 090532/Z/09/Z |  英国 英国 |
|
|---|
 引用 引用 |  ジャーナル: Neuron / 年: 2016 ジャーナル: Neuron / 年: 2016
タイトル: Structural Basis for Plexin Activation and Regulation.
著者: Kong, Y. / Janssen, B.J. / Malinauskas, T. / Vangoor, V.R. / Coles, C.H. / Kaufmann, R. / Ni, T. / Gilbert, R.J. / Padilla-Parra, S. / Pasterkamp, R.J. / Jones, E.Y. |
|---|
| 履歴 | | 登録 | 2016年6月1日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年3月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年8月30日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.2 | 2019年10月16日 | Group: Data collection / カテゴリ: reflns_shell |
|---|
| 改定 1.3 | 2020年7月29日 | Group: Data collection / Derived calculations / Structure summary
カテゴリ: chem_comp / entity ...chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / pdbx_struct_conn_angle / struct_conn / struct_site / struct_site_gen
Item: _chem_comp.name / _chem_comp.type ..._chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード SIGNALING PROTEIN /
SIGNALING PROTEIN /  receptor (受容体) /
receptor (受容体) /  axon guidance (軸索誘導) / PSI-IPT domains
axon guidance (軸索誘導) / PSI-IPT domains 機能・相同性情報
機能・相同性情報 somitogenesis /
somitogenesis /  regulation of cell migration / regulation of cell shape / collagen-containing extracellular matrix / cell surface receptor signaling pathway / identical protein binding /
regulation of cell migration / regulation of cell shape / collagen-containing extracellular matrix / cell surface receptor signaling pathway / identical protein binding /  細胞膜
細胞膜
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.36 Å
分子置換 / 解像度: 1.36 Å  データ登録者
データ登録者 英国, 3件
英国, 3件  引用
引用 ジャーナル: Neuron / 年: 2016
ジャーナル: Neuron / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5l74.cif.gz
5l74.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5l74.ent.gz
pdb5l74.ent.gz PDB形式
PDB形式 5l74.json.gz
5l74.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/l7/5l74
https://data.pdbj.org/pub/pdb/validation_reports/l7/5l74 ftp://data.pdbj.org/pub/pdb/validation_reports/l7/5l74
ftp://data.pdbj.org/pub/pdb/validation_reports/l7/5l74 リンク
リンク 集合体
集合体
 要素
要素
 Mus musculus (ハツカネズミ) / 遺伝子: Plxna2, Kiaa0463 / 細胞株 (発現宿主): HEK293S / 発現宿主:
Mus musculus (ハツカネズミ) / 遺伝子: Plxna2, Kiaa0463 / 細胞株 (発現宿主): HEK293S / 発現宿主: 
 Homo sapiens (ヒト) / 参照: UniProt: P70207
Homo sapiens (ヒト) / 参照: UniProt: P70207 N-アセチルグルコサミン
N-アセチルグルコサミン 塩化物
塩化物 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I04 / 波長: 1.0163 Å
/ ビームライン: I04 / 波長: 1.0163 Å : 1.0163 Å / 相対比: 1
: 1.0163 Å / 相対比: 1  解析
解析 :
:  分子置換 / 解像度: 1.36→52.092 Å / SU ML: 0.13 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 22.79
分子置換 / 解像度: 1.36→52.092 Å / SU ML: 0.13 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 22.79  ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj









