+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4pnm | ||||||
|---|---|---|---|---|---|---|---|
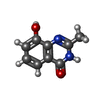
| タイトル | Crystal Structure of human Tankyrase 2 in complex with Nu1025. | ||||||
 要素 要素 | Tankyrase-2 | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / poly(ADP-ribosylation) polymerase (PARP) TRANSFERASE (転移酵素) / poly(ADP-ribosylation) polymerase (PARP) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報XAV939 stabilizes AXIN /  NAD+ ADP-ribosyltransferase / protein localization to chromosome, telomeric region / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein poly-ADP-ribosylation / NAD+ ADP-ribosyltransferase / protein localization to chromosome, telomeric region / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein poly-ADP-ribosylation /  中心体マトリックス / NAD+-protein ADP-ribosyltransferase activity / positive regulation of telomere capping / 中心体マトリックス / NAD+-protein ADP-ribosyltransferase activity / positive regulation of telomere capping /  NAD+ ADP-ribosyltransferase activity ...XAV939 stabilizes AXIN / NAD+ ADP-ribosyltransferase activity ...XAV939 stabilizes AXIN /  NAD+ ADP-ribosyltransferase / protein localization to chromosome, telomeric region / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein poly-ADP-ribosylation / NAD+ ADP-ribosyltransferase / protein localization to chromosome, telomeric region / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein poly-ADP-ribosylation /  中心体マトリックス / NAD+-protein ADP-ribosyltransferase activity / positive regulation of telomere capping / 中心体マトリックス / NAD+-protein ADP-ribosyltransferase activity / positive regulation of telomere capping /  NAD+ ADP-ribosyltransferase activity / NAD+ ADP-ribosyltransferase activity /  転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / positive regulation of telomere maintenance via telomerase / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / positive regulation of telomere maintenance via telomerase /  nucleotidyltransferase activity / TCF dependent signaling in response to WNT / Degradation of AXIN / nucleotidyltransferase activity / TCF dependent signaling in response to WNT / Degradation of AXIN /  Wntシグナル経路 / Regulation of PTEN stability and activity / protein polyubiquitination / positive regulation of canonical Wnt signaling pathway / Wntシグナル経路 / Regulation of PTEN stability and activity / protein polyubiquitination / positive regulation of canonical Wnt signaling pathway /  核膜 / 核膜 /  chromosome, telomeric region / Ub-specific processing proteases / chromosome, telomeric region / Ub-specific processing proteases /  ゴルジ体 / perinuclear region of cytoplasm / ゴルジ体 / perinuclear region of cytoplasm /  enzyme binding / enzyme binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.19 Å シンクロトロン / 解像度: 2.19 Å | ||||||
 データ登録者 データ登録者 | Qiu, W. / Lam, R. / Romanov, V. / Gordon, R. / Gebremeskel, S. / Vodsedalek, J. / Thompson, C. / Beletskaya, I. / Battaile, K.P. / Pai, E.F. / Chirgadze, N.Y. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2014 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2014タイトル: Insights into the binding of PARP inhibitors to the catalytic domain of human tankyrase-2. 著者: Qiu, W. / Lam, R. / Voytyuk, O. / Romanov, V. / Gordon, R. / Gebremeskel, S. / Vodsedalek, J. / Thompson, C. / Beletskaya, I. / Battaile, K.P. / Pai, E.F. / Rottapel, R. / Chirgadze, N.Y. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4pnm.cif.gz 4pnm.cif.gz | 185.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4pnm.ent.gz pdb4pnm.ent.gz | 145.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4pnm.json.gz 4pnm.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pn/4pnm https://data.pdbj.org/pub/pdb/validation_reports/pn/4pnm ftp://data.pdbj.org/pub/pdb/validation_reports/pn/4pnm ftp://data.pdbj.org/pub/pdb/validation_reports/pn/4pnm | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  4pmlC  4pnlC  4pnnC  4pnqC  4pnrC  4pnsC  4pntC  4tjuC  4tjwC  4tjyC  4tk0C  4tk5C  4tkfC  4tkgC  4tkiC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | monomer |
- 要素
要素
| #1: タンパク質 |  / TANK2 / ADP-ribosyltransferase diphtheria toxin-like 6 / ARTD6 / Poly [ADP-ribose] polymerase 5B / ...TANK2 / ADP-ribosyltransferase diphtheria toxin-like 6 / ARTD6 / Poly [ADP-ribose] polymerase 5B / TNKS-2 / TRF1-interacting ankyrin-related ADP-ribose polymerase 2 / Tankyrase II / Tankyrase-like protein / Tankyrase-related protein / TANK2 / ADP-ribosyltransferase diphtheria toxin-like 6 / ARTD6 / Poly [ADP-ribose] polymerase 5B / ...TANK2 / ADP-ribosyltransferase diphtheria toxin-like 6 / ARTD6 / Poly [ADP-ribose] polymerase 5B / TNKS-2 / TRF1-interacting ankyrin-related ADP-ribose polymerase 2 / Tankyrase II / Tankyrase-like protein / Tankyrase-related protein分子量: 25986.291 Da / 分子数: 4 / 断片: PARP, catalytic domain / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: TNKS2, PARP5B, TANK2, TNKL / 発現宿主: Homo sapiens (ヒト) / 遺伝子: TNKS2, PARP5B, TANK2, TNKL / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: Q9H2K2, Escherichia coli (大腸菌) / 参照: UniProt: Q9H2K2,  NAD+ ADP-ribosyltransferase NAD+ ADP-ribosyltransferase#2: 化合物 | ChemComp-ZN / #3: 化合物 | ChemComp-NU1 / #4: 化合物 |  エチレングリコール エチレングリコール#5: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.14 Å3/Da / 溶媒含有率: 42.5 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: 0.2M NaCl, 0.1M Sodium, Hepes buffer at pH7.5, 12-15% iso-propanol |
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 17-ID / 波長: 1 Å / ビームライン: 17-ID / 波長: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2010年6月19日 / 詳細: mirrors | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | モノクロメーター: GRAPHITE / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.19→79.25 Å / Num. obs: 46699 / % possible obs: 99.9 % / 冗長度: 7.05 % / Biso Wilson estimate: 36.76 Å2 / Rmerge(I) obs: 0.099 / Net I/σ(I): 13.44 / Num. measured all: 329620 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1 / Rejects: 0
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.19→76.92 Å / Cor.coef. Fo:Fc: 0.8968 / Cor.coef. Fo:Fc free: 0.8814 / SU R Cruickshank DPI: 0.272 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.282 / SU Rfree Blow DPI: 0.207 / SU Rfree Cruickshank DPI: 0.206
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 123.09 Å2 / Biso mean: 43.63 Å2 / Biso min: 20.47 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.291 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.19→76.92 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.19→2.25 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj