+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3ll5 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of T. acidophilum isopentenyl phosphate kinase product complex | ||||||
 要素 要素 | Gamma-glutamyl kinase related protein | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / alternate mevalonate pathway / isopentenyl phsophate kinase / alpha-beta-alpha sandwich fold / product complex / TRANSFERASE (転移酵素) / alternate mevalonate pathway / isopentenyl phsophate kinase / alpha-beta-alpha sandwich fold / product complex /  Kinase (キナーゼ) Kinase (キナーゼ) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 isopentenyl phosphate kinase / isopentenyl phosphate kinase /  isopentenyl phosphate kinase activity / isoprenoid biosynthetic process / isopentenyl phosphate kinase activity / isoprenoid biosynthetic process /  kinase activity / kinase activity /  リン酸化 / リン酸化 /  ATP binding ATP binding類似検索 - 分子機能 | ||||||
| 生物種 |    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.987 Å 分子置換 / 解像度: 1.987 Å | ||||||
 データ登録者 データ登録者 | Mabanglo, M.F. / Hill, C.P. | ||||||
 引用 引用 |  ジャーナル: Acs Chem.Biol. / 年: 2010 ジャーナル: Acs Chem.Biol. / 年: 2010タイトル: X-ray structures of isopentenyl phosphate kinase. 著者: Mabanglo, M.F. / Schubert, H.L. / Chen, M. / Hill, C.P. / Poulter, C.D. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3ll5.cif.gz 3ll5.cif.gz | 203.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3ll5.ent.gz pdb3ll5.ent.gz | 164.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3ll5.json.gz 3ll5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ll/3ll5 https://data.pdbj.org/pub/pdb/validation_reports/ll/3ll5 ftp://data.pdbj.org/pub/pdb/validation_reports/ll/3ll5 ftp://data.pdbj.org/pub/pdb/validation_reports/ll/3ll5 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 4分子 ABCD
| #1: タンパク質 | 分子量: 28107.473 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)    Thermoplasma acidophilum (好酸性) / 株: DSM1728 / 遺伝子: Ta0103 / プラスミド: pET151/D-TOPO / 発現宿主: Thermoplasma acidophilum (好酸性) / 株: DSM1728 / 遺伝子: Ta0103 / プラスミド: pET151/D-TOPO / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) Codon Plus RIL / 参照: UniProt: Q9HLX1 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) Codon Plus RIL / 参照: UniProt: Q9HLX1 |
|---|
-非ポリマー , 5種, 248分子 
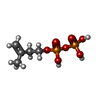

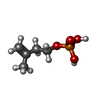





| #2: 化合物 |  アデノシン二リン酸 アデノシン二リン酸#3: 化合物 |  イソペンテニル二リン酸 イソペンテニル二リン酸#4: 化合物 |  アデノシン三リン酸 アデノシン三リン酸#5: 化合物 | #6: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.21 Å3/Da / 溶媒含有率: 44.28 % |
|---|---|
結晶化 | 温度: 274 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 6 詳細: 0.1 M PCB buffer (2:1:2 molar ratio of sodium propionate, sodium cacodylate, Bis-Tris propane, pH 6.0 containing 25% PEG1500, VAPOR DIFFUSION, SITTING DROP, temperature 274K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL11-1 / 波長: 0.97887 Å / ビームライン: BL11-1 / 波長: 0.97887 Å |
| 検出器 | タイプ: MARMOSAIC 325 mm CCD / 検出器: CCD / 日付: 2009年2月27日 詳細: Flat mirror (vertical focusing); single crystal Si(111) bent monochromator (horizontal focusing) |
| 放射 | モノクロメーター: Side scattering bent cube-root I-beam single crystal; asymmetric cut 4.965 degs プロトコル: MAD / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.97887 Å / 相対比: 1 : 0.97887 Å / 相対比: 1 |
| 反射 | 解像度: 1.99→50 Å / Num. obs: 65826 / 冗長度: 1.8 % / Rsym value: 0.062 / Net I/σ(I): 9.48 |
| 反射 シェル | 解像度: 1.99→2.06 Å / 冗長度: 1.7 % / Mean I/σ(I) obs: 3.3 / Rsym value: 0.062 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: T. acidophilum isopentenyl phosphate kinase solved by single wavelength anomalous diffraction 解像度: 1.987→38.292 Å / SU ML: 0.26 / σ(F): 1.34 / 位相誤差: 23.97 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 29.941 Å2 / ksol: 0.425 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.987→38.292 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj



