+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3jcc | ||||||
|---|---|---|---|---|---|---|---|
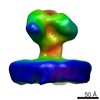
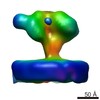
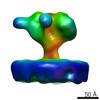
| タイトル | Structure of Simian Immunodeficiency Virus Envelope Spikes bound with CD4 and Monoclonal Antibody 36D5 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) / VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) /  Cryoelectron tomography / Cryoelectron tomography /  immunology (免疫学) / immunology (免疫学) /  AIDS (後天性免疫不全症候群) / AIDS (後天性免疫不全症候群) /  HIV (ヒト免疫不全ウイルス) / HIV (ヒト免疫不全ウイルス) /  VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報helper T cell enhancement of adaptive immune response /  interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell / interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell /  T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation ...helper T cell enhancement of adaptive immune response / T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation ...helper T cell enhancement of adaptive immune response /  interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell / interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell /  T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation / Alpha-defensins / positive regulation of kinase activity / T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation / Alpha-defensins / positive regulation of kinase activity /  regulation of T cell activation / regulation of T cell activation /  T cell receptor complex / extracellular matrix structural constituent / Other interleukin signaling / T cell receptor complex / extracellular matrix structural constituent / Other interleukin signaling /  enzyme-linked receptor protein signaling pathway / Translocation of ZAP-70 to Immunological synapse / Phosphorylation of CD3 and TCR zeta chains / regulation of calcium ion transport / macrophage differentiation / Generation of second messenger molecules / T cell differentiation / PD-1 signaling / positive regulation of protein kinase activity / Binding and entry of HIV virion / enzyme-linked receptor protein signaling pathway / Translocation of ZAP-70 to Immunological synapse / Phosphorylation of CD3 and TCR zeta chains / regulation of calcium ion transport / macrophage differentiation / Generation of second messenger molecules / T cell differentiation / PD-1 signaling / positive regulation of protein kinase activity / Binding and entry of HIV virion /  coreceptor activity / positive regulation of calcium-mediated signaling / coreceptor activity / positive regulation of calcium-mediated signaling /  protein tyrosine kinase binding / positive regulation of interleukin-2 production / protein tyrosine kinase binding / positive regulation of interleukin-2 production /  T細胞 / Vpu mediated degradation of CD4 / calcium-mediated signaling / clathrin-coated endocytic vesicle membrane / T細胞 / Vpu mediated degradation of CD4 / calcium-mediated signaling / clathrin-coated endocytic vesicle membrane /  cell surface receptor protein tyrosine kinase signaling pathway / positive regulation of T cell activation / positive regulation of peptidyl-tyrosine phosphorylation / transmembrane signaling receptor activity / Cargo recognition for clathrin-mediated endocytosis / Downstream TCR signaling / virus receptor activity / cell surface receptor protein tyrosine kinase signaling pathway / positive regulation of T cell activation / positive regulation of peptidyl-tyrosine phosphorylation / transmembrane signaling receptor activity / Cargo recognition for clathrin-mediated endocytosis / Downstream TCR signaling / virus receptor activity /  signaling receptor activity / signaling receptor activity /  Clathrin-mediated endocytosis / MHC class II protein complex binding / positive regulation of canonical NF-kappaB signal transduction / defense response to Gram-negative bacterium / positive regulation of MAPK cascade / Clathrin-mediated endocytosis / MHC class II protein complex binding / positive regulation of canonical NF-kappaB signal transduction / defense response to Gram-negative bacterium / positive regulation of MAPK cascade /  獲得免疫系 / positive regulation of viral entry into host cell / positive regulation of ERK1 and ERK2 cascade / cell surface receptor signaling pathway / 獲得免疫系 / positive regulation of viral entry into host cell / positive regulation of ERK1 and ERK2 cascade / cell surface receptor signaling pathway /  エンドソーム / エンドソーム /  細胞接着 / 細胞接着 /  免疫応答 / positive regulation of protein phosphorylation / 免疫応答 / positive regulation of protein phosphorylation /  脂質ラフト / 脂質ラフト /  小胞体 / external side of plasma membrane / 小胞体 / external side of plasma membrane /  エンベロープ (ウイルス) / エンベロープ (ウイルス) /  lipid binding / endoplasmic reticulum membrane / lipid binding / endoplasmic reticulum membrane /  protein kinase binding / positive regulation of DNA-templated transcription / protein kinase binding / positive regulation of DNA-templated transcription /  enzyme binding / enzyme binding /  シグナル伝達 / protein homodimerization activity / zinc ion binding / identical protein binding / シグナル伝達 / protein homodimerization activity / zinc ion binding / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス)  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  電子線トモグラフィー法 / 電子線トモグラフィー法 /  クライオ電子顕微鏡法 クライオ電子顕微鏡法 | ||||||
 データ登録者 データ登録者 | Hu, G. / Liu, J. / Roux, K. / Taylor, K.A. | ||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2017 ジャーナル: J Virol / 年: 2017タイトル: Structure of Simian Immunodeficiency Virus Envelope Spikes Bound with CD4 and Monoclonal Antibody 36D5. 著者: Guiqing Hu / Jun Liu / Kenneth H Roux / Kenneth A Taylor /  要旨: The human immunodeficiency virus type 1 (HIV-1)/simian immunodeficiency virus (SIV) envelope spike (Env) mediates viral entry into host cells. The V3 loop of the gp120 component of the Env trimer ...The human immunodeficiency virus type 1 (HIV-1)/simian immunodeficiency virus (SIV) envelope spike (Env) mediates viral entry into host cells. The V3 loop of the gp120 component of the Env trimer contributes to the coreceptor binding site and is a target for neutralizing antibodies. We used cryo-electron tomography to visualize the binding of CD4 and the V3 loop monoclonal antibody (MAb) 36D5 to gp120 of the SIV Env trimer. Our results show that 36D5 binds gp120 at the base of the V3 loop and suggest that the antibody exerts its neutralization effect by blocking the coreceptor binding site. The antibody does this without altering the dynamics of the spike motion between closed and open states when CD4 is bound. The interaction between 36D5 and SIV gp120 is similar to the interaction between some broadly neutralizing anti-V3 loop antibodies and HIV-1 gp120. Two conformations of gp120 bound with CD4 are revealed, suggesting an intrinsic dynamic nature of the liganded Env trimer. CD4 binding substantially increases the binding of 36D5 to gp120 in the intact Env trimer, consistent with CD4-induced changes in the conformation of gp120 and the antibody binding site. Binding by MAb 36D5 does not substantially alter the proportions of the two CD4-bound conformations. The position of MAb 36D5 at the V3 base changes little between conformations, indicating that the V3 base serves as a pivot point during the transition between these two states. Glycoprotein spikes on the surfaces of SIV and HIV are the sole targets available to the immune system for antibody neutralization. Spikes evade the immune system by a combination of a thick layer of polysaccharide on the surface (the glycan shield) and movement between spike domains that masks the epitope conformation. Using SIV virions whose spikes were "decorated" with the primary cellular receptor (CD4) and an antibody (36D5) at part of the coreceptor binding site, we visualized multiple conformations trapped by the rapid freezing step, which were separated using statistical analysis. Our results show that the CD4-induced conformational dynamics of the spike enhances binding of the antibody. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3jcc.cif.gz 3jcc.cif.gz | 372.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3jcc.ent.gz pdb3jcc.ent.gz | 306.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3jcc.json.gz 3jcc.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jc/3jcc https://data.pdbj.org/pub/pdb/validation_reports/jc/3jcc ftp://data.pdbj.org/pub/pdb/validation_reports/jc/3jcc ftp://data.pdbj.org/pub/pdb/validation_reports/jc/3jcc | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  Gp120 (HIV) Gp120 (HIV)分子量: 50130.594 Da / 分子数: 3 / 断片: SEE REMARK 999 / 由来タイプ: 天然 由来: (天然)   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス)株: SIV239/251tail/Supt-CCR5 CL.30 / 参照: UniProt: A0A3B6UDT5*PLUS #2: 抗体 | | 分子量: 22291.643 Da / 分子数: 1 / 断片: Fab / 由来タイプ: 天然 / 由来: (天然)   Homo sapiens (ヒト) Homo sapiens (ヒト)#3: 抗体 | | 分子量: 25115.289 Da / 分子数: 1 / 断片: Fab / 由来タイプ: 天然 / 由来: (天然)   Homo sapiens (ヒト) Homo sapiens (ヒト)#4: タンパク質 | | 分子量: 19442.045 Da / 分子数: 1 / 断片: UNP residues 26-200 / 由来タイプ: 天然 / 由来: (天然)   Homo sapiens (ヒト) / 参照: UniProt: P01730 Homo sapiens (ヒト) / 参照: UniProt: P01730配列の詳細 | ENVELOPE GLYCOPROTE | |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: CELL / 3次元再構成法:  電子線トモグラフィー法 電子線トモグラフィー法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ウイルスについての詳細 | 中空か: NO / エンベロープを持つか: YES / ホストのカテゴリ: VERTEBRATES / 単離: STRAIN / タイプ: VIRION | ||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||
急速凍結 | 装置: HOMEMADE PLUNGER / 凍結剤: ETHANE / 詳細: Plunged into liquid ethane. |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI POLARA 300 / 日付: 2008年8月16日 |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 31000 X / 最大 デフォーカス(公称値): 5000 nm / 最小 デフォーカス(公称値): 4000 nm Bright-field microscopy / 倍率(公称値): 31000 X / 最大 デフォーカス(公称値): 5000 nm / 最小 デフォーカス(公称値): 4000 nm |
| 試料ホルダ | 試料ホルダーモデル: OTHER / 資料ホルダタイプ: unidentified / 傾斜角・最大: 65 ° / 傾斜角・最小: -65 ° |
| 撮影 | 電子線照射量: 100 e/Å2 フィルム・検出器のモデル: TVIPS TEMCAM-F415 (4k x 4k) |
- 解析
解析
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
3次元再構成 | 粒子像の数: 1796 / ピクセルサイズ(公称値): 5.7 Å / ピクセルサイズ(実測値): 5.7 Å / 詳細: (Subtomogram Averaging--Applied Symmetry: C1) / 対称性のタイプ: POINT | ||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL | ||||||||||||
| 原子モデル構築 | PDB-ID: 4NCO | ||||||||||||
| 精密化ステップ | サイクル: LAST
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj




















