+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2vxd | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | The structure of the C-terminal domain of Nucleophosmin | ||||||
 要素 要素 | NUCLEOPHOSMIN NPM1 NPM1 | ||||||
 キーワード キーワード |  NUCLEAR PROTEIN / NUCLEAR PROTEIN /  CHROMOSOMAL REARRANGEMENT / CHROMOSOMAL REARRANGEMENT /  PROTO-ONCOGENE (がん遺伝子) / PROTO-ONCOGENE (がん遺伝子) /  PHOSPHOPROTEIN / PHOSPHOPROTEIN /  ALTERNATIVE SPLICING (選択的スプライシング) / AML / ALTERNATIVE SPLICING (選択的スプライシング) / AML /  NUCLEUS (細胞核) / NUCLEUS (細胞核) /  NUCLEOLUS (核小体) / RNA-BINDING / NUCLEOLUS (核小体) / RNA-BINDING /  ACETYLATION (アセチル化) ACETYLATION (アセチル化) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of eIF2 alpha phosphorylation by dsRNA / regulation of mRNA stability involved in cellular response to UV /  regulation of endoribonuclease activity / positive regulation of cell cycle G2/M phase transition / negative regulation of centrosome duplication / regulation of endoribonuclease activity / positive regulation of cell cycle G2/M phase transition / negative regulation of centrosome duplication /  regulation of endodeoxyribonuclease activity / positive regulation of centrosome duplication / regulation of centriole replication / regulation of endodeoxyribonuclease activity / positive regulation of centrosome duplication / regulation of centriole replication /  核小体 / positive regulation of protein localization to nucleolus ...regulation of eIF2 alpha phosphorylation by dsRNA / regulation of mRNA stability involved in cellular response to UV / 核小体 / positive regulation of protein localization to nucleolus ...regulation of eIF2 alpha phosphorylation by dsRNA / regulation of mRNA stability involved in cellular response to UV /  regulation of endoribonuclease activity / positive regulation of cell cycle G2/M phase transition / negative regulation of centrosome duplication / regulation of endoribonuclease activity / positive regulation of cell cycle G2/M phase transition / negative regulation of centrosome duplication /  regulation of endodeoxyribonuclease activity / positive regulation of centrosome duplication / regulation of centriole replication / regulation of endodeoxyribonuclease activity / positive regulation of centrosome duplication / regulation of centriole replication /  核小体 / positive regulation of protein localization to nucleolus / TFAP2A acts as a transcriptional repressor during retinoic acid induced cell differentiation / negative regulation of protein kinase activity by regulation of protein phosphorylation / SARS-CoV-1-host interactions / regulation of centrosome duplication / Tat protein binding / ALK mutants bind TKIs / spindle pole centrosome / cell volume homeostasis / Nuclear import of Rev protein / : / 核小体 / positive regulation of protein localization to nucleolus / TFAP2A acts as a transcriptional repressor during retinoic acid induced cell differentiation / negative regulation of protein kinase activity by regulation of protein phosphorylation / SARS-CoV-1-host interactions / regulation of centrosome duplication / Tat protein binding / ALK mutants bind TKIs / spindle pole centrosome / cell volume homeostasis / Nuclear import of Rev protein / : /  centrosome cycle / nucleocytoplasmic transport / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / centrosome cycle / nucleocytoplasmic transport / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain /  protein kinase inhibitor activity / protein kinase inhibitor activity /  ribosomal large subunit binding / negative regulation of mRNA splicing, via spliceosome / ribosomal large subunit binding / negative regulation of mRNA splicing, via spliceosome /  ribosomal small subunit export from nucleus / ribosomal small subunit export from nucleus /  ribosomal small subunit binding / Signaling by ALK fusions and activated point mutants / ribosomal small subunit binding / Signaling by ALK fusions and activated point mutants /  ribosomal large subunit export from nucleus / ribosomal large subunit export from nucleus /  NF-kappaB binding / Nuclear events stimulated by ALK signaling in cancer / core promoter sequence-specific DNA binding / Deposition of new CENPA-containing nucleosomes at the centromere / NF-kappaB binding / Nuclear events stimulated by ALK signaling in cancer / core promoter sequence-specific DNA binding / Deposition of new CENPA-containing nucleosomes at the centromere /  ribosomal large subunit biogenesis / ribosomal large subunit biogenesis /  ribosome assembly / molecular condensate scaffold activity / SUMOylation of transcription cofactors / positive regulation of translation / positive regulation of protein ubiquitination / ribosome assembly / molecular condensate scaffold activity / SUMOylation of transcription cofactors / positive regulation of translation / positive regulation of protein ubiquitination /  regulation of cell growth / regulation of cell growth /  デオキシリボ核酸 / デオキシリボ核酸 /  intracellular protein transport / PKR-mediated signaling / intracellular protein transport / PKR-mediated signaling /  protein localization / protein localization /  ribosomal small subunit biogenesis / ribosomal small subunit biogenesis /  nucleosome assembly / cellular response to UV / unfolded protein binding / nucleosome assembly / cellular response to UV / unfolded protein binding /  細胞老化 / large ribosomal subunit / small ribosomal subunit / positive regulation of NF-kappaB transcription factor activity / 細胞老化 / large ribosomal subunit / small ribosomal subunit / positive regulation of NF-kappaB transcription factor activity /  histone binding / DNA-binding transcription factor binding / histone binding / DNA-binding transcription factor binding /  transcription coactivator activity / transcription coactivator activity /  rRNA binding / protein stabilization / nuclear speck / rRNA binding / protein stabilization / nuclear speck /  クロマチンリモデリング / クロマチンリモデリング /  ribonucleoprotein complex / negative regulation of cell population proliferation / ribonucleoprotein complex / negative regulation of cell population proliferation /  focal adhesion / focal adhesion /  DNA修復 / DNA修復 /  中心体 / 中心体 /  chromatin binding / positive regulation of cell population proliferation / chromatin binding / positive regulation of cell population proliferation /  核小体 / negative regulation of apoptotic process / 核小体 / negative regulation of apoptotic process /  protein kinase binding / positive regulation of DNA-templated transcription / protein kinase binding / positive regulation of DNA-templated transcription /  シグナル伝達 / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / protein-containing complex / シグナル伝達 / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / protein-containing complex /  RNA binding / RNA binding /  核質 / 核質 /  生体膜 / 生体膜 /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | ||||||
| 手法 |  溶液NMR 溶液NMR | ||||||
 データ登録者 データ登録者 | Bycroft, M. / Grummitt, C.G. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2008 ジャーナル: J.Biol.Chem. / 年: 2008タイトル: Structural Consequences of Nucleophosmin Mutations in Acute Myeloid Leukemia. 著者: Grummitt, C.G. / Townsley, F.M. / Johnson, C.M. / Warren, A.J. / Bycroft, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2vxd.cif.gz 2vxd.cif.gz | 334.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2vxd.ent.gz pdb2vxd.ent.gz | 291.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2vxd.json.gz 2vxd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/vx/2vxd https://data.pdbj.org/pub/pdb/validation_reports/vx/2vxd ftp://data.pdbj.org/pub/pdb/validation_reports/vx/2vxd ftp://data.pdbj.org/pub/pdb/validation_reports/vx/2vxd | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 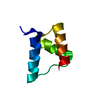
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 |  NPM1 / NPM / NUCLEOLAR PHOSPHOPROTEIN B23 / NUMATRIN / NUCLEOLAR PROTEIN NO38 NPM1 / NPM / NUCLEOLAR PHOSPHOPROTEIN B23 / NUMATRIN / NUCLEOLAR PROTEIN NO38分子量: 6183.121 Da / 分子数: 1 / 断片: C-TERMINAL DOMAIN, RESIDUES 214-265 / 由来タイプ: 組換発現 / 由来: (組換発現)   HOMO SAPIENS (ヒト) / 発現宿主: HOMO SAPIENS (ヒト) / 発現宿主:   ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P06748 ESCHERICHIA COLI (大腸菌) / 参照: UniProt: P06748 |
|---|---|
| 配列の詳細 | TWO EXTRA GLYCINE RESIDUES AT THE N-TERMINUS |
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR |
|---|---|
| NMR実験の詳細 | Text: NONE |
- 試料調製
試料調製
| 詳細 | 内容: 10% WATER/90% D2O |
|---|---|
| 試料状態 | イオン強度: 150 mM / pH: 6.5 / 圧: 1.0 atm / 温度: 298.0 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | ソフトェア番号: 1 詳細: REFINEMENT DETAILS CAN BE FOUND IN THE JRNL CITATION ABOVE | |||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATION 計算したコンフォーマーの数: 150 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj







