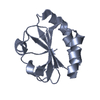
登録情報 データベース : PDB / ID : 2izxタイトル Molecular Basis of AKAP Specificity for PKA Regulatory Subunits AKAP-IS CAMP-DEPENDENT PROTEIN KINASE TYPE II-ALPHA REGULATORY SUBUNIT キーワード / / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 HOMO SAPIENS (ヒト)SYNTHETIC CONSTRUCT (人工物) 手法 / / / 解像度 : 1.3 Å データ登録者 Gold, M.G. / Lygren, B. / Dokurno, P. / Hoshi, N. / McConnachie, G. / Tasken, K. / Carlson, C.R. / Scott, J.D. / Barford, D. ジャーナル : Mol.Cell / 年 : 2006タイトル : Molecular Basis of Akap Specificity for Pka Regulatory Subunits.著者 : Gold, M.G. / Lygren, B. / Dokurno, P. / Hoshi, N. / Mcconnachie, G. / Tasken, K. / Carlson, C.R. / Scott, J.D. / Barford, D. 履歴 登録 2006年7月27日 登録サイト / 処理サイト 改定 1.0 2006年11月8日 Provider / タイプ 改定 1.1 2017年2月8日 Group Derived calculations / Non-polymer description ... Derived calculations / Non-polymer description / Other / Source and taxonomy / Structure summary / Version format compliance
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSFERASE (転移酵素) / CAMP-BINDING /
TRANSFERASE (転移酵素) / CAMP-BINDING /  PHOSPHORYLATION (リン酸化) / NUCLEOTIDE-BINDING / PKA /
PHOSPHORYLATION (リン酸化) / NUCLEOTIDE-BINDING / PKA /  CAMP (CAMP) /
CAMP (CAMP) /  AKAP /
AKAP /  ANCHOR (錨) /
ANCHOR (錨) /  KINASE (キナーゼ) /
KINASE (キナーゼ) /  ACETYLATION (アセチル化)
ACETYLATION (アセチル化) 機能・相同性情報
機能・相同性情報 cAMP-dependent protein kinase regulator activity / negative regulation of cAMP-dependent protein kinase activity / nucleotide-activated protein kinase complex /
cAMP-dependent protein kinase regulator activity / negative regulation of cAMP-dependent protein kinase activity / nucleotide-activated protein kinase complex /  cAMP-dependent protein kinase inhibitor activity / CREB1 phosphorylation through the activation of Adenylate Cyclase / PKA activation / ciliary base /
cAMP-dependent protein kinase inhibitor activity / CREB1 phosphorylation through the activation of Adenylate Cyclase / PKA activation / ciliary base /  cAMP-dependent protein kinase complex / protein kinase A catalytic subunit binding ...ROBO receptors bind AKAP5 /
cAMP-dependent protein kinase complex / protein kinase A catalytic subunit binding ...ROBO receptors bind AKAP5 /  cAMP-dependent protein kinase regulator activity / negative regulation of cAMP-dependent protein kinase activity / nucleotide-activated protein kinase complex /
cAMP-dependent protein kinase regulator activity / negative regulation of cAMP-dependent protein kinase activity / nucleotide-activated protein kinase complex /  cAMP-dependent protein kinase inhibitor activity / CREB1 phosphorylation through the activation of Adenylate Cyclase / PKA activation / ciliary base /
cAMP-dependent protein kinase inhibitor activity / CREB1 phosphorylation through the activation of Adenylate Cyclase / PKA activation / ciliary base /  cAMP-dependent protein kinase complex / protein kinase A catalytic subunit binding / plasma membrane raft / PKA activation in glucagon signalling / DARPP-32 events / Hedgehog 'off' state /
cAMP-dependent protein kinase complex / protein kinase A catalytic subunit binding / plasma membrane raft / PKA activation in glucagon signalling / DARPP-32 events / Hedgehog 'off' state /  cAMP binding / FCGR3A-mediated IL10 synthesis / Vasopressin regulates renal water homeostasis via Aquaporins / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / ADORA2B mediated anti-inflammatory cytokines production / GPER1 signaling / Factors involved in megakaryocyte development and platelet production / intracellular signal transduction / protein domain specific binding /
cAMP binding / FCGR3A-mediated IL10 synthesis / Vasopressin regulates renal water homeostasis via Aquaporins / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / ADORA2B mediated anti-inflammatory cytokines production / GPER1 signaling / Factors involved in megakaryocyte development and platelet production / intracellular signal transduction / protein domain specific binding /  focal adhesion /
focal adhesion /  中心体 /
中心体 /  ubiquitin protein ligase binding / protein-containing complex / extracellular exosome /
ubiquitin protein ligase binding / protein-containing complex / extracellular exosome /  生体膜 /
生体膜 /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.3 Å
分子置換 / 解像度: 1.3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Mol.Cell / 年: 2006
ジャーナル: Mol.Cell / 年: 2006 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2izx.cif.gz
2izx.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2izx.ent.gz
pdb2izx.ent.gz PDB形式
PDB形式 2izx.json.gz
2izx.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/iz/2izx
https://data.pdbj.org/pub/pdb/validation_reports/iz/2izx ftp://data.pdbj.org/pub/pdb/validation_reports/iz/2izx
ftp://data.pdbj.org/pub/pdb/validation_reports/iz/2izx リンク
リンク 集合体
集合体
 要素
要素 CAMP-dependent pathway / CAMP-DEPENDENT PROTEIN KINASE REGULATORY SUBUNIT II
CAMP-dependent pathway / CAMP-DEPENDENT PROTEIN KINASE REGULATORY SUBUNIT II
 HOMO SAPIENS (ヒト) / プラスミド: PGEX6P1 / 発現宿主:
HOMO SAPIENS (ヒト) / プラスミド: PGEX6P1 / 発現宿主: 
 ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: P13861,
ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: P13861,  プロテインキナーゼA
プロテインキナーゼA 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID29 / 波長: 0.97
/ ビームライン: ID29 / 波長: 0.97  : 0.97 Å / 相対比: 1
: 0.97 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj