+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ncr | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | The structure of Rhinovirus 16 when complexed with pleconaril, an antiviral compound | ||||||
 要素 要素 | (coat protein ...) x 4 | ||||||
 キーワード キーワード |  VIRUS (ウイルス) / HRV16 / VIRUS (ウイルス) / HRV16 /  rhinovirus (ライノウイルス) / rhinovirus (ライノウイルス) /  pleconaril / piconavirus / Icosahedral virus pleconaril / piconavirus / Icosahedral virus | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell /  : ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / : ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / ribonucleoside triphosphate phosphatase activity / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell /  : / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / : / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity /  DNA複製 / DNA複製 /  RNA helicase activity / induction by virus of host autophagy / RNA helicase activity / induction by virus of host autophagy /  RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity / RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /  RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / structural molecule activity / virion attachment to host cell / RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / structural molecule activity / virion attachment to host cell /  タンパク質分解 / タンパク質分解 /  RNA binding / RNA binding /  ATP binding / ATP binding /  生体膜 / 生体膜 /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||
| 生物種 |   Human rhinovirus 16 (ライノウイルス) Human rhinovirus 16 (ライノウイルス) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.7 Å 分子置換 / 解像度: 2.7 Å | ||||||
 データ登録者 データ登録者 | Zhang, Y. / Simpson, A.A. / Bator, C.M. / Chakravarty, S. / Pevear, D.C. / Skochko, G.A. / Tull, T.M. / Diana, G. / Rossmann, M.G. | ||||||
 引用 引用 |  ジャーナル: J.Virol. / 年: 2004 ジャーナル: J.Virol. / 年: 2004タイトル: Structural and virological studies of the stages of virus replication that are affected by antirhinovirus compounds 著者: Zhang, Y. / Simpson, A.A. / Ledford, R.M. / Bator, C.M. / Chakravarty, S. / Skochko, G.A. / Demenczuk, T.M. / Watanyar, A. / Pevear, D.C. / Rossmann, M.G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ncr.cif.gz 1ncr.cif.gz | 172.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ncr.ent.gz pdb1ncr.ent.gz | 141.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ncr.json.gz 1ncr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/nc/1ncr https://data.pdbj.org/pub/pdb/validation_reports/nc/1ncr ftp://data.pdbj.org/pub/pdb/validation_reports/nc/1ncr ftp://data.pdbj.org/pub/pdb/validation_reports/nc/1ncr | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | x 60
| ||||||||
| 2 |
| ||||||||
| 3 | x 5
| ||||||||
| 4 | x 6
| ||||||||
| 5 | 
| ||||||||
| 単位格子 |
| ||||||||
| 対称性 | 点対称性: (ヘルマン・モーガン記号 : 532 / シェーンフリース記号 : 532 / シェーンフリース記号 : I (正20面体型対称 : I (正20面体型対称 )) )) |
- 要素
要素
-Coat protein ... , 4種, 4分子 ABCD
| #1: タンパク質 | 分子量: 32501.375 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Human rhinovirus 16 (ライノウイルス) Human rhinovirus 16 (ライノウイルス)属: Rhinovirus  ライノウイルス / 生物種: Human rhinovirus A ライノウイルス / 生物種: Human rhinovirus A ライノウイルス / 参照: UniProt: Q82122 ライノウイルス / 参照: UniProt: Q82122 |
|---|---|
| #2: タンパク質 | 分子量: 28990.615 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Human rhinovirus 16 (ライノウイルス) Human rhinovirus 16 (ライノウイルス)属: Rhinovirus  ライノウイルス / 生物種: Human rhinovirus A ライノウイルス / 生物種: Human rhinovirus A ライノウイルス / 参照: UniProt: Q82122 ライノウイルス / 参照: UniProt: Q82122 |
| #3: タンパク質 | 分子量: 26314.168 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Human rhinovirus 16 (ライノウイルス) Human rhinovirus 16 (ライノウイルス)属: Rhinovirus  ライノウイルス / 生物種: Human rhinovirus A ライノウイルス / 生物種: Human rhinovirus A ライノウイルス / 参照: UniProt: Q82122 ライノウイルス / 参照: UniProt: Q82122 |
| #4: タンパク質 | 分子量: 7374.025 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)   Human rhinovirus 16 (ライノウイルス) Human rhinovirus 16 (ライノウイルス)属: Rhinovirus  ライノウイルス / 生物種: Human rhinovirus A ライノウイルス / 生物種: Human rhinovirus A ライノウイルス / 参照: UniProt: Q82122 ライノウイルス / 参照: UniProt: Q82122 |
-非ポリマー , 4種, 341分子 
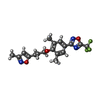





| #5: 化合物 | ChemComp-ZN / |
|---|---|
| #6: 化合物 | ChemComp-W11 /  Pleconaril Pleconaril |
| #7: 化合物 | ChemComp-MYR /  ミリスチン酸 ミリスチン酸 |
| #8: 水 | ChemComp-HOH /  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
結晶化 | *PLUS 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | 解像度: 2.7→20 Å / Num. obs: 452981 / Observed criterion σ(I): 0 |
| 反射 | *PLUS % possible obs: 40.9 % / Num. measured all: 1107533 / Rmerge(I) obs: 0.133 |
| 反射 シェル | *PLUS % possible obs: 34.9 % / Rmerge(I) obs: 0.395 |
- 解析
解析
| ソフトウェア | 名称: CNS / バージョン: 1 / 分類: 精密化 | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 2.7→20 Å / 交差検証法: THROUGHOUT / σ(F): 0 分子置換 / 解像度: 2.7→20 Å / 交差検証法: THROUGHOUT / σ(F): 0
| ||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.7→20 Å
| ||||||||||||||||||||
| 精密化 | *PLUS Rfactor Rfree : 0.248 / Rfactor Rwork : 0.248 / Rfactor Rwork : 0.249 : 0.249 | ||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー



























 PDBj
PDBj







