+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1gsf | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | GLUTATHIONE TRANSFERASE A1-1 COMPLEXED WITH ETHACRYNIC ACID | ||||||
 要素 要素 | GLUTATHIONE TRANSFERASE A1-1 | ||||||
 キーワード キーワード | TRANSFERASE (GLUTATHIONE) / A1-1 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 異性化酵素; 分子内で酸化還元酵素として働くもの; C=C結合の転位 / glutathione derivative biosynthetic process / 異性化酵素; 分子内で酸化還元酵素として働くもの; C=C結合の転位 / glutathione derivative biosynthetic process /  steroid delta-isomerase activity / linoleic acid metabolic process / steroid delta-isomerase activity / linoleic acid metabolic process /  薬物代謝 / 薬物代謝 /  glutathione peroxidase activity / Azathioprine ADME / Heme degradation / glutathione peroxidase activity / Azathioprine ADME / Heme degradation /  酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / NFE2L2 regulating anti-oxidant/detoxification enzymes ... 酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / NFE2L2 regulating anti-oxidant/detoxification enzymes ... 異性化酵素; 分子内で酸化還元酵素として働くもの; C=C結合の転位 / glutathione derivative biosynthetic process / 異性化酵素; 分子内で酸化還元酵素として働くもの; C=C結合の転位 / glutathione derivative biosynthetic process /  steroid delta-isomerase activity / linoleic acid metabolic process / steroid delta-isomerase activity / linoleic acid metabolic process /  薬物代謝 / 薬物代謝 /  glutathione peroxidase activity / Azathioprine ADME / Heme degradation / glutathione peroxidase activity / Azathioprine ADME / Heme degradation /  酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / NFE2L2 regulating anti-oxidant/detoxification enzymes / prostaglandin metabolic process / 酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / NFE2L2 regulating anti-oxidant/detoxification enzymes / prostaglandin metabolic process /  グルタチオン-S-トランスフェラーゼ / グルタチオン-S-トランスフェラーゼ /  glutathione transferase activity / glutathione metabolic process / epithelial cell differentiation / xenobiotic metabolic process / glutathione transferase activity / glutathione metabolic process / epithelial cell differentiation / xenobiotic metabolic process /  fatty acid binding / extracellular exosome / fatty acid binding / extracellular exosome /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / 解像度: 2.7 Å X線回折 / 解像度: 2.7 Å | ||||||
 データ登録者 データ登録者 | L'Hermite, G. / Sinning, I. / Cameron, A.D. / Jones, T.A. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 1995 ジャーナル: Structure / 年: 1995タイトル: Structural analysis of human alpha-class glutathione transferase A1-1 in the apo-form and in complexes with ethacrynic acid and its glutathione conjugate. 著者: Cameron, A.D. / Sinning, I. / L'Hermite, G. / Olin, B. / Board, P.G. / Mannervik, B. / Jones, T.A. #1:  ジャーナル: J.Mol.Biol. / 年: 1993 ジャーナル: J.Mol.Biol. / 年: 1993タイトル: Structure Determination and Refinement of Human Alpha Class Glutathione Transferase A1-1, and a Comparison with the Mu and Pi Class Enzyme 著者: Sinning, I. / Kleywegt, G.J. / Cowan, S.W. / Reinemer, P. / Dirr, H.W. / Huber, R. / Gilliland, G.L. / Armstrong, R.N. / Ji, X. / Board, P.G. / Olin, B. / Mannervik, B. / Jones, T.A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1gsf.cif.gz 1gsf.cif.gz | 172.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1gsf.ent.gz pdb1gsf.ent.gz | 142.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1gsf.json.gz 1gsf.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gs/1gsf https://data.pdbj.org/pub/pdb/validation_reports/gs/1gsf ftp://data.pdbj.org/pub/pdb/validation_reports/gs/1gsf ftp://data.pdbj.org/pub/pdb/validation_reports/gs/1gsf | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||
| 2 | 
| ||||||||||||||||
| 単位格子 |
| ||||||||||||||||
| Atom site foot note | 1: CIS PROLINE - PRO A 56 / 2: CIS PROLINE - PRO B 56 | ||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
| ||||||||||||||||
| 詳細 | MTRIX THE TRANSFORMATIONS PRESENTED ON MTRIX RECORDS BELOW DESCRIBE NON-CRYSTALLOGRAPHIC RELATIONSHIPS AMONG THE VARIOUS DOMAINS IN THIS ENTRY. APPLYING THE APPROPRIATE MTRIX TRANSFORMATION TO THE RESIDUES LISTED FIRST WILL YIELD APPROXIMATE COORDINATES FOR THE RESIDUES LISTED SECOND. APPLIED TO TRANSFORMED TO MTRIX RESIDUES RESIDUES RMSD M1 A 2 .. A 438 TRANSFORMS A CHAIN OF DIMER TO A CHAIN OF SECOND DIMER OF THE ASYMMETRIC UNIT M2 B 2 .. B 438 TRANSFORMS B CHAIN OF DIMER TO B CHAIN OF SECOND DIMER OF THE ASYMMETRIC UNIT M3 A 2 .. A 438 B 2 .. B 438 0.001 TRANSFORMS A CHAIN OF DIMER TO B CHAIN OF DIMER |
- 要素
要素
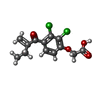
| #1: タンパク質 | 分子量: 25538.924 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 器官: LIVER Homo sapiens (ヒト) / 器官: LIVER 肝臓 / プラスミド: PTACGST2 / 発現宿主: 肝臓 / プラスミド: PTACGST2 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): JM103 Escherichia coli (大腸菌) / 株 (発現宿主): JM103参照: UniProt: P08263,  グルタチオン-S-トランスフェラーゼ グルタチオン-S-トランスフェラーゼ#2: 化合物 | ChemComp-EAA /  Etacrynic acid Etacrynic acid#3: 水 | ChemComp-HOH / |  水 水配列の詳細 | RESIDUES ARE NUMBERED FROM 2 TO 222. THE ETHACRYNIC | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.5 Å3/Da / 溶媒含有率: 50.9 % | ||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | *PLUS 温度: 20 ℃ / 手法: 蒸気拡散法 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射光源 | 波長: 1.54 |
|---|---|
| 検出器 | タイプ: RIGAKU RAXIS / 検出器: IMAGE PLATE / 日付: 1993年10月21日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.54 Å / 相対比: 1 : 1.54 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→10 Å / Num. obs: 26072 / % possible obs: 94.7 % / Observed criterion σ(I): 0 / 冗長度: 1.6 % / Rmerge(I) obs: 0.078 |
| 反射 | *PLUS 最高解像度: 2.7 Å / Num. measured all: 41606 / Rmerge(I) obs: 0.078 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.7→7.5 Å / σ(F): 0 詳細: RESIDUE ARG 89 HAS BEEN MODELLED IN 2 CONFORMATIONS AS FOR THE HIGHER RESOLUTION GST A 1-1 STRUCTURE COMPLEXED WITH A GLUTATHIONE, ETHACRYNIC ACID CONJUGATE. THIS RESIDUE LIES AT THE DIMER ...詳細: RESIDUE ARG 89 HAS BEEN MODELLED IN 2 CONFORMATIONS AS FOR THE HIGHER RESOLUTION GST A 1-1 STRUCTURE COMPLEXED WITH A GLUTATHIONE, ETHACRYNIC ACID CONJUGATE. THIS RESIDUE LIES AT THE DIMER INTERFACE CLOSE TO AN NCS COPY OF ITSELF.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 43 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.7→7.5 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / 分類: refinement X-PLOR / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj