+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1d8d | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CO-CRYSTAL STRUCTURE OF RAT PROTEIN FARNESYLTRANSFERASE COMPLEXED WITH A K-RAS4B PEPTIDE SUBSTRATE AND FPP ANALOG AT 2.0A RESOLUTION | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / TRANSFERASE (転移酵素) /  FTASE / PFT / PFTASE / FTASE / PFT / PFTASE /  FARNESYLTRANSFERASE / FARNESYLTRANSFERASE /  FARNESYL TRANSFERASE / CAAX / RAS / FARNESYL TRANSFERASE / CAAX / RAS /  CANCER (悪性腫瘍) CANCER (悪性腫瘍) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Apoptotic cleavage of cellular proteins / Inactivation, recovery and regulation of the phototransduction cascade / RAS processing / protein geranylgeranyltransferase activity / CAAX-protein geranylgeranyltransferase activity / CAAX-protein geranylgeranyltransferase complex / protein farnesylation /  protein geranylgeranyltransferase type I / regulation of fibroblast proliferation / positive regulation of tubulin deacetylation ...Apoptotic cleavage of cellular proteins / Inactivation, recovery and regulation of the phototransduction cascade / RAS processing / protein geranylgeranyltransferase activity / CAAX-protein geranylgeranyltransferase activity / CAAX-protein geranylgeranyltransferase complex / protein farnesylation / protein geranylgeranyltransferase type I / regulation of fibroblast proliferation / positive regulation of tubulin deacetylation ...Apoptotic cleavage of cellular proteins / Inactivation, recovery and regulation of the phototransduction cascade / RAS processing / protein geranylgeranyltransferase activity / CAAX-protein geranylgeranyltransferase activity / CAAX-protein geranylgeranyltransferase complex / protein farnesylation /  protein geranylgeranyltransferase type I / regulation of fibroblast proliferation / positive regulation of tubulin deacetylation / protein geranylgeranyltransferase type I / regulation of fibroblast proliferation / positive regulation of tubulin deacetylation /  protein farnesyltransferase / protein farnesyltransferase /  protein farnesyltransferase activity / protein farnesyltransferase activity /  protein farnesyltransferase complex / protein farnesyltransferase complex /  Rab geranylgeranyltransferase activity / protein geranylgeranylation / positive regulation of skeletal muscle acetylcholine-gated channel clustering / Rab geranylgeranyltransferase activity / protein geranylgeranylation / positive regulation of skeletal muscle acetylcholine-gated channel clustering /  farnesyltranstransferase activity / negative regulation of nitric-oxide synthase biosynthetic process / microtubule associated complex / forebrain astrocyte development / farnesyltranstransferase activity / negative regulation of nitric-oxide synthase biosynthetic process / microtubule associated complex / forebrain astrocyte development /  regulation of synaptic transmission, GABAergic / negative regulation of epithelial cell differentiation / epithelial tube branching involved in lung morphogenesis / type I pneumocyte differentiation / regulation of synaptic transmission, GABAergic / negative regulation of epithelial cell differentiation / epithelial tube branching involved in lung morphogenesis / type I pneumocyte differentiation /  enzyme-linked receptor protein signaling pathway / Rac protein signal transduction / skeletal muscle cell differentiation / positive regulation of Rac protein signal transduction / positive regulation of nitric-oxide synthase biosynthetic process / Signaling by RAS GAP mutants / Signaling by RAS GTPase mutants / Activation of RAS in B cells / RAS signaling downstream of NF1 loss-of-function variants / alpha-tubulin binding / RUNX3 regulates p14-ARF / SOS-mediated signalling / Activated NTRK3 signals through RAS / Activated NTRK2 signals through RAS / SHC1 events in ERBB4 signaling / response to inorganic substance / Signalling to RAS / glial cell proliferation / SHC-related events triggered by IGF1R / Activated NTRK2 signals through FRS2 and FRS3 / Estrogen-stimulated signaling through PRKCZ / SHC-mediated cascade:FGFR3 / MET activates RAS signaling / positive regulation of cell cycle / PTK6 Regulates RHO GTPases, RAS GTPase and MAP kinases / Signaling by PDGFRA transmembrane, juxtamembrane and kinase domain mutants / Signaling by PDGFRA extracellular domain mutants / SHC-mediated cascade:FGFR2 / SHC-mediated cascade:FGFR4 / protein-membrane adaptor activity / Signaling by FGFR4 in disease / SHC-mediated cascade:FGFR1 / Erythropoietin activates RAS / homeostasis of number of cells within a tissue / positive regulation of glial cell proliferation / FRS-mediated FGFR3 signaling / Signaling by CSF3 (G-CSF) / Signaling by FLT3 ITD and TKD mutants / FRS-mediated FGFR2 signaling / FRS-mediated FGFR4 signaling / Signaling by FGFR3 in disease / p38MAPK events / FRS-mediated FGFR1 signaling / Tie2 Signaling / Signaling by FGFR2 in disease / GRB2 events in EGFR signaling / striated muscle cell differentiation / SHC1 events in EGFR signaling / EGFR Transactivation by Gastrin / Signaling by FLT3 fusion proteins / Ras activation upon Ca2+ influx through NMDA receptor / FLT3 Signaling / Signaling by FGFR1 in disease / GRB2 events in ERBB2 signaling / NCAM signaling for neurite out-growth / CD209 (DC-SIGN) signaling / SHC1 events in ERBB2 signaling / Downstream signal transduction / Insulin receptor signalling cascade / Constitutive Signaling by Overexpressed ERBB2 / enzyme-linked receptor protein signaling pathway / Rac protein signal transduction / skeletal muscle cell differentiation / positive regulation of Rac protein signal transduction / positive regulation of nitric-oxide synthase biosynthetic process / Signaling by RAS GAP mutants / Signaling by RAS GTPase mutants / Activation of RAS in B cells / RAS signaling downstream of NF1 loss-of-function variants / alpha-tubulin binding / RUNX3 regulates p14-ARF / SOS-mediated signalling / Activated NTRK3 signals through RAS / Activated NTRK2 signals through RAS / SHC1 events in ERBB4 signaling / response to inorganic substance / Signalling to RAS / glial cell proliferation / SHC-related events triggered by IGF1R / Activated NTRK2 signals through FRS2 and FRS3 / Estrogen-stimulated signaling through PRKCZ / SHC-mediated cascade:FGFR3 / MET activates RAS signaling / positive regulation of cell cycle / PTK6 Regulates RHO GTPases, RAS GTPase and MAP kinases / Signaling by PDGFRA transmembrane, juxtamembrane and kinase domain mutants / Signaling by PDGFRA extracellular domain mutants / SHC-mediated cascade:FGFR2 / SHC-mediated cascade:FGFR4 / protein-membrane adaptor activity / Signaling by FGFR4 in disease / SHC-mediated cascade:FGFR1 / Erythropoietin activates RAS / homeostasis of number of cells within a tissue / positive regulation of glial cell proliferation / FRS-mediated FGFR3 signaling / Signaling by CSF3 (G-CSF) / Signaling by FLT3 ITD and TKD mutants / FRS-mediated FGFR2 signaling / FRS-mediated FGFR4 signaling / Signaling by FGFR3 in disease / p38MAPK events / FRS-mediated FGFR1 signaling / Tie2 Signaling / Signaling by FGFR2 in disease / GRB2 events in EGFR signaling / striated muscle cell differentiation / SHC1 events in EGFR signaling / EGFR Transactivation by Gastrin / Signaling by FLT3 fusion proteins / Ras activation upon Ca2+ influx through NMDA receptor / FLT3 Signaling / Signaling by FGFR1 in disease / GRB2 events in ERBB2 signaling / NCAM signaling for neurite out-growth / CD209 (DC-SIGN) signaling / SHC1 events in ERBB2 signaling / Downstream signal transduction / Insulin receptor signalling cascade / Constitutive Signaling by Overexpressed ERBB2 /  低分子量GTPアーゼ / G protein activity / Signaling by phosphorylated juxtamembrane, extracellular and kinase domain KIT mutants / VEGFR2 mediated cell proliferation / response to cytokine / FCERI mediated MAPK activation / regulation of long-term neuronal synaptic plasticity / Signaling by ERBB2 TMD/JMD mutants / RAF activation / Signaling by high-kinase activity BRAF mutants / Constitutive Signaling by EGFRvIII / 低分子量GTPアーゼ / G protein activity / Signaling by phosphorylated juxtamembrane, extracellular and kinase domain KIT mutants / VEGFR2 mediated cell proliferation / response to cytokine / FCERI mediated MAPK activation / regulation of long-term neuronal synaptic plasticity / Signaling by ERBB2 TMD/JMD mutants / RAF activation / Signaling by high-kinase activity BRAF mutants / Constitutive Signaling by EGFRvIII /  visual learning / visual learning /  wound healing / MAP2K and MAPK activation / Signaling by ERBB2 ECD mutants / lipid metabolic process wound healing / MAP2K and MAPK activation / Signaling by ERBB2 ECD mutants / lipid metabolic process類似検索 - 分子機能 | ||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | ||||||
| 手法 |  X線回折 / 解像度: 2 Å X線回折 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Long, S.B. / Casey, P.J. / Beese, L.S. | ||||||
 引用 引用 |  ジャーナル: Structure Fold.Des. / 年: 2000 ジャーナル: Structure Fold.Des. / 年: 2000タイトル: The basis for K-Ras4B binding specificity to protein farnesyltransferase revealed by 2 A resolution ternary complex structures. 著者: Long, S.B. / Casey, P.J. / Beese, L.S. #1:  ジャーナル: Science / 年: 1997 ジャーナル: Science / 年: 1997タイトル: Crystal Structure of Protein Farnesyltransferase at 2.25A Resolution 著者: Park, H.-W. / Boduluri, S.R. / Moomaw, J.F. / Casey, P.J. / Beese, L.S. #2:  ジャーナル: Biochemistry / 年: 1998 ジャーナル: Biochemistry / 年: 1998タイトル: Co-crystal Structure of Mammalian Protein Farnesyltransferase with a Farnesyl Diphosphate Substrate 著者: Long, S.B. / Casey, P.J. / Beese, L.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1d8d.cif.gz 1d8d.cif.gz | 173.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1d8d.ent.gz pdb1d8d.ent.gz | 134.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1d8d.json.gz 1d8d.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/d8/1d8d https://data.pdbj.org/pub/pdb/validation_reports/d8/1d8d ftp://data.pdbj.org/pub/pdb/validation_reports/d8/1d8d ftp://data.pdbj.org/pub/pdb/validation_reports/d8/1d8d | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-Farnesyltransferase ... , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 44098.145 Da / 分子数: 1 / 断片: alpha subunit / 由来タイプ: 組換発現 / 由来: (組換発現)   Rattus norvegicus (ドブネズミ) / 器官: BRAIN Rattus norvegicus (ドブネズミ) / 器官: BRAIN 脳 脳発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: Q04631,  squalene synthase squalene synthase |
|---|---|
| #2: タンパク質 | 分子量: 48722.281 Da / 分子数: 1 / 断片: beta subunit / 由来タイプ: 組換発現 / 由来: (組換発現)   Rattus norvegicus (ドブネズミ) / 器官: BRAIN Rattus norvegicus (ドブネズミ) / 器官: BRAIN 脳 脳発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: Q02293,  squalene synthase squalene synthase |
-タンパク質・ペプチド , 1種, 1分子 P
| #3: タンパク質・ペプチド | 分子量: 1298.723 Da / 分子数: 1 / 由来タイプ: 合成 詳細: This synthetic peptide is derived from human K-Ras4B, residues 178-188. 参照: UniProt: P01116 |
|---|
-非ポリマー , 4種, 453分子 





| #4: 化合物 | ChemComp-ZN / | ||||
|---|---|---|---|---|---|
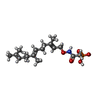
| #5: 化合物 | ChemComp-ACT /  酢酸塩 酢酸塩#6: 化合物 | ChemComp-FII / [( | #7: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.1 Å3/Da / 溶媒含有率: 60.35 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 温度: 290 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5.7 詳細: Peg 8000, ammonium acetate, DTT, pH 5.7, VAPOR DIFFUSION, HANGING DROP, temperature 290K | ||||||||||||||||||||
| 結晶化 | *PLUS 温度: 17 ℃ | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 96 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 |
| 検出器 | タイプ: RIGAKU RAXIS IV / 検出器: IMAGE PLATE / 日付: 1998年10月23日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.5418 Å / 相対比: 1 : 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2→35 Å / Num. all: 77998 / Num. obs: 76088 / % possible obs: 97.5 % / Observed criterion σ(I): 0 / 冗長度: 4.2 % / Biso Wilson estimate: 27.1 Å2 / Rmerge(I) obs: 0.045 / Net I/σ(I): 13.9 |
| 反射 シェル | 解像度: 2→2.02 Å / 冗長度: 2.3 % / Rmerge(I) obs: 0.197 / Num. unique all: 2154 / % possible all: 83.9 |
| 反射 | *PLUS Num. measured all: 320401 |
| 反射 シェル | *PLUS % possible obs: 83.9 % |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2→35 Å / Rfactor Rfree error: 0.003 / Data cutoff high absF: 10000000 / Data cutoff low absF: 0.001 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 2 / σ(I): -3 / 立体化学のターゲット値: Engh & Huber
| |||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 34.2 Å2
| |||||||||||||||||||||||||
| Refine analyze |
| |||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→35 Å
| |||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2→2.09 Å / Rfactor Rfree error: 0.013 / Total num. of bins used: 8
| |||||||||||||||||||||||||
| Xplor file |
| |||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.851 / 分類: refinement X-PLOR / バージョン: 3.851 / 分類: refinement | |||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj























