+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ceb | ||||||
|---|---|---|---|---|---|---|---|
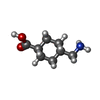
| タイトル | THE STRUCTURE OF THE NON-COVALENT COMPLEX OF RECOMBINANT KRINGLE 1 DOMAIN OF HUMAN PLASMINOGEN WITH AMCHA (TRANS-4-AMINOMETHYLCYCLOHEXANE-1-CARBOXYLIC ACID) | ||||||
 要素 要素 | PLASMINOGEN プラスミン プラスミン | ||||||
 キーワード キーワード |  SERINE PROTEASE (セリンプロテアーゼ) SERINE PROTEASE (セリンプロテアーゼ) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 プラスミン / trans-synaptic signaling by BDNF, modulating synaptic transmission / trophoblast giant cell differentiation / プラスミン / trans-synaptic signaling by BDNF, modulating synaptic transmission / trophoblast giant cell differentiation /  tissue remodeling / protein antigen binding / tissue remodeling / protein antigen binding /  再生 (生物学) / mononuclear cell migration / negative regulation of cell-cell adhesion mediated by cadherin / Signaling by PDGF / positive regulation of fibrinolysis ... 再生 (生物学) / mononuclear cell migration / negative regulation of cell-cell adhesion mediated by cadherin / Signaling by PDGF / positive regulation of fibrinolysis ... プラスミン / trans-synaptic signaling by BDNF, modulating synaptic transmission / trophoblast giant cell differentiation / プラスミン / trans-synaptic signaling by BDNF, modulating synaptic transmission / trophoblast giant cell differentiation /  tissue remodeling / protein antigen binding / tissue remodeling / protein antigen binding /  再生 (生物学) / mononuclear cell migration / negative regulation of cell-cell adhesion mediated by cadherin / Signaling by PDGF / positive regulation of fibrinolysis / Dissolution of Fibrin Clot / negative regulation of cell-substrate adhesion / myoblast differentiation / biological process involved in interaction with symbiont / labyrinthine layer blood vessel development / muscle cell cellular homeostasis / Activation of Matrix Metalloproteinases / 再生 (生物学) / mononuclear cell migration / negative regulation of cell-cell adhesion mediated by cadherin / Signaling by PDGF / positive regulation of fibrinolysis / Dissolution of Fibrin Clot / negative regulation of cell-substrate adhesion / myoblast differentiation / biological process involved in interaction with symbiont / labyrinthine layer blood vessel development / muscle cell cellular homeostasis / Activation of Matrix Metalloproteinases /  apolipoprotein binding / extracellular matrix disassembly / positive regulation of blood vessel endothelial cell migration / negative regulation of fibrinolysis / apolipoprotein binding / extracellular matrix disassembly / positive regulation of blood vessel endothelial cell migration / negative regulation of fibrinolysis /  fibrinolysis / Degradation of the extracellular matrix / serine-type peptidase activity / platelet alpha granule lumen / Schaffer collateral - CA1 synapse / fibrinolysis / Degradation of the extracellular matrix / serine-type peptidase activity / platelet alpha granule lumen / Schaffer collateral - CA1 synapse /  kinase binding / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / kinase binding / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) /  凝固・線溶系 / Platelet degranulation / protein-folding chaperone binding / collagen-containing extracellular matrix / blood microparticle / 凝固・線溶系 / Platelet degranulation / protein-folding chaperone binding / collagen-containing extracellular matrix / blood microparticle /  endopeptidase activity / endopeptidase activity /  protease binding / protein domain specific binding / negative regulation of cell population proliferation / external side of plasma membrane / protease binding / protein domain specific binding / negative regulation of cell population proliferation / external side of plasma membrane /  signaling receptor binding / serine-type endopeptidase activity / glutamatergic synapse / signaling receptor binding / serine-type endopeptidase activity / glutamatergic synapse /  enzyme binding / enzyme binding /  細胞膜 / 細胞膜 /  タンパク質分解 / タンパク質分解 /  extracellular space / extracellular exosome / extracellular region / extracellular space / extracellular exosome / extracellular region /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / 解像度: 2.07 Å X線回折 / 解像度: 2.07 Å | ||||||
 データ登録者 データ登録者 | Tulinsky, A. / Mathews, I.I. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1996 ジャーナル: Biochemistry / 年: 1996タイトル: Crystal structures of the recombinant kringle 1 domain of human plasminogen in complexes with the ligands epsilon-aminocaproic acid and trans-4-(aminomethyl)cyclohexane-1-carboxylic Acid. 著者: Mathews, I.I. / Vanderhoff-Hanaver, P. / Castellino, F.J. / Tulinsky, A. #1:  ジャーナル: Eur.J.Biochem. / 年: 1994 ジャーナル: Eur.J.Biochem. / 年: 1994タイトル: 1H-NMR Assignments and Secondary Structure of Human Plasminogen Kringle 1 著者: Rejante, M.R. / Llinas, M. #2:  ジャーナル: Blood Coagulation Fibrinolysis / 年: 1994 ジャーナル: Blood Coagulation Fibrinolysis / 年: 1994タイトル: The Structure of Recombinant Plasminogen Kringle 1 and the Fibrin Binding Site 著者: Wu, T.-P. / Padmanabhan, K.P. / Tulinsky, A. #3:  ジャーナル: Proteins / 年: 1988 ジャーナル: Proteins / 年: 1988タイトル: Lysine(Slash)Fibrin Binding Sites of Kringles Modeled After the Structure of Kringle 1 of Prothrombin 著者: Tulinsky, A. / Park, C.H. / Mao, B. / Llinas, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ceb.cif.gz 1ceb.cif.gz | 45.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ceb.ent.gz pdb1ceb.ent.gz | 35.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ceb.json.gz 1ceb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ce/1ceb https://data.pdbj.org/pub/pdb/validation_reports/ce/1ceb ftp://data.pdbj.org/pub/pdb/validation_reports/ce/1ceb ftp://data.pdbj.org/pub/pdb/validation_reports/ce/1ceb | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 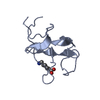
| ||||||||
| 2 | 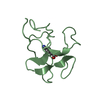
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: CIS PROLINE - PRO A 30 / 2: CIS PROLINE - PRO B 30 | ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (0.99956, -0.02947, 0.003414), ベクター  : : |
- 要素
要素
| #1: タンパク質 |  プラスミン / K1PG プラスミン / K1PG分子量: 10166.165 Da / 分子数: 2 / 断片: KRINGLE 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 器官: BLOOD Homo sapiens (ヒト) / 器官: BLOOD 血液 / 発現宿主: 血液 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P00747, Escherichia coli (大腸菌) / 参照: UniProt: P00747,  プラスミン プラスミン#2: 化合物 |  トラネキサム酸 トラネキサム酸#3: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.91 Å3/Da / 溶媒含有率: 35.65 % | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶 | *PLUS 溶媒含有率: 37 % | |||||||||||||||
結晶化 | *PLUS pH: 6.5 / 手法: 蒸気拡散法, ハンギングドロップ法 / 詳細: seeding | |||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 295 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 |
| 検出器 | タイプ: RIGAKU RAXIS IIC / 検出器: IMAGE PLATE |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.5418 Å / 相対比: 1 : 1.5418 Å / 相対比: 1 |
| 反射 | 最高解像度: 2.06 Å / Num. obs: 7028 / % possible obs: 70 % / Observed criterion σ(I): 1 / 冗長度: 2.6 % / Rmerge(I) obs: 0.0795 / Net I/σ(I): 1 |
| 反射 シェル | 解像度: 2.07→2.25 Å / Rmerge(I) obs: 0.14 / Mean I/σ(I) obs: 1 / % possible all: 56 |
| 反射 | *PLUS 最高解像度: 2.07 Å / 最低解像度: 10 Å / Num. measured all: 17924 |
| 反射 シェル | *PLUS 最高解像度: 2.07 Å / 最低解像度: 2.5 Å / % possible obs: 60 % / Rmerge(I) obs: 0.133 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.07→7 Å / σ(F): 3 詳細: NO ELECTRON DENSITY WAS OBSERVED FOR THE INTERKRINGLE RESIDUES 3A - 2A AND 80 - 86 IN BOTH MOLECULES.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.07→7 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj