+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1aeb | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | SPECIFICITY OF LIGAND BINDING TO A BURIED POLAR CAVITY AT THE ACTIVE SITE OF CYTOCHROME C PEROXIDASE (3-METHYLTHIAZOLE) | ||||||
 要素 要素 | CYTOCHROME C PEROXIDASE シトクロムcペルオキシダーゼ シトクロムcペルオキシダーゼ | ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / OXIDOREDUCTASE (酸化還元酵素) /  PEROXIDASE (ペルオキシダーゼ) / TRANSIT PEPTIDE PEROXIDASE (ペルオキシダーゼ) / TRANSIT PEPTIDE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 シトクロムcペルオキシダーゼ / シトクロムcペルオキシダーゼ /  cytochrome-c peroxidase activity / cytochrome-c peroxidase activity /  peroxidase activity / response to reactive oxygen species / hydrogen peroxide catabolic process / peroxidase activity / response to reactive oxygen species / hydrogen peroxide catabolic process /  ミトコンドリア / cellular response to oxidative stress / ミトコンドリア / cellular response to oxidative stress /  ミトコンドリアマトリックス / ミトコンドリアマトリックス /  heme binding / heme binding /  ミトコンドリア / ミトコンドリア /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å | ||||||
 データ登録者 データ登録者 | Musah, R.A. / Jensen, G.M. / Fitzgerald, M.M. / Mcree, D.E. / Goodin, D.B. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2002 ジャーナル: J.Mol.Biol. / 年: 2002タイトル: Artificial protein cavities as specific ligand-binding templates: characterization of an engineered heterocyclic cation-binding site that preserves the evolved specificity of the parent protein. 著者: Musah, R.A. / Jensen, G.M. / Bunte, S.W. / Rosenfeld, R.J. / Goodin, D.B. #1:  ジャーナル: Biochemistry / 年: 1994 ジャーナル: Biochemistry / 年: 1994タイトル: Small Molecule Binding to an Artificially Created Cavity at the Active Site of Cytochrome C Peroxidase 著者: Fitzgerald, M.M. / Churchill, M.J. / Mcree, D.E. / Goodin, D.B. #2:  ジャーナル: Biochemistry / 年: 1993 ジャーナル: Biochemistry / 年: 1993タイトル: The Asp-His-Fe Triad of Cytochrome C Peroxidase Controls the Reduction Potential, Electronic Structure, and Coupling of the Tryptophan Free Radical to the Heme 著者: Goodin, D.B. / Mcree, D.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1aeb.cif.gz 1aeb.cif.gz | 81.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1aeb.ent.gz pdb1aeb.ent.gz | 64.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1aeb.json.gz 1aeb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ae/1aeb https://data.pdbj.org/pub/pdb/validation_reports/ae/1aeb ftp://data.pdbj.org/pub/pdb/validation_reports/ae/1aeb ftp://data.pdbj.org/pub/pdb/validation_reports/ae/1aeb | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1aedC  1aeeC  1aefC  1aegC  1aehC  1aejC  1aekC  1aemC  1aenC  1aeoC  1aeqC  1aa4S S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 |  シトクロムcペルオキシダーゼ / CCP/W191G シトクロムcペルオキシダーゼ / CCP/W191G分子量: 33459.242 Da / 分子数: 1 / 変異: W191G / 由来タイプ: 組換発現 / 詳細: CRYSTAL FORM BY 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)細胞株: BL21 / 細胞内の位置: MITOCHONDRIA  ミトコンドリア / 遺伝子: CCP / Organelle: MITOCHONDRIA ミトコンドリア / 遺伝子: CCP / Organelle: MITOCHONDRIA ミトコンドリア / プラスミド: PT7CCP / 生物種 (発現宿主): Escherichia coli / 細胞内の位置 (発現宿主): CYTOPLASM / 遺伝子 (発現宿主): CCP (MKT) / 発現宿主: ミトコンドリア / プラスミド: PT7CCP / 生物種 (発現宿主): Escherichia coli / 細胞内の位置 (発現宿主): CYTOPLASM / 遺伝子 (発現宿主): CCP (MKT) / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21 (DE3) Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21 (DE3)参照: UniProt: P00431,  シトクロムcペルオキシダーゼ シトクロムcペルオキシダーゼ |
|---|---|
| #2: 化合物 | ChemComp-HEM /  Heme B Heme B |
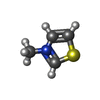
| #3: 化合物 | ChemComp-3MT / |
| #4: 水 | ChemComp-HOH /  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.2 Å3/Da / 溶媒含有率: 61 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | pH: 6 詳細: 20% MPD, 40 MM PHOSPHATE PH 6.0. A SINGLE CRYSTAL OF W191G WAS SOAKED IN 50MM 3-METHYLTHIAZOLIUM IODIDE IN 40% MPD AND 60 MM PHOSPHATE AT PH 4.5. | ||||||||||||||||||||
| 結晶化 | *PLUS 温度: 18 ℃ / 手法: 蒸気拡散法, シッティングドロップ法 | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 290 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 |
| 検出器 | タイプ: SIEMENS / 検出器: AREA DETECTOR / 日付: 1995年11月1日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.5418 Å / 相対比: 1 : 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.1→15 Å / Num. obs: 21612 / % possible obs: 87 % / Observed criterion σ(I): 0 / 冗長度: 1.3 % / Rmerge(I) obs: 0.069 / Rsym value: 0.055 / Net I/σ(I): 14.92 |
| 反射 シェル | 解像度: 2.04→2.16 Å / 冗長度: 1.1 % / Rmerge(I) obs: 0.24 / Mean I/σ(I) obs: 2.04 / Rsym value: 0.55 / % possible all: 75 |
| 反射 | *PLUS 最低解像度: 15 Å / Num. obs: 22213 / % possible obs: 89 % / Rmerge(I) obs: 0.067 |
- 解析
解析
| ソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1AA4 解像度: 2.1→7 Å / Data cutoff high absF: 10000000 / Data cutoff low absF: 0.5 / σ(F): 0 /
| ||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.1→7 Å
|
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj