+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9906 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of membrane protein | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | PTS / ManYZ /  transporter (運搬体タンパク質) / transporter (運搬体タンパク質) /  Mannose (マンノース) / Mannose (マンノース) /  PROTEIN TRANSPORT PROTEIN TRANSPORT | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mannose transmembrane transport / protein-N(PI)-phosphohistidine-mannose phosphotransferase system transporter activity / glucose import across plasma membrane / phosphoenolpyruvate-dependent sugar phosphotransferase system / transmembrane transporter complex /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /   Escherichia coli str. K-12 substr. MG1655 (大腸菌) / Escherichia coli str. K-12 substr. MG1655 (大腸菌) /   Escherichia coli (strain K12) (大腸菌) Escherichia coli (strain K12) (大腸菌) | ||||||||||||
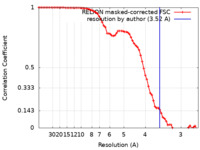
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.52 Å クライオ電子顕微鏡法 / 解像度: 3.52 Å | ||||||||||||
 データ登録者 データ登録者 | Wang JW / Zeng JW | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2019 ジャーナル: Cell Res / 年: 2019タイトル: Structure of the mannose transporter of the bacterial phosphotransferase system. 著者: Xueli Liu / Jianwei Zeng / Kai Huang / Jiawei Wang /  | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9906.map.gz emd_9906.map.gz | 19.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9906-v30.xml emd-9906-v30.xml emd-9906.xml emd-9906.xml | 11.3 KB 11.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9906_fsc.xml emd_9906_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9906.png emd_9906.png | 63.7 KB | ||
| Filedesc metadata |  emd-9906.cif.gz emd-9906.cif.gz | 5.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9906 http://ftp.pdbj.org/pub/emdb/structures/EMD-9906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9906 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9906.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9906.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.338 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : ManYZ trimer complex
| 全体 | 名称: ManYZ trimer complex |
|---|---|
| 要素 |
|
-超分子 #1: ManYZ trimer complex
| 超分子 | 名称: ManYZ trimer complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K12 Escherichia coli (大腸菌) / 株: K12 |
| 分子量 | 理論値: 176 kDa/nm |
-分子 #1: PTS mannose transporter subunit IID
| 分子 | 名称: PTS mannose transporter subunit IID / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli str. K-12 substr. MG1655 (大腸菌) Escherichia coli str. K-12 substr. MG1655 (大腸菌) |
| 分子量 | 理論値: 30.983373 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MVDTTQTTTE KKLTQSDIRG VFLRSNLFQG SWNFERMQAL GFCFSMVPAI RRLYPENNEA RKQAIRRHLE FFNTQPFVAA PILGVTLAL EEQRANGAEI DDGAINGIKV GLMGPLAGVG DPIFWGTVRP VFAALGAGIA MSGSLLGPLL FFILFNLVRL A TRYYGVAY ...文字列: MVDTTQTTTE KKLTQSDIRG VFLRSNLFQG SWNFERMQAL GFCFSMVPAI RRLYPENNEA RKQAIRRHLE FFNTQPFVAA PILGVTLAL EEQRANGAEI DDGAINGIKV GLMGPLAGVG DPIFWGTVRP VFAALGAGIA MSGSLLGPLL FFILFNLVRL A TRYYGVAY GYSKGIDIVK DMGGGFLQKL TEGASILGLF VMGALVNKWT HVNIPLVVSR ITDQTGKEHV TTVQTILDQL MP GLVPLLL TFACMWLLRK KVNPLWIIVG FFVIGIAGYA CGLLGL UniProtKB:  UNIPROTKB: A0A387CW08 UNIPROTKB: A0A387CW08 |
-分子 #2: PTS system mannose-specific EIIC component
| 分子 | 名称: PTS system mannose-specific EIIC component / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (strain K12) (大腸菌) / 株: K12 Escherichia coli (strain K12) (大腸菌) / 株: K12 |
| 分子量 | 理論値: 28.752783 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MEITTLQIVL VFIVACIAGM GSILDEFQFH RPLIACTLVG IVLGDMKTGI IIGGTLEMIA LGWMNIGAAV APDAALASII STILVIAGH QSIGAGIALA IPLAAAGQVL TIIVRTITVA FQHAADKAAD NGNLTAISWI HVSSLFLQAM RVAIPAVIVA L SVGTSEVQ ...文字列: MEITTLQIVL VFIVACIAGM GSILDEFQFH RPLIACTLVG IVLGDMKTGI IIGGTLEMIA LGWMNIGAAV APDAALASII STILVIAGH QSIGAGIALA IPLAAAGQVL TIIVRTITVA FQHAADKAAD NGNLTAISWI HVSSLFLQAM RVAIPAVIVA L SVGTSEVQ NMLNAIPEVV TNGLNIAGGM IVVVGYAMVI NMMRAGYLMP FFYLGFVTAA FTNFNLVALG VIGTVMAVLY IQ LSPKYNR VAGAPAQAAG NNDLDNELDH HHHHHHH UniProtKB: PTS system mannose-specific EIIC component |
-分子 #3: alpha-D-mannopyranose
| 分子 | 名称: alpha-D-mannopyranose / タイプ: ligand / ID: 3 / コピー数: 3 / 式: MAN |
|---|---|
| 分子量 | 理論値: 180.156 Da |
| Chemical component information |  ChemComp-MAN: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー