+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9796 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
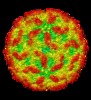
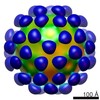
| タイトル | AAV5 in complex with AAVR | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  adeno-associated virus (アデノ随伴ウイルス) / AAV5 / adeno-associated virus (アデノ随伴ウイルス) / AAV5 /  receptor (受容体) / AAVR / receptor (受容体) / AAVR /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=1 icosahedral viral capsid /  neuron migration / cytoplasmic vesicle / neuron migration / cytoplasmic vesicle /  ゴルジ体 / structural molecule activity / ゴルジ体 / structural molecule activity /  核小体 / 核小体 /  ゴルジ体 / ゴルジ体 /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Adeno-associated virus - 5 (アデノ随伴ウイルス) / Adeno-associated virus - 5 (アデノ随伴ウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.18 Å クライオ電子顕微鏡法 / 解像度: 3.18 Å | |||||||||
 データ登録者 データ登録者 | Lou Z / Zhang R | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Divergent engagements between adeno-associated viruses with their cellular receptor AAVR. 著者: Ran Zhang / Guangxue Xu / Lin Cao / Zixian Sun / Yong He / Mengtian Cui / Yuna Sun / Shentao Li / Huapeng Li / Lan Qin / Mingxu Hu / Zhengjia Yuan / Zipei Rao / Wei Ding / Zihe Rao / Zhiyong Lou /  要旨: Adeno-associated virus (AAV) receptor (AAVR) is an essential receptor for the entry of multiple AAV serotypes with divergent rules; however, the mechanism remains unclear. Here, we determine the ...Adeno-associated virus (AAV) receptor (AAVR) is an essential receptor for the entry of multiple AAV serotypes with divergent rules; however, the mechanism remains unclear. Here, we determine the structures of the AAV1-AAVR and AAV5-AAVR complexes, revealing the molecular details by which PKD1 recognizes AAV5 and PKD2 is solely engaged with AAV1. PKD2 lies on the plateau region of the AAV1 capsid. However, the AAV5-AAVR interface is strikingly different, in which PKD1 is bound at the opposite side of the spike of the AAV5 capsid than the PKD2-interacting region of AAV1. Residues in strands F/G and the CD loop of PKD1 interact directly with AAV5, whereas residues in strands B/C/E and the BC loop of PKD2 make contact with AAV1. These findings further the understanding of the distinct mechanisms by which AAVR recognizes various AAV serotypes and provide an example of a single receptor engaging multiple viral serotypes with divergent rules. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9796.map.gz emd_9796.map.gz | 64.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9796-v30.xml emd-9796-v30.xml emd-9796.xml emd-9796.xml | 11.3 KB 11.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9796.png emd_9796.png | 241.2 KB | ||
| Filedesc metadata |  emd-9796.cif.gz emd-9796.cif.gz | 5.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9796 http://ftp.pdbj.org/pub/emdb/structures/EMD-9796 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9796 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9796 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9796.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9796.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Adeno-associated virus - 5
| 全体 | 名称:   Adeno-associated virus - 5 (アデノ随伴ウイルス) Adeno-associated virus - 5 (アデノ随伴ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Adeno-associated virus - 5
| 超分子 | 名称: Adeno-associated virus - 5 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: capsid protein VP1
| 超分子 | 名称: capsid protein VP1 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Adeno-associated virus - 5 (アデノ随伴ウイルス) Adeno-associated virus - 5 (アデノ随伴ウイルス) |
-超分子 #3: PKD1
| 超分子 | 名称: PKD1 / タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Adeno-associated virus - 5 (アデノ随伴ウイルス) Adeno-associated virus - 5 (アデノ随伴ウイルス) |
| 分子量 | 理論値: 58.213078 KDa |
| 配列 | 文字列: DGVGNASGDW HCDSTWMGDR VVTKSTRTWV LPSYNNHQYR EIKSGSVDGS NANAYFGYST PWGYFDFNRF HSHWSPRDWQ RLINNYWGF RPRSLRVKIF NIQVKEVTVQ DSTTTIANNL TSTVQVFTDD DYQLPYVVGN GTEGCLPAFP PQVFTLPQYG Y ATLNRDNT ...文字列: DGVGNASGDW HCDSTWMGDR VVTKSTRTWV LPSYNNHQYR EIKSGSVDGS NANAYFGYST PWGYFDFNRF HSHWSPRDWQ RLINNYWGF RPRSLRVKIF NIQVKEVTVQ DSTTTIANNL TSTVQVFTDD DYQLPYVVGN GTEGCLPAFP PQVFTLPQYG Y ATLNRDNT ENPTERSSFF CLEYFPSKML RTGNNFEFTY NFEEVPFHSS FAPSQNLFKL ANPLVDQYLY RFVSTNNTGG VQ FNKNLAG RYANTYKNWF PGPMGRTQGW NLGSGVNRAS VSAFATTNRM ELEGASYQVP PQPNGMTNNL QGSNTYALEN TMI FNSQPA NPGTTATYLE GNMLITSESE TQPVNRVAYN VGGQMATNNQ SSTTAPATGT YNLQEIVPGS VWMERDVYLQ GPIW AKIPE TGAHFHPSPA MGGFGLKHPP PMMLIKNTPV PGNITSFSDV PVSSFITQYS TGQVTVEMEW ELKKENSKRW NPEIQ YTNN YNDPQFVDFA PDSTGEYRTT RPIGTRYLTR PL UniProtKB:  カプシド カプシド |
-分子 #2: Dyslexia-associated protein KIAA0319-like protein
| 分子 | 名称: Dyslexia-associated protein KIAA0319-like protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.911322 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: VIKELVVSAG ESVQITLPKN EVQLNAYVLQ EPPKGETYTY DWQLITHPRD YSGEMEGKHS QILKLSKLTP GLYEFKVIVE GQNAHGEGY VNVTVKPE UniProtKB: Dyslexia-associated protein KIAA0319-like protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 36.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.18 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 12590 |
 ムービー
ムービー コントローラー
コントローラー