+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9642 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of CVA10 mature virus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | picornavirus uncoating /  receptor binding (受容体) / receptor binding (受容体) /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane /  : ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / : ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane /  : / : /  カプシド / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / カプシド / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity /  RNA helicase activity / induction by virus of host autophagy / RNA helicase activity / induction by virus of host autophagy /  RNA依存性RNAポリメラーゼ / symbiont entry into host cell / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity / RNA依存性RNAポリメラーゼ / symbiont entry into host cell / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /  RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / structural molecule activity / virion attachment to host cell / RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / structural molecule activity / virion attachment to host cell /  ATP hydrolysis activity / ATP hydrolysis activity /  タンパク質分解 / タンパク質分解 /  RNA binding / RNA binding /  ATP binding / ATP binding /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |    Coxsackievirus A10 (コクサッキーウイルス) Coxsackievirus A10 (コクサッキーウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.0 Å クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Zhu L / Sun Y | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: Structures of Coxsackievirus A10 unveil the molecular mechanisms of receptor binding and viral uncoating. 著者: Ling Zhu / Yao Sun / Jinyan Fan / Bin Zhu / Lei Cao / Qiang Gao / Yanjun Zhang / Hongrong Liu / Zihe Rao / Xiangxi Wang /  要旨: Coxsackievirus A10 (CVA10), a human type-A Enterovirus (HEV-A), can cause diseases ranging from hand-foot-and-mouth disease to polio-myelitis-like disease. CVA10, together with some other HEV-As, ...Coxsackievirus A10 (CVA10), a human type-A Enterovirus (HEV-A), can cause diseases ranging from hand-foot-and-mouth disease to polio-myelitis-like disease. CVA10, together with some other HEV-As, utilizing the molecule KREMEN1 as an entry receptor, constitutes a KREMEN1-dependent subgroup within HEV-As. Currently, there is no vaccine or antiviral therapy available for treating diseases caused by CVA10. The atomic-resolution structure of the CVA10 virion, which is within the KREMEN1-dependent subgroup, shows significant conformational differences in the putative receptor binding sites and serotype-specific epitopes, when compared to the SCARB2-dependent subgroup of HEV-A, such as EV71, highlighting specific differences between the sub-groups. We also report two expanded structures of CVA10, an empty particle and uncoating intermediate at atomic resolution, as well as a medium-resolution genome structure reconstructed using a symmetry-mismatch method. Structural comparisons coupled with previous results, reveal an ordered signal transmission process for enterovirus uncoating, converting exo-genetic receptor-attachment inputs into a generic RNA release mechanism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9642.map.gz emd_9642.map.gz | 167 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9642-v30.xml emd-9642-v30.xml emd-9642.xml emd-9642.xml | 12.5 KB 12.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9642.png emd_9642.png | 253.7 KB | ||
| Filedesc metadata |  emd-9642.cif.gz emd-9642.cif.gz | 5.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9642 http://ftp.pdbj.org/pub/emdb/structures/EMD-9642 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9642 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9642 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9642.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9642.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
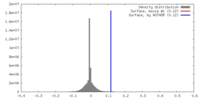
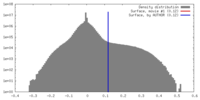
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Coxsackievirus A10
| 全体 | 名称:    Coxsackievirus A10 (コクサッキーウイルス) Coxsackievirus A10 (コクサッキーウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Coxsackievirus A10
| 超分子 | 名称: Coxsackievirus A10 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #2-#4 / NCBI-ID: 42769 / 生物種: Coxsackievirus A10 / ウイルスタイプ: VIRION / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Coxsackievirus A10 (コクサッキーウイルス) Coxsackievirus A10 (コクサッキーウイルス) |
| 分子量 | 理論値: 33.101188 KDa |
| 組換発現 | 生物種:   Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 配列 | 文字列: GDPVEDIIHD ALGSTARRAI SSATNVESAA NTTPSSHRLE TGRVPALQAA ETGATSNATD ENMIETRCVV NRNGVLETTI NHFFSRSGL VGVVNLTDGG TDTTGYVTWD IDIMGFVQLR RKCEMFTYMR FNAEFTFVTT TKNGEARPYM LQYMYVPPGA P KPTGRDAF ...文字列: GDPVEDIIHD ALGSTARRAI SSATNVESAA NTTPSSHRLE TGRVPALQAA ETGATSNATD ENMIETRCVV NRNGVLETTI NHFFSRSGL VGVVNLTDGG TDTTGYVTWD IDIMGFVQLR RKCEMFTYMR FNAEFTFVTT TKNGEARPYM LQYMYVPPGA P KPTGRDAF QWQTATNPSV FVKLTDPPAQ VSVPFMSPAS AYQWFYDGYP TFGQHPETSN TTYGLCPNNM MGTFAVRVVS RE ASQLKLQ TRVYMKLKHV RAWVPRPIRS QPYLLKNFPN YDSSKITNSA RDRSSIKQAN UniProtKB: Genome polyprotein |
-分子 #2: VP2
| 分子 | 名称: VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Coxsackievirus A10 (コクサッキーウイルス) Coxsackievirus A10 (コクサッキーウイルス) |
| 分子量 | 理論値: 27.783105 KDa |
| 組換発現 | 生物種:   Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 配列 | 文字列: SPSVEACGYS DRVAQLTVGN SSITTQEAAN IVLAYGEWPE YCPDTDATAV DKPTRPDVSV NRFYTLDSKM WQENSTGWYW KFPDVLNKT GVFGQNAQFH YLYRSGFCLH VQCNASKFHQ GALLVAVIPE FVIAGRGSNT KPNEAPHPGF TTTFPGTTGA T FHDPYVLD ...文字列: SPSVEACGYS DRVAQLTVGN SSITTQEAAN IVLAYGEWPE YCPDTDATAV DKPTRPDVSV NRFYTLDSKM WQENSTGWYW KFPDVLNKT GVFGQNAQFH YLYRSGFCLH VQCNASKFHQ GALLVAVIPE FVIAGRGSNT KPNEAPHPGF TTTFPGTTGA T FHDPYVLD SGVPLSQALI YPHQWINLRT NNCATVIVPY INAVPFDSAI NHSNFGLIVI PVSPLKYSSG ATTAIPITIT IA PLNSEFG GLRQAVSQ UniProtKB: Genome polyprotein |
-分子 #3: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Coxsackievirus A10 (コクサッキーウイルス) Coxsackievirus A10 (コクサッキーウイルス) |
| 分子量 | 理論値: 26.279826 KDa |
| 組換発現 | 生物種:   Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 配列 | 文字列: GIPAELRPGT NQFLTTDDDT AAPILPGFTP TPTIHIPGEV HSLLELCRVE TILEVNNTTE ATGLTRLLIP VSSQNKADEL CAAFMVDPG RIGPWQSTLV GQICRYYTQW SGSLKVTFMF TGSFMATGKM LVAYSPPGSA QPANRETAML GTHVIWDFGL Q SSVSLVIP ...文字列: GIPAELRPGT NQFLTTDDDT AAPILPGFTP TPTIHIPGEV HSLLELCRVE TILEVNNTTE ATGLTRLLIP VSSQNKADEL CAAFMVDPG RIGPWQSTLV GQICRYYTQW SGSLKVTFMF TGSFMATGKM LVAYSPPGSA QPANRETAML GTHVIWDFGL Q SSVSLVIP WISNTHFRTA KTGGNYDYYT AGVVTLWYQT NYVVPPETPG EAYIIAMGAD LYKFTLKICK DTDEVTQQAV LQ UniProtKB: Genome polyprotein |
-分子 #4: VP4
| 分子 | 名称: VP4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Coxsackievirus A10 (コクサッキーウイルス) Coxsackievirus A10 (コクサッキーウイルス) |
| 分子量 | 理論値: 7.464104 KDa |
| 組換発現 | 生物種:   Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 配列 | 文字列: MGAQVSTQKS GSHETGNVAT GGSTINFTNI NYYKDSYAAS ATRQDFTQDP KKFTQPVLDS IRELSAPLN UniProtKB: Genome polyprotein |
-分子 #5: SPHINGOSINE
| 分子 | 名称: SPHINGOSINE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: SPH |
|---|---|
| 分子量 | 理論値: 299.492 Da |
| Chemical component information |  ChemComp-SPH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 25.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP |
|---|---|
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 4586 |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)