+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9634 | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of Enterovirus D68 mature virion in complex with Fab 15C5 and 11G1 | ||||||||||||||||||||||||||||||||||||
 マップデータ マップデータ | None | ||||||||||||||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane /  : ...: / : ...: /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane /  : / : /  カプシド / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / カプシド / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity /  peptidase activity / host cell cytoplasm / peptidase activity / host cell cytoplasm /  RNA helicase activity / induction by virus of host autophagy / RNA helicase activity / induction by virus of host autophagy /  RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity / RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /  RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / structural molecule activity / virion attachment to host cell / RNA-dependent RNA polymerase activity / DNA-templated transcription / host cell nucleus / structural molecule activity / virion attachment to host cell /  ATP hydrolysis activity / ATP hydrolysis activity /  タンパク質分解 / タンパク質分解 /  RNA binding / RNA binding /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||||||||||||||||||||||||||
| 生物種 |   Mus musculus (ハツカネズミ) / Mus musculus (ハツカネズミ) /   Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) | ||||||||||||||||||||||||||||||||||||
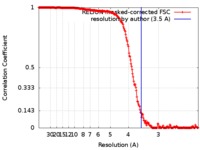
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Zheng QB / Zhu R / Xu LF / He MZ / Yan XD / Cheng T / Li SW | ||||||||||||||||||||||||||||||||||||
| 資金援助 |  中国, 中国,  米国, 11件 米国, 11件
| ||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2019 ジャーナル: Nat Microbiol / 年: 2019タイトル: Atomic structures of enterovirus D68 in complex with two monoclonal antibodies define distinct mechanisms of viral neutralization. 著者: Qingbing Zheng / Rui Zhu / Longfa Xu / Maozhou He / Xiaodong Yan / Dongxiao Liu / Zhichao Yin / Yangtao Wu / Yongchao Li / Lisheng Yang / Wangheng Hou / Shuxuan Li / Zizhen Li / Zhenqin Chen ...著者: Qingbing Zheng / Rui Zhu / Longfa Xu / Maozhou He / Xiaodong Yan / Dongxiao Liu / Zhichao Yin / Yangtao Wu / Yongchao Li / Lisheng Yang / Wangheng Hou / Shuxuan Li / Zizhen Li / Zhenqin Chen / Zhihai Li / Hai Yu / Ying Gu / Jun Zhang / Timothy S Baker / Z Hong Zhou / Barney S Graham / Tong Cheng / Shaowei Li / Ningshao Xia /   要旨: Enterovirus D68 (EV-D68) undergoes structural transformation between mature, cell-entry intermediate (A-particle) and empty forms throughout its life cycle. Structural information for the various ...Enterovirus D68 (EV-D68) undergoes structural transformation between mature, cell-entry intermediate (A-particle) and empty forms throughout its life cycle. Structural information for the various forms and antibody-bound capsids will facilitate the development of effective vaccines and therapeutics against EV-D68 infection, which causes childhood respiratory and paralytic diseases worldwide. Here, we report the structures of three EV-D68 capsid states representing the virus at major phases. We further describe two original monoclonal antibodies (15C5 and 11G1) with distinct structurally defined mechanisms for virus neutralization. 15C5 and 11G1 engage the capsid loci at icosahedral three-fold and five-fold axes, respectively. To block viral attachment, 15C5 binds three forms of capsids, and triggers mature virions to transform into A-particles, mimicking engagement by the functional receptor ICAM-5, whereas 11G1 exclusively recognizes the A-particle. Our data provide a structural and molecular explanation for the transition of picornavirus capsid conformations and demonstrate distinct mechanisms for antibody-mediated neutralization. | ||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9634.map.gz emd_9634.map.gz | 766 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9634-v30.xml emd-9634-v30.xml emd-9634.xml emd-9634.xml | 18.4 KB 18.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9634_fsc.xml emd_9634_fsc.xml | 20.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9634.png emd_9634.png | 199.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9634 http://ftp.pdbj.org/pub/emdb/structures/EMD-9634 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9634 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9634 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6aj9MC  9629C  9631C  9632C  9633C  9635C  9636C  6aj0C  6aj2C  6aj3C  6aj7C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9634.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9634.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.128 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Enterovirus D68
| 全体 | 名称:   Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Enterovirus D68
| 超分子 | 名称: Enterovirus D68 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 42789 / 生物種: Enterovirus D68 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: VL of Fab 11G1
| 分子 | 名称: VL of Fab 11G1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 12.625044 KDa |
| 配列 | 文字列: DIVMSQSPSS LAVSVGEKVT MSCKSSQSLL YSNNQKNYLA WYQQKPGQSP QLLIYWASTR ESGVPDRFTG SESGTDFTLT ISSVKAEDL AVFYCQQYYN YPWTFGGGTK LEIK |
-分子 #2: VH of Fab 11G1
| 分子 | 名称: VH of Fab 11G1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 13.517063 KDa |
| 配列 | 文字列: QVQLQQSGAE LVRPGVSVKI SCKGSGYIFT DYVMHWVKQS HSKSLEWIGV ISTYYGDGSY NQKFKGKATM TVDKSSSTAY MELARLTSE DSAIYYCVRG ENYYGVSGTM DYWGQGTSVT VSS |
-分子 #3: VL of Fab 15C5
| 分子 | 名称: VL of Fab 15C5 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 11.656906 KDa |
| 配列 | 文字列: DIQMIQSPSS MFASLGDRVS LSCRASQDIR GNLDWYQQKP GGTIKLLIYS TSNLNSGVPS RFSGSGSGSD YSLTISSLES EDFADYYCL QRNAYPLTFG GGTKLEIK |
-分子 #4: VH of Fab 15C5
| 分子 | 名称: VH of Fab 15C5 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 13.027412 KDa |
| 配列 | 文字列: QVQLQQSGTE LAKPGASVKM SCKASGYTFT NYWMHWFKQR PGQGLEWIGY VNPNTDYTDY NHKFKDKATL TADKSSSTAY MQLSSLTSE DSAVYFCARG TFGNYASFAY WGQGTLV |
-分子 #5: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) |
| 分子量 | 理論値: 32.673035 KDa |
| 配列 | 文字列: IESIIKTATD TVKSEINAEL GVVPSLNAVE TGATSNTEPE EAIQTRTVIN QHGVSETLVE NFLSRAALVS KRSFEYKDHT SSAAQADKN FFKWTINTRS FVQLRRKLEL FTYLRFDAEI TILTTVAVNG SGNNTYVGLP DLTLQAMFVP TGALTPEKQD S FHWQSGSN ...文字列: IESIIKTATD TVKSEINAEL GVVPSLNAVE TGATSNTEPE EAIQTRTVIN QHGVSETLVE NFLSRAALVS KRSFEYKDHT SSAAQADKN FFKWTINTRS FVQLRRKLEL FTYLRFDAEI TILTTVAVNG SGNNTYVGLP DLTLQAMFVP TGALTPEKQD S FHWQSGSN ASVFFKISDP PARITIPFMC INSAYSVFYD GFAGFEKNGL YGINPADTIG NLCVRIVNEH QPVGFTVTVR VY MKPKHIK AWAPRPPRTL PYMSIANANY KGKERAPNAL NAIIGNRDSV KTMPHNIV |
-分子 #6: Capsid protein VP2
| 分子 | 名称: Capsid protein VP2 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) |
| 分子量 | 理論値: 27.567135 KDa |
| 配列 | 文字列: SPSAEACGYS DRVLQLKLGN SAIVTQEAAN YCCAYGEWPN YLPDHEAVAI DKPTQPETAT DRFYTLKSVK WETGSTGWWW KLPDALNNI GMFGQNVQHH YLYRSGFLIH VQCNATKFHQ GALLVVAIPE HQRGAHNTNT SPGFDDIMKG EEGGTFNHPY V LDDGTSLA ...文字列: SPSAEACGYS DRVLQLKLGN SAIVTQEAAN YCCAYGEWPN YLPDHEAVAI DKPTQPETAT DRFYTLKSVK WETGSTGWWW KLPDALNNI GMFGQNVQHH YLYRSGFLIH VQCNATKFHQ GALLVVAIPE HQRGAHNTNT SPGFDDIMKG EEGGTFNHPY V LDDGTSLA CATIFPHQWI NLRTNNSATI VLPWMNAAPM DFPLRHNQWT LAIIPVVPLG TRTTSSMVPI TVSIAPMCCE FN GLRHAIT Q |
-分子 #7: Capsid protein VP3
| 分子 | 名称: Capsid protein VP3 / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) |
| 分子量 | 理論値: 27.142842 KDa |
| 配列 | 文字列: GVPTYLLPGS GQFLTTDDHS SAPALPCFNP TPEMHIPGQV RNMLEVVQVE SMMEINNTES AVGMERLKVD ISALTDVDQL LFNIPLDIQ LDGPLRNTLV GNISRYYTHW SGSLEMTFMF CGSFMATGKL ILCYTPPGGS CPTTRETAML GTHIVWDFGL Q SSVTLIIP ...文字列: GVPTYLLPGS GQFLTTDDHS SAPALPCFNP TPEMHIPGQV RNMLEVVQVE SMMEINNTES AVGMERLKVD ISALTDVDQL LFNIPLDIQ LDGPLRNTLV GNISRYYTHW SGSLEMTFMF CGSFMATGKL ILCYTPPGGS CPTTRETAML GTHIVWDFGL Q SSVTLIIP WISGSHYRMF NNDAKSTNAN VGYVTCFMQT NLIVPSESSD TCSLIGFIAA KDDFSLRLMR DSPDIGQLDH LH AAEAAYQ |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 25.0 e/Å2 |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー