+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9366 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Atomic structures and deletion mutant reveal different capsid-binding patterns and functional significance of tegument protein pp150 in murine and human cytomegaloviruses with implications for therapeutic development | ||||||||||||||||||
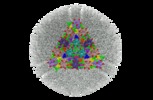
 マップデータ マップデータ | 3D reconstruction for MCMV capsid associated with pM32 tegument protein. Contour level=4.0 was estimated while showing the map using step=2 in Chimera. | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=16 icosahedral viral capsid /  viral tegument / viral tegument /  viral capsid assembly / viral process / viral capsid assembly / viral process /  カプシド / host cell nucleus / structural molecule activity / カプシド / host cell nucleus / structural molecule activity /  DNA binding DNA binding類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  MuHV-1 (ウイルス) / MuHV-1 (ウイルス) /  Murid herpesvirus 1 (strain Smith) (ウイルス) Murid herpesvirus 1 (strain Smith) (ウイルス) | ||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.0 Å クライオ電子顕微鏡法 / 解像度: 5.0 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Liu W / Dai XH / Jih J / Chan K / Trang P / Yu XK / Balogun R / Mei Y / Liu FY / Zhou ZH | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2019 ジャーナル: PLoS Pathog / 年: 2019タイトル: Atomic structures and deletion mutant reveal different capsid-binding patterns and functional significance of tegument protein pp150 in murine and human cytomegaloviruses with ...タイトル: Atomic structures and deletion mutant reveal different capsid-binding patterns and functional significance of tegument protein pp150 in murine and human cytomegaloviruses with implications for therapeutic development. 著者: Wei Liu / Xinghong Dai / Jonathan Jih / Karen Chan / Phong Trang / Xuekui Yu / Rilwan Balogun / Ye Mei / Fenyong Liu / Z Hong Zhou /   要旨: Cytomegalovirus (CMV) infection causes birth defects and life-threatening complications in immunosuppressed patients. Lack of vaccine and need for more effective drugs have driven widespread ongoing ...Cytomegalovirus (CMV) infection causes birth defects and life-threatening complications in immunosuppressed patients. Lack of vaccine and need for more effective drugs have driven widespread ongoing therapeutic development efforts against human CMV (HCMV), mostly using murine CMV (MCMV) as the model system for preclinical animal tests. The recent publication (Yu et al., 2017, DOI: 10.1126/science.aam6892) of an atomic model for HCMV capsid with associated tegument protein pp150 has infused impetus for rational design of novel vaccines and drugs, but the absence of high-resolution structural data on MCMV remains a significant knowledge gap in such development efforts. Here, by cryoEM with sub-particle reconstruction method, we have obtained the first atomic structure of MCMV capsid with associated pp150. Surprisingly, the capsid-binding patterns of pp150 differ between HCMV and MCMV despite their highly similar capsid structures. In MCMV, pp150 is absent on triplex Tc and exists as a "Λ"-shaped dimer on other triplexes, leading to only 260 groups of two pp150 subunits per capsid in contrast to 320 groups of three pp150 subunits each in a "Δ"-shaped fortifying configuration. Many more amino acids contribute to pp150-pp150 interactions in MCMV than in HCMV, making MCMV pp150 dimer inflexible thus incompatible to instigate triplex Tc-binding as observed in HCMV. While pp150 is essential in HCMV, our pp150-deletion mutant of MCMV remained viable though with attenuated infectivity and exhibiting defects in retaining viral genome. These results thus invalidate targeting pp150, but lend support to targeting capsid proteins, when using MCMV as a model for HCMV pathogenesis and therapeutic studies. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9366.map.gz emd_9366.map.gz | 1.4 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9366-v30.xml emd-9366-v30.xml emd-9366.xml emd-9366.xml | 26.7 KB 26.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9366.png emd_9366.png | 309.2 KB | ||
| その他 |  emd_9366_additional_1.map.gz emd_9366_additional_1.map.gz emd_9366_additional_2.map.gz emd_9366_additional_2.map.gz emd_9366_additional_3.map.gz emd_9366_additional_3.map.gz | 220.8 MB 219.3 MB 219.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9366 http://ftp.pdbj.org/pub/emdb/structures/EMD-9366 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9366 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9366 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9366.map.gz / 形式: CCP4 / 大きさ: 7.8 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9366.map.gz / 形式: CCP4 / 大きさ: 7.8 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction for MCMV capsid associated with pM32 tegument protein. Contour level=4.0 was estimated while showing the map using step=2 in Chimera. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.351 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Sub-particle reconstruction for MCMV 2-fold region
| ファイル | emd_9366_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sub-particle reconstruction for MCMV 2-fold region | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Sub-particle reconstruction for MCMV 3-fold region
| ファイル | emd_9366_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sub-particle reconstruction for MCMV 3-fold region | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Sub-particle reconstruction for MCMV 5-fold region
| ファイル | emd_9366_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sub-particle reconstruction for MCMV 5-fold region | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Murid herpesvirus 1 (strain Smith)
| 全体 | 名称:  Murid herpesvirus 1 (strain Smith) (ウイルス) Murid herpesvirus 1 (strain Smith) (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Murid herpesvirus 1 (strain Smith)
| 超分子 | 名称: Murid herpesvirus 1 (strain Smith) / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10367 / 生物種: Murid herpesvirus 1 (strain Smith) / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|
-分子 #1: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  MuHV-1 (ウイルス) / 株: Smith MuHV-1 (ウイルス) / 株: Smith |
| 分子量 | 理論値: 151.673031 KDa |
| 配列 | 文字列: MGENWTATEL LPKLDVPIDL LTHIKLSVGE EMFNNFRLYY GDDPERYNLS FEAIFGTYCN KIEWVTFLGT ALATAAHAIM FHDLNKMTT GKMLFYIQVP RVATGAGIPT SRQTTVMVSK YSEKSPITIP FEISAACLTH LKETFEDTLL DKLLNADAVN T VLRAVKNT ...文字列: MGENWTATEL LPKLDVPIDL LTHIKLSVGE EMFNNFRLYY GDDPERYNLS FEAIFGTYCN KIEWVTFLGT ALATAAHAIM FHDLNKMTT GKMLFYIQVP RVATGAGIPT SRQTTVMVSK YSEKSPITIP FEISAACLTH LKETFEDTLL DKLLNADAVN T VLRAVKNT ADAMERGLID TFLRVLLRHA PPCFVLRTLM EHGTIARRMA TRVQRANIAQ GFKSKMLATI FLLDRSRDRG QL TRYLDML TDCVTESILD NPETYTVGGG ERLAGVIVST HTVVQALLNA LGGSIRRTGV KTPASYGKFV LSKENAVTAI AHH AIMADF SQHADRIQQS SQKDLPESQF LDQRLTFTET QMDVLKVGER LVALEHLRKV YKNTDVQDPL ERDVELTFYF PVGL HVPSG RAYSTAENKI KLVDTAENQL PTTVYFYNKD RIPQRISHAE ALKTLCHPAI HDAGPCLEAF AQAGPPQGDD RVRAL CRRE FVREHMAHAT RRLVHFYQAR IDPPRTANEA KHDFSTKEFA KVDNYLLFTE LHPFFDFCFH TENGQVRPLC TPRIMV GNL PEALAPADFH DLRAKQALEL TKVRAPEGHE ATLQVLRASL TDHQYPELFY LIESLIHGDP AAFETGIELV TRCVNNY WR QRGLLAFANS YDMVRLIATR LGDGAVVPAA YTHYRNLLSI TRFVARTCEL TGLNGRLCDE PLLAYVSALH DPRLWPPF V QALPRNANLV RVVADDVPLD AAHIEERNPG TSDVARMIAM DQAEPLFVDA RRTSDEEMVA QKVYYLCLVP AVLNNHACG AGLNLKHLLV KLFYTKFFLT ADPDSLTAGE EALTNNPLLA ALVRDVATDE NVTANQAAEE LFHLVAHVPE NAQMLEIRAA LDPAQRHGA PSAGFESLQH VLYNGFCMTT VPKLLQEYLT VIPFHRFYSD PGLAATANHD IRVFLNDFPQ YQRCDGGFPL S PIFAHEYH HWHRTPFSCY SAACAHTLES VLTLAIMHHK MSPVSIAALS RMGLHPGFAL TVVRTDTFET DTLLYSTKAS TA VIINTPI VTKEDRDINT VFHVSQNINT VEMGLGYGAT TCTAHLRRVR SDMGSRMQDL FQVFPMHVYR NEDVDAWVRQ ATG ARRTEV LDSEAISILT FGRKTDKGGP ALLHGQRATC EVILTPVSAD LEFFRYPNNP RGRSSSMLGV DPYDEDAALA TLYD HTSPD PQTFVSTNNP WGSQRGSLGD VIYNTRNREK LGHNPSFYSP CAQFFTTDDI INANKTLFKT VEEYLNRSQD CIHGE TDLQ YICVEGTNSI VEKPCRFLQE ALTQHTGTTQ ALMESQLKGT SKLGLDETHY GNYSIGETIP LQQSILFNS |
-分子 #2: Tegument protein
| 分子 | 名称: Tegument protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  MuHV-1 (ウイルス) / 株: Smith MuHV-1 (ウイルス) / 株: Smith |
| 分子量 | 理論値: 78.749961 KDa |
| 配列 | 文字列: MSARGRAAGD DGRQAELMAT LGFVRLSKSS VGKVKKFLNN LYDLKSINLC RHPRVIAECR GTDLSRETQL YNEMVLWLRY HEKLTARRP GHLPLLTRIR QDYDKLFGFV AARPELCGFD GLTEVNVFDD AVYGDDYVPR VDVFLRGLED LARCLCAQGP D KPARAVIM ...文字列: MSARGRAAGD DGRQAELMAT LGFVRLSKSS VGKVKKFLNN LYDLKSINLC RHPRVIAECR GTDLSRETQL YNEMVLWLRY HEKLTARRP GHLPLLTRIR QDYDKLFGFV AARPELCGFD GLTEVNVFDD AVYGDDYVPR VDVFLRGLED LARCLCAQGP D KPARAVIM GFINMRAEEV NRLMDNVRDA AERVLVYEVL DVRDPLNEDP SVLVHNRLVY LCRLAYAISK SWQTLSHMCL DR INSLRRR LILAFHDRPA FARVYARNAL ERPVDGTTAY NLLRRLEEDF LLFRNALRWG DPDWGLESEL ESEGDNSDAG SDL DLEDEE DDDDGGGPGG HDDESGGNRT PDPGMSLHDD TGIANTCLIG GDDEDCGGGS GRCLEFDPDS ERCLGVKMVN GRAL RWWNP TGIMVDNEAA VWIDEHGRVM DKPPPKELRK ASSDDGGNKK NPPPKKNVTP PVSGSNSVGG GVQTPSTASG KRTGK KKEG GGGYLLRSRS TDDDEVRKMK KDGTIDDRAD RELKMALQKA RESTADSDLS TILPRTEPLR KVAFVGDPVA AFGDTV RTT SSSKGFDDGP FTTQGASVLL PPPLGGPGST LTLPPDLPDL SGVLADEDVQ DSRYGKIPKS RTKKHPTFPE NYKQRPP PH NKDDEYYWDE TGDNMPVGED GGGVLEDLRK GLEGIDLKTG GGGSLQPPLS QQFAGSPFAG SDGDGGGLVK KSSSSH |
-分子 #3: Small capsomere-interacting protein
| 分子 | 名称: Small capsomere-interacting protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  MuHV-1 (ウイルス) / 株: Smith MuHV-1 (ウイルス) / 株: Smith |
| 分子量 | 理論値: 9.845198 KDa |
| 配列 | 文字列: MSTNVSSAAS GGGSSGGSSG ASSGGGGGGS GGSSKKEEER RKQFGANVLN LAPAMVAQPV ISTMIPKYMK MGGHEDKLAY QLDLLRMLS IAKKATVIQ |
-分子 #4: Minor capsid protein
| 分子 | 名称: Minor capsid protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  MuHV-1 (ウイルス) / 株: Smith MuHV-1 (ウイルス) / 株: Smith |
| 分子量 | 理論値: 33.281289 KDa |
| 配列 | 文字列: MANVSSFGPM KREVMEFDPE DPYKVSKMRK LERSIAKGYV YGADHQAITA RFFVRESLGE VEQKNLGVLM FRLDTGIEMP STVLVSLFF LSMVAENVSA ATKNTLAAIY GREGEAIRTW LRDGAWRLHR VVHPLGCTNS ITPGATCLIT CSMRGHSYNM L KTEIYPLL ...文字列: MANVSSFGPM KREVMEFDPE DPYKVSKMRK LERSIAKGYV YGADHQAITA RFFVRESLGE VEQKNLGVLM FRLDTGIEMP STVLVSLFF LSMVAENVSA ATKNTLAAIY GREGEAIRTW LRDGAWRLHR VVHPLGCTNS ITPGATCLIT CSMRGHSYNM L KTEIYPLL VPKEIYLDLD GESTDEIRFV YFVITYDYNS DRQGRPSAFV VVSRITHRHT LINVLRYRFR VSRFHFLNNS IS GYGPSTG CLGTLQRLGW FCSRDSRSGI VASRAGQLSV VKLEKFYVDV GPLVEFA |
-分子 #5: Triplex capsid protein 2
| 分子 | 名称: Triplex capsid protein 2 / タイプ: protein_or_peptide / ID: 5 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  MuHV-1 (ウイルス) / 株: Smith MuHV-1 (ウイルス) / 株: Smith |
| 分子量 | 理論値: 34.660262 KDa |
| 配列 | 文字列: METTVLVTFE QRLTTGDVGK LSRLIGAVIP IPYRHHLLGS SQVGLDAVVK DKTRDYSRMR ARMREMTLTI MRRVEGNQMI LGVPTHGQC YTIRNTGPVS WEKGDVLTTL PPVFSGEVTG LVSVSDWDLV LPWIVPMALA TEINQRMMML ALLSLDRSHE E VRAATAQL ...文字列: METTVLVTFE QRLTTGDVGK LSRLIGAVIP IPYRHHLLGS SQVGLDAVVK DKTRDYSRMR ARMREMTLTI MRRVEGNQMI LGVPTHGQC YTIRNTGPVS WEKGDVLTTL PPVFSGEVTG LVSVSDWDLV LPWIVPMALA TEINQRMMML ALLSLDRSHE E VRAATAQL RVVRYRDATL TLPEITIDDT VLIDMRNVCI SLSMIANLSS EVTLAYVRKL ALEDSNMLLM KCQEILGRRM PQ VGVGAGS SGDRNDPPAR SRTNYNITPT EELNKLTALF VMIRQITDVI SEQPAFLVCD VSPDDKSALC IYKG |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: PBS buffer, PH=7.4 |
|---|---|
| グリッド | 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: The grids were manually plunged into the Ethane.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 47000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: AGFA SCIENTA FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 実像数: 2200 / 平均電子線量: 25.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 58254 |
|---|---|
| CTF補正 | ソフトウェア - 名称: CTFFIND |
| 初期モデル | モデルのタイプ: OTHER / 詳細: a sphere density map |
| 初期 角度割当 | タイプ: COMMON LINE |
| 最終 角度割当 | タイプ: COMMON LINE |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 5.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 47982 ) / 解像度のタイプ: BY AUTHOR / 解像度: 5.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 47982 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 200 |
|---|---|
| 得られたモデル |  PDB-6nhj: |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X