+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9313 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the HO BMC shell: subregion classified for BMC-T: TD-TDTDTD | |||||||||
 マップデータ マップデータ | Four BMC-T positions classified: TD-TDTDTD | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  microcompartment / shell / compartmentalization / BMC fold / microcompartment / shell / compartmentalization / BMC fold /  STRUCTURAL PROTEIN (タンパク質) STRUCTURAL PROTEIN (タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Greber BJ / Sutter M / Kerfeld CA | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2019 ジャーナル: Structure / 年: 2019タイトル: The Plasticity of Molecular Interactions Governs Bacterial Microcompartment Shell Assembly. 著者: Basil J Greber / Markus Sutter / Cheryl A Kerfeld /  要旨: Bacterial microcompartments (BMCs) are composed of an enzymatic core encapsulated by a selectively permeable protein shell that enhances catalytic efficiency. Many pathogenic bacteria derive ...Bacterial microcompartments (BMCs) are composed of an enzymatic core encapsulated by a selectively permeable protein shell that enhances catalytic efficiency. Many pathogenic bacteria derive competitive advantages from their BMC-based catabolism, implicating BMCs as drug targets. BMC shells are of interest for bioengineering due to their diverse and selective permeability properties and because they self-assemble. A complete understanding of shell composition and organization is a prerequisite for biotechnological applications. Here, we report the cryoelectron microscopy structure of a BMC shell at 3.0-Å resolution, using an image-processing strategy that allowed us to determine the previously uncharacterized structural details of the interactions formed by the BMC-T and BMC-T shell subunits in the context of the assembled shell. We found unexpected structural plasticity among these interactions, resulting in distinct shell populations assembled from varying numbers of the BMC-T and BMC-T subunits. We discuss the implications of these findings on shell assembly and function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9313.map.gz emd_9313.map.gz | 17 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9313-v30.xml emd-9313-v30.xml emd-9313.xml emd-9313.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9313.png emd_9313.png | 163.6 KB | ||
| Filedesc metadata |  emd-9313.cif.gz emd-9313.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9313 http://ftp.pdbj.org/pub/emdb/structures/EMD-9313 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9313 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9313 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6n09MC  9296C  9307C  9308C  9309C  9310C  9311C  9312C  9314C  9315C  6mzuC  6mzvC  6mzxC  6mzyC  6n06C  6n07C  6n0fC  6n0gC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9313.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9313.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Four BMC-T positions classified: TD-TDTDTD | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
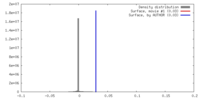
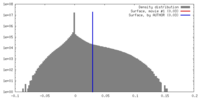
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bacterial microcompartment shell from Haliangium ochraceum
| 全体 | 名称: Bacterial microcompartment shell from Haliangium ochraceum |
|---|---|
| 要素 |
|
-超分子 #1: Bacterial microcompartment shell from Haliangium ochraceum
| 超分子 | 名称: Bacterial microcompartment shell from Haliangium ochraceum タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) |
| 分子量 | 理論値: 6.5 MDa |
-分子 #1: Microcompartments protein
| 分子 | 名称: Microcompartments protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア)株: DSM 14365 / JCM 11303 / SMP-2 |
| 分子量 | 理論値: 22.904137 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSITLRTYIF LDALQPQLAT FIGKTARGFL PVPGQASLWV EIAPGIAINR VTDAALKATK VQPAVQVVER AYGLLEVHHF DQGEVLAAG STILDKLEVR EEGRLKPQVM THQIIRAVEA YQTQIINRNS QGMMILPGES LFILETQPAG YAVLAANEAE K AANVHLVN ...文字列: MSITLRTYIF LDALQPQLAT FIGKTARGFL PVPGQASLWV EIAPGIAINR VTDAALKATK VQPAVQVVER AYGLLEVHHF DQGEVLAAG STILDKLEVR EEGRLKPQVM THQIIRAVEA YQTQIINRNS QGMMILPGES LFILETQPAG YAVLAANEAE K AANVHLVN VTPYGAFGRL YLAGSEAEID AAAEAAEAAI RSVSGVAQES FRDR UniProtKB:  Bacterial microcompartment protein trimer-2 Bacterial microcompartment protein trimer-2 |
-分子 #2: Microcompartments protein
| 分子 | 名称: Microcompartments protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 36 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア)株: DSM 14365 / JCM 11303 / SMP-2 |
| 分子量 | 理論値: 10.126718 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MADALGMIEV RGFVGMVEAA DAMVKAAKVE LIGYEKTGGG YVTAVVRGDV AAVKAATEAG QRAAERVGEV VAVHVIPRPH VNVDAALPL GRTPGMDKSA UniProtKB:  Bacterial microcompartment protein homohexamer Bacterial microcompartment protein homohexamer |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: HOLEY / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: CARBON / 支持フィルム - #1 - トポロジー: CONTINUOUS / 詳細: unspecified | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 5-7 second incubation of the sample on the grid before blotting and plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 3.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 48543 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-30 / 撮影したグリッド数: 1 / 実像数: 928 / 平均露光時間: 4.5 sec. / 平均電子線量: 25.0 e/Å2 詳細: 928 images retained after inspection for image quality. |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 31800 詳細: 1000 particles were picked manually to generate reference templates for subsequent auto-picking in RELION 1.4. |
|---|---|
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: 詳細: The reference was re-scaled and placed in a 512x512x512 pixel box to match the pixel size and appropriate box size for the data. |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 3次元分類 | クラス数: 2 / ソフトウェア - 名称: RELION (ver. 1.4) 詳細: Three sequential classifications for four BMC-T positions in total, using the symmetry-expanded particle dataset |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.4) 詳細: The selected particle subset was refined without masking and subsequently masked to reveal only the subregion of the BMC shell to which the focused classification had been applied. 使用した粒子像数: 106640 |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)