+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9138 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV-1 vaccine elicited Fab BDA1 in complex with the HIV Env trimer BG505 SOSIPv5.2 | |||||||||
 マップデータ マップデータ | HIV-1 vaccine elicited Fab BDA1 in complex with the HIV Env trimer BG505 SOSIPv5.2 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 21.0 Å ネガティブ染色法 / 解像度: 21.0 Å | |||||||||
 データ登録者 データ登録者 | Cottrell CA / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2019 ジャーナル: Cell / 年: 2019タイトル: Slow Delivery Immunization Enhances HIV Neutralizing Antibody and Germinal Center Responses via Modulation of Immunodominance. 著者: Kimberly M Cirelli / Diane G Carnathan / Bartek Nogal / Jacob T Martin / Oscar L Rodriguez / Amit A Upadhyay / Chiamaka A Enemuo / Etse H Gebru / Yury Choe / Federico Viviano / Catherine ...著者: Kimberly M Cirelli / Diane G Carnathan / Bartek Nogal / Jacob T Martin / Oscar L Rodriguez / Amit A Upadhyay / Chiamaka A Enemuo / Etse H Gebru / Yury Choe / Federico Viviano / Catherine Nakao / Matthias G Pauthner / Samantha Reiss / Christopher A Cottrell / Melissa L Smith / Raiza Bastidas / William Gibson / Amber N Wolabaugh / Mariane B Melo / Benjamin Cossette / Venkatesh Kumar / Nirav B Patel / Talar Tokatlian / Sergey Menis / Daniel W Kulp / Dennis R Burton / Ben Murrell / William R Schief / Steven E Bosinger / Andrew B Ward / Corey T Watson / Guido Silvestri / Darrell J Irvine / Shane Crotty /   要旨: Conventional immunization strategies will likely be insufficient for the development of a broadly neutralizing antibody (bnAb) vaccine for HIV or other difficult pathogens because of the ...Conventional immunization strategies will likely be insufficient for the development of a broadly neutralizing antibody (bnAb) vaccine for HIV or other difficult pathogens because of the immunological hurdles posed, including B cell immunodominance and germinal center (GC) quantity and quality. We found that two independent methods of slow delivery immunization of rhesus monkeys (RMs) resulted in more robust T follicular helper (T) cell responses and GC B cells with improved Env-binding, tracked by longitudinal fine needle aspirates. Improved GCs correlated with the development of >20-fold higher titers of autologous nAbs. Using a new RM genomic immunoglobulin locus reference, we identified differential IgV gene use between immunization modalities. Ab mapping demonstrated targeting of immunodominant non-neutralizing epitopes by conventional bolus-immunized animals, whereas slow delivery-immunized animals targeted a more diverse set of epitopes. Thus, alternative immunization strategies can enhance nAb development by altering GCs and modulating the immunodominance of non-neutralizing epitopes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9138.map.gz emd_9138.map.gz | 20.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9138-v30.xml emd-9138-v30.xml emd-9138.xml emd-9138.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9138.png emd_9138.png | 25.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9138 http://ftp.pdbj.org/pub/emdb/structures/EMD-9138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9138 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  0569C  0570C  0571C  0572C  0573C  0574C  0575C  0576C  0577C  0578C  0579C  0580C  0581C  0582C  9175C  9176C  9177C  9178C  9179C  9180C  9181C  9182C  9183C 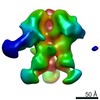 9184C 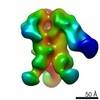 9185C 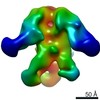 9186C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9138.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9138.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HIV-1 vaccine elicited Fab BDA1 in complex with the HIV Env trimer BG505 SOSIPv5.2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
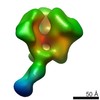
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
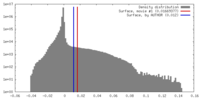
| ボクセルのサイズ | X=Y=Z: 1.98 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 vaccine elicited Fab BDA1 in complex with the HIV Env trime...
| 全体 | 名称: HIV-1 vaccine elicited Fab BDA1 in complex with the HIV Env trimer BG505 SOSIPv5.2 |
|---|---|
| 要素 |
|
-超分子 #1: HIV-1 vaccine elicited Fab BDA1 in complex with the HIV Env trime...
| 超分子 | 名称: HIV-1 vaccine elicited Fab BDA1 in complex with the HIV Env trimer BG505 SOSIPv5.2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: SOSIP trimer
| 超分子 | 名称: SOSIP trimer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-超分子 #3: BDA1 Fab
| 超分子 | 名称: BDA1 Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: BG505 SOSIPv5.2 gp120
| 分子 | 名称: BG505 SOSIPv5.2 gp120 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETKKHNVWA THCCVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPCV KLTPLCVTLQ CTNVTNNITD DMRGELKNCS FNMTTELRDK KQKVYSLFYR LDVVQINENQ GNRSNNSNKE YRLINCNTSA ...文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETKKHNVWA THCCVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPCV KLTPLCVTLQ CTNVTNNITD DMRGELKNCS FNMTTELRDK KQKVYSLFYR LDVVQINENQ GNRSNNSNKE YRLINCNTSA ITQACPKVSF EPIPIHYCAP AGFAILKCKD KKFNGTGPCP SVSTVQCTHG IKPVVSTQLL LNGSLAEEEV MIRSENITNN AKNILVQFNT PVQINCTRPN NNTRKSIRIG PGQWFYATGD IIGDIRQAHC NVSKATWNET LGKVVKQLRK HFGNNTIIRF ANSSGGDLEV TTHSFNCGGE FFYCNTSGLF NSTWISNTSV QGSNSTGSND SITLPCRIKQ IINMWQRIGQ AMYAPPIQGV IRCVSNITGL ILTRDGGSTN STTETFRPGG GDMRDNWRSE LYKYKVVKIE PLGVAPTRCK RRVVGRRRRR R |
-分子 #2: BG505 SOSIPv5.2 gp41
| 分子 | 名称: BG505 SOSIPv5.2 gp41 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAPEC QQHLLKLTVW GIKQLQARVL AVERYLRDQQ LLGIWGCSGK LICCTNVPWN SSWSNRNLSE IWDNMTWLQW DKEISNYTQI IYGLLEESQN QQEKNEQDLL ALD |
-分子 #3: BDA1 Fab heavy chain
| 分子 | 名称: BDA1 Fab heavy chain / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSETLSL TCAVSGASIS IYWWGWIRQP PGKGLEWIGE IIGSSGSTNS NPSFKSRVTI SKDASKNQFS LNLNSVTAAD TAVYYCVRVG AAISLPFDYW GQGVLVTVSS |
-分子 #4: BDA1 Fab light chain
| 分子 | 名称: BDA1 Fab light chain / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SYELTQPPSV SVSPGQTARI TCSGDALPKK YAYWFQQKPG QSPVLIIYED NKRPSGIPER FSGSSSGTVA TLTISGAQVE DEGDYYCYSR HSSGNHGLFG GGTRLTVL |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate |
| グリッド | 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 撮影 | フィルム・検出器のモデル: FEI CETA (4k x 4k) / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 撮影したグリッド数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 25.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 122593 |
|---|---|
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 3次元分類 | クラス数: 2 / 平均メンバー数/クラス: 15000 / ソフトウェア - 名称: RELION (ver. 3.0b0) |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 21.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0b0) / 使用した粒子像数: 15024 |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)