+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9038 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | B41 SOSIP.664 in complex with soluble CD4 (D1-D2), the co-receptor mimicking antibody 21c and the broadly neutralizing antibody 8ANC195 | |||||||||
 マップデータ マップデータ | Sharpened map of complex used for model building and refinement. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報helper T cell enhancement of adaptive immune response /  interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell / interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell /  T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation ...helper T cell enhancement of adaptive immune response / T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation ...helper T cell enhancement of adaptive immune response /  interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell / interleukin-16 binding / interleukin-16 receptor activity / maintenance of protein location in cell /  T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation / Alpha-defensins / positive regulation of kinase activity / T cell selection / MHC class II protein binding / cellular response to granulocyte macrophage colony-stimulating factor stimulus / interleukin-15-mediated signaling pathway / positive regulation of monocyte differentiation / Nef Mediated CD4 Down-regulation / Alpha-defensins / positive regulation of kinase activity /  regulation of T cell activation / regulation of T cell activation /  T cell receptor complex / extracellular matrix structural constituent / Other interleukin signaling / T cell receptor complex / extracellular matrix structural constituent / Other interleukin signaling /  enzyme-linked receptor protein signaling pathway / Translocation of ZAP-70 to Immunological synapse / Phosphorylation of CD3 and TCR zeta chains / regulation of calcium ion transport / macrophage differentiation / Generation of second messenger molecules / T cell differentiation / PD-1 signaling / positive regulation of protein kinase activity / Binding and entry of HIV virion / enzyme-linked receptor protein signaling pathway / Translocation of ZAP-70 to Immunological synapse / Phosphorylation of CD3 and TCR zeta chains / regulation of calcium ion transport / macrophage differentiation / Generation of second messenger molecules / T cell differentiation / PD-1 signaling / positive regulation of protein kinase activity / Binding and entry of HIV virion /  coreceptor activity / positive regulation of calcium-mediated signaling / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / coreceptor activity / positive regulation of calcium-mediated signaling / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering /  protein tyrosine kinase binding / positive regulation of establishment of T cell polarity / positive regulation of interleukin-2 production / virus-mediated perturbation of host defense response / protein tyrosine kinase binding / positive regulation of establishment of T cell polarity / positive regulation of interleukin-2 production / virus-mediated perturbation of host defense response /  T細胞 / host cell endosome membrane / Vpu mediated degradation of CD4 / calcium-mediated signaling / clathrin-coated endocytic vesicle membrane / T細胞 / host cell endosome membrane / Vpu mediated degradation of CD4 / calcium-mediated signaling / clathrin-coated endocytic vesicle membrane /  cell surface receptor protein tyrosine kinase signaling pathway / positive regulation of T cell activation / positive regulation of peptidyl-tyrosine phosphorylation / transmembrane signaling receptor activity / Cargo recognition for clathrin-mediated endocytosis / Downstream TCR signaling / virus receptor activity / cell surface receptor protein tyrosine kinase signaling pathway / positive regulation of T cell activation / positive regulation of peptidyl-tyrosine phosphorylation / transmembrane signaling receptor activity / Cargo recognition for clathrin-mediated endocytosis / Downstream TCR signaling / virus receptor activity /  signaling receptor activity / signaling receptor activity /  Clathrin-mediated endocytosis / MHC class II protein complex binding / clathrin-dependent endocytosis of virus by host cell / positive regulation of canonical NF-kappaB signal transduction / defense response to Gram-negative bacterium / Clathrin-mediated endocytosis / MHC class II protein complex binding / clathrin-dependent endocytosis of virus by host cell / positive regulation of canonical NF-kappaB signal transduction / defense response to Gram-negative bacterium /  獲得免疫系 / positive regulation of MAPK cascade / positive regulation of viral entry into host cell / positive regulation of ERK1 and ERK2 cascade / cell surface receptor signaling pathway / 獲得免疫系 / positive regulation of MAPK cascade / positive regulation of viral entry into host cell / positive regulation of ERK1 and ERK2 cascade / cell surface receptor signaling pathway /  viral protein processing / viral protein processing /  エンドソーム / エンドソーム /  細胞接着 / 細胞接着 /  免疫応答 / positive regulation of protein phosphorylation / 免疫応答 / positive regulation of protein phosphorylation /  脂質ラフト / fusion of virus membrane with host plasma membrane / 脂質ラフト / fusion of virus membrane with host plasma membrane /  小胞体 / external side of plasma membrane / fusion of virus membrane with host endosome membrane / 小胞体 / external side of plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / エンベロープ (ウイルス) /  lipid binding / structural molecule activity / virion attachment to host cell / endoplasmic reticulum membrane / lipid binding / structural molecule activity / virion attachment to host cell / endoplasmic reticulum membrane /  protein kinase binding / host cell plasma membrane / virion membrane / positive regulation of DNA-templated transcription / protein kinase binding / host cell plasma membrane / virion membrane / positive regulation of DNA-templated transcription /  enzyme binding / enzyme binding /  シグナル伝達 / protein homodimerization activity / zinc ion binding / identical protein binding / シグナル伝達 / protein homodimerization activity / zinc ion binding / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
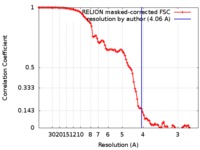
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.06 Å クライオ電子顕微鏡法 / 解像度: 4.06 Å | |||||||||
 データ登録者 データ登録者 | Barnes CO / Bjorkman PJ | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2018 ジャーナル: Cell Host Microbe / 年: 2018タイトル: Partially Open HIV-1 Envelope Structures Exhibit Conformational Changes Relevant for Coreceptor Binding and Fusion. 著者: Haoqing Wang / Christopher O Barnes / Zhi Yang / Michel C Nussenzweig / Pamela J Bjorkman /  要旨: HIV-1 Env, a trimer of gp120-gp41 heterodimers, mediates membrane fusion after binding host receptor CD4. Receptor binding displaces V1V2 loops from Env's apex, allowing coreceptor binding and ...HIV-1 Env, a trimer of gp120-gp41 heterodimers, mediates membrane fusion after binding host receptor CD4. Receptor binding displaces V1V2 loops from Env's apex, allowing coreceptor binding and opening Env to enable gp41-mediated fusion. We present 3.54 Å and 4.06 Å cryoelectron microscopy structures of partially open soluble native-like Env trimers (SOSIPs) bound to CD4. One structure, a complex with a coreceptor-mimicking antibody that binds both CD4 and gp120, stabilizes the displaced V1V2 and reveals its structure. Comparing partially and fully open Envs with closed Envs shows that gp41 rearrangements are independent of the CD4-induced rearrangements that result in V1V2 displacement and formation of a 4-stranded bridging sheet. These findings suggest ordered conformational changes before coreceptor binding: (1) gp120 opening inducing side-chain rearrangements and a compact gp41 central helix conformation, and (2) 4-stranded bridging-sheet formation and V1V2 displacement. These analyses illuminate potential receptor-induced Env changes and inform design of therapeutics disrupting viral entry. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9038.map.gz emd_9038.map.gz | 85.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9038-v30.xml emd-9038-v30.xml emd-9038.xml emd-9038.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9038_fsc.xml emd_9038_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9038.png emd_9038.png | 96.9 KB | ||
| マスクデータ |  emd_9038_msk_1.map emd_9038_msk_1.map emd_9038_msk_2.map emd_9038_msk_2.map | 91.1 MB 91.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_9038_additional.map.gz emd_9038_additional.map.gz | 85.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9038 http://ftp.pdbj.org/pub/emdb/structures/EMD-9038 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9038 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9038 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9038.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9038.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of complex used for model building and refinement. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_9038_msk_1.map emd_9038_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
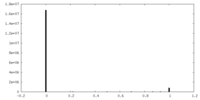
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_9038_msk_2.map emd_9038_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
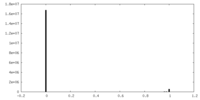
| 密度ヒストグラム |
-追加マップ: Sharpened map of focused refinement of 21c, gp120,...
| ファイル | emd_9038_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of focused refinement of 21c, gp120, and sCD4. Used for model building and refinement of V1V2 region, 21c-gp120 interface, and 21c-sCD4 interface. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
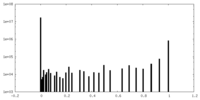
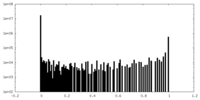
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : B41 SOSIP-sCD4-21c-8ANC195 complex
| 全体 | 名称: B41 SOSIP-sCD4-21c-8ANC195 complex |
|---|---|
| 要素 |
|
-超分子 #1: B41 SOSIP-sCD4-21c-8ANC195 complex
| 超分子 | 名称: B41 SOSIP-sCD4-21c-8ANC195 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#7 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 700 KDa |
-分子 #1: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.357824 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: AVGLGAFILG FLGAAGSTMG AASMALTVQA RLLLSGIVQQ QNNLLRAPEA QQHMLQLTVW GIKQLQARVL AVERYLRDQQ LLGIWGCSG KIICCTNVPW NDSWSNKTIN EIWDNMTWMQ WEKEIDNYTQ HIYTLLEVSQ IQQEKNEQEL LELD |
-分子 #2: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 57.702469 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAAKKW VTVYYGVPVW KEATTTLFCA SDAKAYDTEV HNVWATHACV PTDPNPQEI VLGNVTENFN MWKNNMVEQM HEDIISLWDQ SLKPCVKLTP LCVTLNCNNV NTNNTNNSTN ATISDWEKME T GEMKNCSF ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAAKKW VTVYYGVPVW KEATTTLFCA SDAKAYDTEV HNVWATHACV PTDPNPQEI VLGNVTENFN MWKNNMVEQM HEDIISLWDQ SLKPCVKLTP LCVTLNCNNV NTNNTNNSTN ATISDWEKME T GEMKNCSF NVTTSIRDKI KKEYALFYKL DVVPLENKNN INNTNITNYR LINCNTSVIT QACPKVSFEP IPIHYCAPAG FA ILKCNSK TFNGSGPCTN VSTVQCTHGI RPVVSTQLLL NGSLAEEEIV IRSENITDNA KTIIVQLNEA VEINCTRPNN NTR KSIHIG PGRAFYATGD IIGNIRQAHC NISKARWNET LGQIVAKLEE QFPNKTIIFN HSSGGDPEIV THSFNCGGEF FYCN TTPLF NSTWNNTRTD DYPTGGEQNI TLQCRIKQII NMWQGVGKAM YAPPIRGQIR CSSNITGLLL TRDGGRDQNG TETFR PGGG NMRDNWRSEL YKYKVVKIEP LGIAPTACKR RV |
-分子 #3: T-cell surface glycoprotein CD4
| 分子 | 名称: T-cell surface glycoprotein CD4 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 20.50326 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: KKVVLGKKGD TVELTCTASQ KKSIQFHWKN SNQIKILGNQ GSFLTKGPSK LNDRADSRRS LWDQGNFPLI IKNLKIEDSD TYICEVEDQ KEEVQLLVFG LTANSDTHLL QGQSLTLTLE SPPGSSPSVQ CRSPRGKNIQ GGKTLSVSQL ELQDSGTWTC T VLQNQKKV EFKIDIVVLA FQKASNT |
-分子 #4: 21c Fab VH domain
| 分子 | 名称: 21c Fab VH domain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.61959 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQVVQSGAE VRKPGASVKV SCKVSGFTLT GLSIHWVRQA PGKGLEWMGG FGPEENEIIY AQKFQGRVSM TEDTSTNTAY MELSSLRSE DTAVYYCATG GNYYNLWTGY YPLAYWGQGT LVTVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS ...文字列: QVQVVQSGAE VRKPGASVKV SCKVSGFTLT GLSIHWVRQA PGKGLEWMGG FGPEENEIIY AQKFQGRVSM TEDTSTNTAY MELSSLRSE DTAVYYCATG GNYYNLWTGY YPLAYWGQGT LVTVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKKVEPKSC DKT |
-分子 #5: 21c Fab VL domain
| 分子 | 名称: 21c Fab VL domain / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.815264 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSVLTQPPSV SAAPGQKVTI SCSGSSSNIG KNYVSWYQQL PGAAPKLLIF DDTQRPSGIP DRFSGSKSGT SATLAITGLQ TGDEADYYC GTWDSSLSTG QLFGGGTKLT VLGQPKAAPS VTLFPPSSEE LQANKATLVC LISDFYPGAV TVAWKADSSP V KAGVETTT ...文字列: QSVLTQPPSV SAAPGQKVTI SCSGSSSNIG KNYVSWYQQL PGAAPKLLIF DDTQRPSGIP DRFSGSKSGT SATLAITGLQ TGDEADYYC GTWDSSLSTG QLFGGGTKLT VLGQPKAAPS VTLFPPSSEE LQANKATLVC LISDFYPGAV TVAWKADSSP V KAGVETTT PSKQSNNKYA ASSYLSLTPE QWKSHRSYSC QVTHEGSTVE KTMAHAECS |
-分子 #6: 8ANC195 G52K5 Fab VH domain
| 分子 | 名称: 8ANC195 G52K5 Fab VH domain / タイプ: protein_or_peptide / ID: 6 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.12527 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QIHLVQSGTE VKKPGSSVTV SCKAYGVNTF GLYAVNWVRQ APGQSLEYIG QIWRWKSSAS HHFRGRVLIS AVDLTGSSPP ISSLEIKNL TSDDTAVYFC TTTSTYDKWS GLHHDGVMAF SSWGQGTLIS VSAASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP ...文字列: QIHLVQSGTE VKKPGSSVTV SCKAYGVNTF GLYAVNWVRQ APGQSLEYIG QIWRWKSSAS HHFRGRVLIS AVDLTGSSPP ISSLEIKNL TSDDTAVYFC TTTSTYDKWS GLHHDGVMAF SSWGQGTLIS VSAASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP VTVSWNSGAL TSGVHTFPAV LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK RVEPKSCDKT HH HHHH |
-分子 #7: 8ANC195 G52K5 Fab VL domain
| 分子 | 名称: 8ANC195 G52K5 Fab VL domain / タイプ: protein_or_peptide / ID: 7 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.401984 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPST LSASIGDTVR ISCRASQSIT GNWVAWYQQR PGKAPRLLIY RGAALLGGVP SRFSGSAAGT DFTLTIGNLQ AEDFGTFYC QQYDTYPGTF GQGTKVEVKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: DIQMTQSPST LSASIGDTVR ISCRASQSIT GNWVAWYQQR PGKAPRLLIY RGAALLGGVP SRFSGSAAGT DFTLTIGNLQ AEDFGTFYC QQYDTYPGTF GQGTKVEVKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #13: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 13 / コピー数: 27 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.35 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: 0.26 mA | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.7 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.7 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 2531 / 平均露光時間: 10.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6edu: |
 ムービー
ムービー コントローラー
コントローラー
































 Z
Z Y
Y X
X