+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8977 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
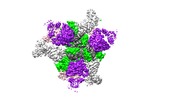
| タイトル | Cryo-EM structure at 3.2 A resolution of HIV-1 fusion peptide-directed antibody, A12V163-b.01, elicited by vaccination of Rhesus macaques, in complex with stabilized HIV-1 Env BG505 DS-SOSIP, which was also bound to antibodies VRC03 and PGT122 | |||||||||
 マップデータ マップデータ | primary map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / エンベロープ (ウイルス) ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / structural molecule activity / virion attachment to host cell / host cell plasma membrane / virion membrane / identical protein binding / エンベロープ (ウイルス) / structural molecule activity / virion attachment to host cell / host cell plasma membrane / virion membrane / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /   Macaca mulatta (アカゲザル) / Macaca mulatta (アカゲザル) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
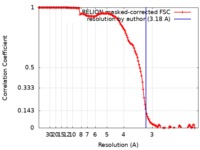
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.18 Å クライオ電子顕微鏡法 / 解像度: 3.18 Å | |||||||||
 データ登録者 データ登録者 | Acharya P / Eng ET / Kwong PD | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2019 ジャーナル: Cell / 年: 2019タイトル: Antibody Lineages with Vaccine-Induced Antigen-Binding Hotspots Develop Broad HIV Neutralization. 著者: Rui Kong / Hongying Duan / Zizhang Sheng / Kai Xu / Priyamvada Acharya / Xuejun Chen / Cheng Cheng / Adam S Dingens / Jason Gorman / Mallika Sastry / Chen-Hsiang Shen / Baoshan Zhang / ...著者: Rui Kong / Hongying Duan / Zizhang Sheng / Kai Xu / Priyamvada Acharya / Xuejun Chen / Cheng Cheng / Adam S Dingens / Jason Gorman / Mallika Sastry / Chen-Hsiang Shen / Baoshan Zhang / Tongqing Zhou / Gwo-Yu Chuang / Cara W Chao / Ying Gu / Alexander J Jafari / Mark K Louder / Sijy O'Dell / Ariana P Rowshan / Elise G Viox / Yiran Wang / Chang W Choi / Martin M Corcoran / Angela R Corrigan / Venkata P Dandey / Edward T Eng / Hui Geng / Kathryn E Foulds / Yicheng Guo / Young D Kwon / Bob Lin / Kevin Liu / Rosemarie D Mason / Martha C Nason / Tiffany Y Ohr / Li Ou / Reda Rawi / Edward K Sarfo / Arne Schön / John P Todd / Shuishu Wang / Hui Wei / Winston Wu / / James C Mullikin / Robert T Bailer / Nicole A Doria-Rose / Gunilla B Karlsson Hedestam / Diana G Scorpio / Julie Overbaugh / Jesse D Bloom / Bridget Carragher / Clinton S Potter / Lawrence Shapiro / Peter D Kwong / John R Mascola /   要旨: The vaccine-mediated elicitation of antibodies (Abs) capable of neutralizing diverse HIV-1 strains has been a long-standing goal. To understand how broadly neutralizing antibodies (bNAbs) can be ...The vaccine-mediated elicitation of antibodies (Abs) capable of neutralizing diverse HIV-1 strains has been a long-standing goal. To understand how broadly neutralizing antibodies (bNAbs) can be elicited, we identified, characterized, and tracked five neutralizing Ab lineages targeting the HIV-1-fusion peptide (FP) in vaccinated macaques over time. Genetic and structural analyses revealed two of these lineages to belong to a reproducible class capable of neutralizing up to 59% of 208 diverse viral strains. B cell analysis indicated each of the five lineages to have been initiated and expanded by FP-carrier priming, with envelope (Env)-trimer boosts inducing cross-reactive neutralization. These Abs had binding-energy hotspots focused on FP, whereas several FP-directed Abs induced by immunization with Env trimer-only were less FP-focused and less broadly neutralizing. Priming with a conserved subregion, such as FP, can thus induce Abs with binding-energy hotspots coincident with the target subregion and capable of broad neutralization. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8977.map.gz emd_8977.map.gz | 14.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8977-v30.xml emd-8977-v30.xml emd-8977.xml emd-8977.xml | 27.3 KB 27.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8977_fsc.xml emd_8977_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8977.png emd_8977.png | 154 KB | ||
| その他 |  emd_8977_additional.map.gz emd_8977_additional.map.gz | 14 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8977 http://ftp.pdbj.org/pub/emdb/structures/EMD-8977 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8977 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8977 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6mpgMC  9189C  9319C  9320C  9359C  6mphC  6mqcC  6mqeC  6mqmC  6mqrC  6mqsC  6n16C  6n1vC  6n1wC  6nf2C  6osyC  6ot1C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8977.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8977.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Refined map from cryoSparc 0.6.5
| ファイル | emd_8977_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refined map from cryoSparc 0.6.5 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
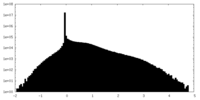
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Quaternary complex of HIV-1 Env BG505 DS-SOSIP with antibodies A1...
+超分子 #1: Quaternary complex of HIV-1 Env BG505 DS-SOSIP with antibodies A1...
+超分子 #2: HIV-1 Env BG505 DS-SOSIP gp120
+超分子 #3: HIV-1 Env BG505 DS-SOSIP gp41
+超分子 #4: A12V163-b.01
+超分子 #5: VRC03
+超分子 #6: PGT122
+分子 #1: HIV-1 Env BG505 DS-SOSIP gp120
+分子 #2: HIV-1 Env BG505 DS-SOSIP gp41
+分子 #3: A12V163-b.01 Heavy chain
+分子 #4: A12V163-b.01 Light chain
+分子 #5: VRC03 heavy chain
+分子 #6: VRC03 light chain
+分子 #7: PGT122 heavy chain
+分子 #8: PGT122 light chain
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 298 K / 装置: SPOTITON |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 0.8 µm / 倍率(補正後): 22500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 69.98 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6mpg: |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X