+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8900 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain reconstruction of human autophagy associated protein, ATG2A | |||||||||
 マップデータ マップデータ | Negative stain reconstruction of human ATG2A protein | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle membrane contact site / lipid transfer activity / positive regulation of autophagosome assembly / nucleophagy / piecemeal microautophagy of the nucleus / phagophore assembly site membrane /  phosphatidylinositol-3-phosphate binding / phagophore assembly site / reticulophagy / phosphatidylinositol-3-phosphate binding / phagophore assembly site / reticulophagy /  autophagosome assembly ...organelle membrane contact site / lipid transfer activity / positive regulation of autophagosome assembly / nucleophagy / piecemeal microautophagy of the nucleus / phagophore assembly site membrane / autophagosome assembly ...organelle membrane contact site / lipid transfer activity / positive regulation of autophagosome assembly / nucleophagy / piecemeal microautophagy of the nucleus / phagophore assembly site membrane /  phosphatidylinositol-3-phosphate binding / phagophore assembly site / reticulophagy / phosphatidylinositol-3-phosphate binding / phagophore assembly site / reticulophagy /  autophagosome assembly / autophagosome assembly /  lipid droplet / endoplasmic reticulum membrane lipid droplet / endoplasmic reticulum membrane類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 29.4 Å ネガティブ染色法 / 解像度: 29.4 Å | |||||||||
 データ登録者 データ登録者 | Chowdhury S / Otomo C / Leitner A / Ohashi K / Aebersold R / Lander GC / Otomo T | |||||||||
| 資金援助 |  米国, European Union, 2件 米国, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Insights into autophagosome biogenesis from structural and biochemical analyses of the ATG2A-WIPI4 complex. 著者: Saikat Chowdhury / Chinatsu Otomo / Alexander Leitner / Kazuto Ohashi / Ruedi Aebersold / Gabriel C Lander / Takanori Otomo /   要旨: Autophagy is an enigmatic cellular process in which double-membrane compartments, called "autophagosomes, form de novo adjacent to the endoplasmic reticulum (ER) and package cytoplasmic contents for ...Autophagy is an enigmatic cellular process in which double-membrane compartments, called "autophagosomes, form de novo adjacent to the endoplasmic reticulum (ER) and package cytoplasmic contents for delivery to lysosomes. Expansion of the precursor membrane phagophore requires autophagy-related 2 (ATG2), which localizes to the PI3P-enriched ER-phagophore junction. We combined single-particle electron microscopy, chemical cross-linking coupled with mass spectrometry, and biochemical analyses to characterize human ATG2A in complex with the PI3P effector WIPI4. ATG2A is a rod-shaped protein that can bridge neighboring vesicles through interactions at each of its tips. WIPI4 binds to one of the tips, enabling the ATG2A-WIPI4 complex to tether a PI3P-containing vesicle to another PI3P-free vesicle. These data suggest that the ATG2A-WIPI4 complex mediates ER-phagophore association and/or tethers vesicles to the ER-phagophore junction, establishing the required organization for phagophore expansion via the transfer of lipid membranes from the ER and/or the vesicles to the phagophore. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8900.map.gz emd_8900.map.gz | 249.7 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8900-v30.xml emd-8900-v30.xml emd-8900.xml emd-8900.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8900.png emd_8900.png | 26.4 KB | ||
| その他 |  emd_8900_additional.map.gz emd_8900_additional.map.gz | 2.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8900 http://ftp.pdbj.org/pub/emdb/structures/EMD-8900 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8900 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8900 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8900.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8900.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain reconstruction of human ATG2A protein | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened negative stain EM map of human ATG2A protein
| ファイル | emd_8900_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened negative stain EM map of human ATG2A protein | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
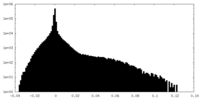
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human ATG2A protein
| 全体 | 名称: Human ATG2A protein |
|---|---|
| 要素 |
|
-超分子 #1: Human ATG2A protein
| 超分子 | 名称: Human ATG2A protein / タイプ: complex / ID: 1 / 親要素: 0 / 詳細: Full length protein |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)組換株: 9 |
| 分子量 | 理論値: 200 KDa |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate |
| グリッド | モデル: Maxtaform / 材質: COPPER/RHODIUM / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 2.0 µm / 最小 デフォーカス(補正後): 0.62 µm / 倍率(補正後): 52000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 温度 | 最低: 273.15 K / 最高: 298.15 K |
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 15.6 µm / 撮影したグリッド数: 1 / 実像数: 590 / 平均露光時間: 0.42 sec. / 平均電子線量: 20.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 37021 |
|---|---|
| CTF補正 | ソフトウェア - 名称: RELION (ver. 1.4) |
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: 詳細: A negative stain reconstruction of human ATG2-WIPI4 complex was low pass filtered to 60 Angstrom and used as initial model. |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 29.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.4) / 使用した粒子像数: 6386 |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X