+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8865 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative-stain reconstruction of an N-terminally MBP tagged Poz1 in the S. pombe CTP complex (Ccq1 2-716, Tpz1 406-508, Poz1 2-249) | |||||||||
 マップデータ マップデータ | Negative-stain reconstruction of an N-terminally MBP tagged Poz1 in the S. pombe CTP complex (Ccq1 2-716, Tpz1 406-508, Poz1 2-249) | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報telomere-telomerase complex assembly / telomere cap complex /  chromosome, telomeric repeat region / meiotic chromosome segregation / meiotic telomere clustering / chromosome, telomeric repeat region / meiotic chromosome segregation / meiotic telomere clustering /  chromosome, subtelomeric region / mitotic telomere maintenance via semi-conservative replication / chromosome, subtelomeric region / mitotic telomere maintenance via semi-conservative replication /  テロメア / telomere capping / protein localization to chromosome, telomeric region ...telomere-telomerase complex assembly / telomere cap complex / テロメア / telomere capping / protein localization to chromosome, telomeric region ...telomere-telomerase complex assembly / telomere cap complex /  chromosome, telomeric repeat region / meiotic chromosome segregation / meiotic telomere clustering / chromosome, telomeric repeat region / meiotic chromosome segregation / meiotic telomere clustering /  chromosome, subtelomeric region / mitotic telomere maintenance via semi-conservative replication / chromosome, subtelomeric region / mitotic telomere maintenance via semi-conservative replication /  テロメア / telomere capping / protein localization to chromosome, telomeric region / telomeric DNA binding / telomere maintenance via telomerase / telomere organization / テロメア / telomere capping / protein localization to chromosome, telomeric region / telomeric DNA binding / telomere maintenance via telomerase / telomere organization /  telomere maintenance / molecular adaptor activity / telomere maintenance / molecular adaptor activity /  DNA binding / DNA binding /  核質 / 核質 /  細胞核 / 細胞核 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Schizosaccharomyces pombe (分裂酵母) Schizosaccharomyces pombe (分裂酵母) | |||||||||
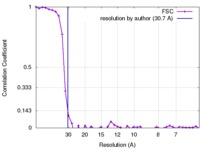
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 30.7 Å ネガティブ染色法 / 解像度: 30.7 Å | |||||||||
 データ登録者 データ登録者 | Scott HW / Kim JK / Yu C / Huang L / Qiao F / Taylor DJ | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2017 ジャーナル: J Mol Biol / 年: 2017タイトル: Spatial Organization and Molecular Interactions of the Schizosaccharomyces pombe Ccq1-Tpz1-Poz1 Shelterin Complex. 著者: Harry Scott / Jin-Kwang Kim / Clinton Yu / Lan Huang / Feng Qiao / Derek J Taylor /  要旨: The shelterin complex is a macromolecular assembly of proteins that binds to and protects telomeric DNA, which composes the ends of all linear chromosomes. Shelterin proteins prevent chromosome ends ...The shelterin complex is a macromolecular assembly of proteins that binds to and protects telomeric DNA, which composes the ends of all linear chromosomes. Shelterin proteins prevent chromosome ends from fusing together and from eliciting erroneous induction of DNA damage response pathways. In addition, shelterin proteins play key roles in regulating the recruitment and activation of telomerase, an enzyme that extends telomeric DNA. In fission yeast, Schizosaccharomyces pombe, interactions between the shelterin proteins Ccq1, Tpz1, and Poz1 are important for regulating telomerase-mediated telomere synthesis and thus telomere length homeostasis. Here, we used electron microscopy combined with genetic labeling to define the three-dimensional arrangement of the S. pombe Ccq1-Tpz1-Poz1 (CTP) complex. Crosslinking mass spectrometry was used to identify individual residues that are in proximity to the protein-protein interfaces of the assembled CTP complex. Together, our data provide a first glimpse into the architectural design of the CTP complex and reveals unique interactions that are important in maintaining the S. pombe telomere in a non-extendible state. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8865.map.gz emd_8865.map.gz | 2.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8865-v30.xml emd-8865-v30.xml emd-8865.xml emd-8865.xml | 12.4 KB 12.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8865_fsc.xml emd_8865_fsc.xml | 4.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8865.png emd_8865.png | 11.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8865 http://ftp.pdbj.org/pub/emdb/structures/EMD-8865 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8865 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8865 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8865.map.gz / 形式: CCP4 / 大きさ: 3.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8865.map.gz / 形式: CCP4 / 大きさ: 3.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative-stain reconstruction of an N-terminally MBP tagged Poz1 in the S. pombe CTP complex (Ccq1 2-716, Tpz1 406-508, Poz1 2-249) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.004 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Negative-stain reconstruction of an N-terminally MBP tagged Poz1 ...
| 全体 | 名称: Negative-stain reconstruction of an N-terminally MBP tagged Poz1 in the S. pombe CTP complex (Ccq1 2-716, Tpz1 406-508, Poz1 2-249) |
|---|---|
| 要素 |
|
-超分子 #1: Negative-stain reconstruction of an N-terminally MBP tagged Poz1 ...
| 超分子 | 名称: Negative-stain reconstruction of an N-terminally MBP tagged Poz1 in the S. pombe CTP complex (Ccq1 2-716, Tpz1 406-508, Poz1 2-249) タイプ: complex / ID: 1 / 親要素: 0 / 詳細: Complex is a dimer of trimers |
|---|---|
| 由来(天然) | 生物種:   Schizosaccharomyces pombe (分裂酵母) Schizosaccharomyces pombe (分裂酵母) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.064 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl acetate | ||||||||||||
| グリッド | 前処理 - タイプ: GLOW DISCHARGE / 詳細: 15 mA |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 特殊光学系 | エネルギーフィルター - 名称: omega in-column |
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サイズ - 横: 4000 pixel / デジタル化 - サイズ - 縦: 4000 pixel / デジタル化 - サンプリング間隔: 15.6 µm / 平均電子線量: 25.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)