+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8748 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM reconstruction of the Cafeteria roenbergensis virus capsid suggests a novel assembly pathway for giant viruses | |||||||||
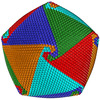
 マップデータ マップデータ | Cryo-EM reconstruction of Cafeteria roenbergensis virus with icosahedral averaging | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Cafeteria roenbergensis virus BV-PW1 (ウイルス) Cafeteria roenbergensis virus BV-PW1 (ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 21.0 Å クライオ電子顕微鏡法 / 解像度: 21.0 Å | |||||||||
 データ登録者 データ登録者 | Xiao C / Fischer MG / Bolotaulo DM / Ulloa-Rondeau N / Avila GA / Suttle CA | |||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2017 ジャーナル: Sci Rep / 年: 2017タイトル: Cryo-EM reconstruction of the Cafeteria roenbergensis virus capsid suggests novel assembly pathway for giant viruses. 著者: Chuan Xiao / Matthias G Fischer / Duer M Bolotaulo / Nancy Ulloa-Rondeau / Gustavo A Avila / Curtis A Suttle /    要旨: Whereas the protein composition and overall shape of several giant virus capsids have been described, the mechanism by which these large capsids assemble remains enigmatic. Here, we present a ...Whereas the protein composition and overall shape of several giant virus capsids have been described, the mechanism by which these large capsids assemble remains enigmatic. Here, we present a reconstruction of the capsid of Cafeteria roenbergensis virus (CroV), one of the largest viruses analyzed by cryo-electron microscopy (cryo-EM) to date. The CroV capsid has a diameter of 3,000 Å and a Triangulation number of 499. Unlike related mimiviruses, the CroV capsid is not decorated with glycosylated surface fibers, but features 30 Å-long surface protrusions that are formed by loops of the major capsid protein. Based on the orientation of capsomers in the cryo-EM reconstruction, we propose that the capsids of CroV and related giant viruses are assembled by a newly conceived assembly pathway that initiates at a five-fold vertex and continuously proceeds outwards in a spiraling fashion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8748.map.gz emd_8748.map.gz | 455.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8748-v30.xml emd-8748-v30.xml emd-8748.xml emd-8748.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8748_fsc.xml emd_8748_fsc.xml | 21.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8748.png emd_8748.png | 329.2 KB | ||
| マスクデータ |  emd_8748_msk_1.map emd_8748_msk_1.map emd_8748_msk_2.map emd_8748_msk_2.map | 524.1 MB 524.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_8748_half_map_1.map.gz emd_8748_half_map_1.map.gz emd_8748_half_map_2.map.gz emd_8748_half_map_2.map.gz | 299.8 MB 299.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8748 http://ftp.pdbj.org/pub/emdb/structures/EMD-8748 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8748 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8748 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8748.map.gz / 形式: CCP4 / 大きさ: 524.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8748.map.gz / 形式: CCP4 / 大きさ: 524.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of Cafeteria roenbergensis virus with icosahedral averaging | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.156 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_8748_msk_1.map emd_8748_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_8748_msk_2.map emd_8748_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Even map without soft masking of the reconstruction...
| ファイル | emd_8748_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Even map without soft masking of the reconstruction of Cafeteria roenbergensis virus with icosahedral averaging | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Odd map without soft masking of the reconstruction...
| ファイル | emd_8748_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Odd map without soft masking of the reconstruction of Cafeteria roenbergensis virus with icosahedral averaging | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cafeteria roenbergensis virus BV-PW1
| 全体 | 名称:   Cafeteria roenbergensis virus BV-PW1 (ウイルス) Cafeteria roenbergensis virus BV-PW1 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Cafeteria roenbergensis virus BV-PW1
| 超分子 | 名称: Cafeteria roenbergensis virus BV-PW1 / タイプ: virus / ID: 1 / 親要素: 0 詳細: The virus was purified following the protocol described in Fischer, M.G., et. al. The virion of Cafeteria roenbergensis virus (CroV) contains a complex suite of proteins for transcription and ...詳細: The virus was purified following the protocol described in Fischer, M.G., et. al. The virion of Cafeteria roenbergensis virus (CroV) contains a complex suite of proteins for transcription and DNA repair - Virology 466-467, 82-94 (2014). NCBI-ID: 693272 / 生物種: Cafeteria roenbergensis virus BV-PW1 / Sci species strain: BV-PW1 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Cafeteria roenbergensis (真核生物) Cafeteria roenbergensis (真核生物) |
| ウイルス殻 | Shell ID: 1 / 名称: CroV / 直径: 3000.0 Å / T番号(三角分割数): 499 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| グリッド | モデル: Quantifoil S7/2 / 材質: COPPER |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM300FEG/ST |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: KODAK SO-163 FILM / 平均電子線量: 25.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)