+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8578 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative Stain EM of C05 mutant Fab (VPGSGW) and CR9114 Fab in complex with H1 HA Trimer | |||||||||
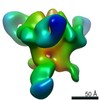
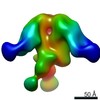
 マップデータ マップデータ | EM negative stain map of CR9114 and VPGSGW fabs in complex with H1 hemagglutinin trimer. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane類似検索 - 分子機能 | |||||||||
| 生物種 |   Influenza A virus (A/Solomon Islands/3/2006(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Solomon Islands/3/2006(H1N1)) (A型インフルエンザウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 18.0 Å ネガティブ染色法 / 解像度: 18.0 Å | |||||||||
 データ登録者 データ登録者 | Turner HL / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: In vitro evolution of an influenza broadly neutralizing antibody is modulated by hemagglutinin receptor specificity. 著者: Nicholas C Wu / Geramie Grande / Hannah L Turner / Andrew B Ward / Jia Xie / Richard A Lerner / Ian A Wilson /  要旨: The relatively recent discovery and characterization of human broadly neutralizing antibodies (bnAbs) against influenza virus provide valuable insights into antiviral and vaccine development. ...The relatively recent discovery and characterization of human broadly neutralizing antibodies (bnAbs) against influenza virus provide valuable insights into antiviral and vaccine development. However, the factors that influence the evolution of high-affinity bnAbs remain elusive. We therefore explore the functional sequence space of bnAb C05, which targets the receptor-binding site (RBS) of influenza haemagglutinin (HA) via a long CDR H3. We combine saturation mutagenesis with yeast display to enrich for C05 variants of CDR H3 that bind to H1 and H3 HAs. The C05 variants evolve up to 20-fold higher affinity but increase specificity to each HA subtype used in the selection. Structural analysis reveals that the fine specificity is strongly influenced by a highly conserved substitution that regulates receptor binding in different subtypes. Overall, this study suggests that subtle natural variations in the HA RBS between subtypes and species may differentially influence the evolution of high-affinity bnAbs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8578.map.gz emd_8578.map.gz | 10.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8578-v30.xml emd-8578-v30.xml emd-8578.xml emd-8578.xml | 9.2 KB 9.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8578.png emd_8578.png | 33.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8578 http://ftp.pdbj.org/pub/emdb/structures/EMD-8578 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8578 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8578 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8578.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8578.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM negative stain map of CR9114 and VPGSGW fabs in complex with H1 hemagglutinin trimer. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Negative stain EM map of C05 Mutant Fab (VPGSGW) and CR9114 fabs ...
| 全体 | 名称: Negative stain EM map of C05 Mutant Fab (VPGSGW) and CR9114 fabs in complex with H1 HA trimer. |
|---|---|
| 要素 |
|
-超分子 #1: Negative stain EM map of C05 Mutant Fab (VPGSGW) and CR9114 fabs ...
| 超分子 | 名称: Negative stain EM map of C05 Mutant Fab (VPGSGW) and CR9114 fabs in complex with H1 HA trimer. タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A/Solomon Islands/3/2006(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Solomon Islands/3/2006(H1N1)) (A型インフルエンザウイルス)株: A/Solomon Islands/3/2006(H1N1) |
| 組換発現 | 生物種:   Drosophila (ハエ) Drosophila (ハエ) |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.019 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 名称: Tris Buffer Saline / 詳細: Solution was made fresh using 0.2um filter. |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate 詳細: 0.2% Uranyl Formate. Samples were placed on 400 mesh copper grids covered with nitrocellulose. Samples were blotted off and UF was added for a total of 30 seconds before being blotted off. |
| グリッド | モデル: EMS / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 93 / 平均電子線量: 25.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 18874 |
|---|---|
| 初期 角度割当 | タイプ: COMMON LINE |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: SPARX |
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 18.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: sxali3d.py / 使用した粒子像数: 18874 |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















