+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8325 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
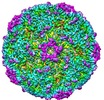
| タイトル | Beta-propiolactone treated coxsackievirus A16 empty procapsid | |||||||||
 マップデータ マップデータ | Beta-propiolactone treated coxsackievirus A16 empty procapsid | |||||||||
 試料 試料 | coxsackievirus A16 capsids != COXSACKIEVIRUS A16 coxsackievirus A16 capsids
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane /  : ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / : ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity /  ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / ピコルナイン2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane /  picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / cytoplasmic vesicle membrane /  : / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / : / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity /  RNA helicase activity / induction by virus of host autophagy / RNA helicase activity / induction by virus of host autophagy /  RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity / RNA依存性RNAポリメラーゼ / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /  RNA-dependent RNA polymerase activity / DNA-templated transcription / structural molecule activity / virion attachment to host cell / RNA-dependent RNA polymerase activity / DNA-templated transcription / structural molecule activity / virion attachment to host cell /  ATP hydrolysis activity / ATP hydrolysis activity /  タンパク質分解 / タンパク質分解 /  RNA binding / RNA binding /  ATP binding / ATP binding /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |    COXSACKIEVIRUS A16 (コクサッキーウイルス) COXSACKIEVIRUS A16 (コクサッキーウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.5 Å クライオ電子顕微鏡法 / 解像度: 6.5 Å | |||||||||
 データ登録者 データ登録者 | Chen F / Yao C | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2017 ジャーナル: J Virol / 年: 2017タイトル: Beta-Propiolactone Inactivation of Coxsackievirus A16 Induces Structural Alteration and Surface Modification of Viral Capsids. 著者: Chen Fan / Xiaohua Ye / Zhiqiang Ku / Liangliang Kong / Qingwei Liu / Cong Xu / Yao Cong / Zhong Huang /  要旨: Beta-propiolactone (BPL) is an inactivating agent that is widely used in the vaccine industry. However, its effects on vaccine protein antigens and its mechanisms of action remain poorly understood. ...Beta-propiolactone (BPL) is an inactivating agent that is widely used in the vaccine industry. However, its effects on vaccine protein antigens and its mechanisms of action remain poorly understood. Here we present cryo-electron microscopy (cryo-EM) structures of BPL-treated coxsackievirus A16 (CVA16) mature virions and procapsids at resolutions of 3.9 Å and 6.5 Å, respectively. Notably, both particles were found to adopt an expanded conformation resembling the 135S-like uncoating intermediate, with characteristic features including an opened 2-fold channel, the externalization of the N terminus of VP1 capsid protein, and the absence of pocket factor. However, major neutralizing epitopes are very well preserved on these particles. Further biochemical analyses revealed that BPL treatment impairs the abilities of CVA16 particles to bind to the attachment receptor heparan sulfate and to a conformation-dependent monoclonal antibody in a BPL dose-dependent manner, indicating that BPL is able to modify surface-exposed amino acid residues. Taken together, our results demonstrate that BPL treatment may induce alteration of the overall structure and surface properties of a nonenveloped viral capsid, thus revealing a novel mode of action of BPL. Beta-propiolactone (BPL) is commonly used as an inactivating reagent to produce viral vaccines. It is recognized that BPL inactivates viral infectivity through modification of viral nucleic acids. However, its effect on viral proteins remains largely unknown. Here, we present high-resolution cryo-EM structures of BPL-treated coxsackievirus A16 (CVA16) mature virions and procapsids, which reveals an expanded overall conformation and characteristic features that are typical for the 135S-like uncoating intermediate. We further show that the BPL concentration affects the binding of inactivated CVA16 particles to their receptor/antibody. Thus, BPL treatment can alter the overall structure and surface properties of viral capsids, which may lead to antigenic and immunogenic variations. Our findings provide important information for future development of BPL-inactivated vaccines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8325.map.gz emd_8325.map.gz | 192.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8325-v30.xml emd-8325-v30.xml emd-8325.xml emd-8325.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8325.png emd_8325.png | 313.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8325 http://ftp.pdbj.org/pub/emdb/structures/EMD-8325 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8325 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8325 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8325.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8325.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Beta-propiolactone treated coxsackievirus A16 empty procapsid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : coxsackievirus A16 capsids
| 全体 | 名称: coxsackievirus A16 capsids |
|---|---|
| 要素 |
|
-超分子 #1: COXSACKIEVIRUS A16
| 超分子 | 名称: COXSACKIEVIRUS A16 / タイプ: virus / ID: 1 / 親要素: 0 / NCBI-ID: 31704 / 生物種: COXSACKIEVIRUS A16 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 5 MDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.0032 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 構成要素 - 濃度: 0.15 M / 構成要素 - 式: PBS / 構成要素 - 名称: phosphate buffered saline |
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 120 K / 装置: FEI VITROBOT MARK III / 詳細: blot for 2 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 0.007 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 57000 Bright-field microscopy / Cs: 0.007 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 57000 |
| 特殊光学系 | 球面収差補正装置: Microscope was modified with a Cs corrector |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 70.0 K / 最高: 90.0 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - 画像ごとのフレーム数: 3-18 / 撮影したグリッド数: 1 / 実像数: 1350 / 平均露光時間: 1.1 sec. / 平均電子線量: 25.0 e/Å2 詳細: Images were recorded on a Falcon II direct electron detector in the 18-frame movie mode |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 1642 |
|---|---|
| CTF補正 | ソフトウェア - 名称: jspr 詳細: CTF fitting was automatically performed using fitctf2.py program in jspr |
| 初期モデル | モデルのタイプ: INSILICO MODEL 詳細: the data was performed the reference-free 2D analysis and initial model building utilizing EMAN2.1 |
| 初期 角度割当 | タイプ: COMMON LINE / ソフトウェア - 名称: jspr |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: jspr |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 6.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: jspr / 使用した粒子像数: 1642 ) / 解像度のタイプ: BY AUTHOR / 解像度: 6.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: jspr / 使用した粒子像数: 1642 |
 ムービー
ムービー コントローラー
コントローラー