+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8240 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EBOV GP in complex with IgG c2G4 and c13C6 Fabs | ||||||||||||
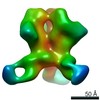
 マップデータ マップデータ | EBOV GP in complex with variable Fab domains of IgGs c2G4 and c13C6 | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell endoplasmic reticulum /  viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell /  脂質ラフト / fusion of virus membrane with host endosome membrane / 脂質ラフト / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) ...host cell endoplasmic reticulum / エンベロープ (ウイルス) ...host cell endoplasmic reticulum /  viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell /  脂質ラフト / fusion of virus membrane with host endosome membrane / 脂質ラフト / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / エンベロープ (ウイルス) /  lipid binding / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell plasma membrane / virion membrane / extracellular region / identical protein binding lipid binding / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell plasma membrane / virion membrane / extracellular region / identical protein binding類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Zaire ebolavirus (エボラウイルス) / Zaire ebolavirus (エボラウイルス) /   Zaire ebolavirus (strain Mayinga-76) (エボラウイルス) / Zaire ebolavirus (strain Mayinga-76) (エボラウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.3 Å クライオ電子顕微鏡法 / 解像度: 4.3 Å | ||||||||||||
 データ登録者 データ登録者 | Pallesen J / Murin CD / de Val N / Cottrell CA / Hastie KM / Turner HL / Fusco ML / Flyak AI / Zeitlin L / Crowe Jr JE ...Pallesen J / Murin CD / de Val N / Cottrell CA / Hastie KM / Turner HL / Fusco ML / Flyak AI / Zeitlin L / Crowe Jr JE / Andersen KG / Saphire EO / Ward AB | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2016 ジャーナル: Nat Microbiol / 年: 2016タイトル: Structures of Ebola virus GP and sGP in complex with therapeutic antibodies. 著者: Jesper Pallesen / Charles D Murin / Natalia de Val / Christopher A Cottrell / Kathryn M Hastie / Hannah L Turner / Marnie L Fusco / Andrew I Flyak / Larry Zeitlin / James E Crowe / Kristian G ...著者: Jesper Pallesen / Charles D Murin / Natalia de Val / Christopher A Cottrell / Kathryn M Hastie / Hannah L Turner / Marnie L Fusco / Andrew I Flyak / Larry Zeitlin / James E Crowe / Kristian G Andersen / Erica Ollmann Saphire / Andrew B Ward /  要旨: The Ebola virus (EBOV) GP gene encodes two glycoproteins. The major product is a soluble, dimeric glycoprotein (sGP) that is secreted abundantly. Despite the abundance of sGP during infection, little ...The Ebola virus (EBOV) GP gene encodes two glycoproteins. The major product is a soluble, dimeric glycoprotein (sGP) that is secreted abundantly. Despite the abundance of sGP during infection, little is known regarding its structure or functional role. A minor product, resulting from transcriptional editing, is the transmembrane-anchored, trimeric viral surface glycoprotein (GP). GP mediates attachment to and entry into host cells, and is the intended target of antibody therapeutics. Because large portions of sequence are shared between GP and sGP, it has been hypothesized that sGP may potentially subvert the immune response or may contribute to pathogenicity. In this study, we present cryo-electron microscopy structures of GP and sGP in complex with GP-specific and GP/sGP cross-reactive antibodies undergoing human clinical trials. The structure of the sGP dimer presented here, in complex with both an sGP-specific antibody and a GP/sGP cross-reactive antibody, permits us to unambiguously assign the oligomeric arrangement of sGP and compare its structure and epitope presentation to those of GP. We also provide biophysical evaluation of naturally occurring GP/sGP mutations that fall within the footprints identified by our high-resolution structures. Taken together, our data provide a detailed and more complete picture of the accessible Ebolavirus glycoprotein landscape and a structural basis to evaluate patient and vaccine antibody responses towards differently structured products of the GP gene. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8240.map.gz emd_8240.map.gz | 51.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8240-v30.xml emd-8240-v30.xml emd-8240.xml emd-8240.xml | 28.5 KB 28.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8240_fsc.xml emd_8240_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8240.png emd_8240.png | 151.1 KB | ||
| その他 |  emd_8240_additional_1.map.gz emd_8240_additional_1.map.gz emd_8240_additional_2.map.gz emd_8240_additional_2.map.gz emd_8240_additional_3.map.gz emd_8240_additional_3.map.gz | 42.8 MB 42.8 MB 50.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8240 http://ftp.pdbj.org/pub/emdb/structures/EMD-8240 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8240 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8240 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8240.map.gz / 形式: CCP4 / 大きさ: 55.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8240.map.gz / 形式: CCP4 / 大きさ: 55.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EBOV GP in complex with variable Fab domains of IgGs c2G4 and c13C6 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: EBOV GP in complex with variable Fab domains of IgGs c2G4 and c13C6
| ファイル | emd_8240_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EBOV GP in complex with variable Fab domains of IgGs c2G4 and c13C6 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: EBOV GP in complex with variable Fab domains of IgGs c2G4 and c13C6
| ファイル | emd_8240_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EBOV GP in complex with variable Fab domains of IgGs c2G4 and c13C6 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: EBOV GP in complex with variable Fab domains of IgGs c2G4 and c13C6
| ファイル | emd_8240_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EBOV GP in complex with variable Fab domains of IgGs c2G4 and c13C6 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ebola virus trimeric surface glycoprotein in complex with IgG c2G...
| 全体 | 名称: Ebola virus trimeric surface glycoprotein in complex with IgG c2G4 and c13C6 Fabs |
|---|---|
| 要素 |
|
-超分子 #1: Ebola virus trimeric surface glycoprotein in complex with IgG c2G...
| 超分子 | 名称: Ebola virus trimeric surface glycoprotein in complex with IgG c2G4 and c13C6 Fabs タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:   Zaire ebolavirus (エボラウイルス) / 株: Mayinga-76 Zaire ebolavirus (エボラウイルス) / 株: Mayinga-76 |
| 分子量 | 実験値: 540 KDa |
-分子 #1: Ebola surface glycoprotein, GP1
| 分子 | 名称: Ebola surface glycoprotein, GP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Zaire ebolavirus (strain Mayinga-76) (エボラウイルス) Zaire ebolavirus (strain Mayinga-76) (エボラウイルス)株: Mayinga-76 |
| 分子量 | 理論値: 50.974031 KDa |
| 組換発現 | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 配列 | 文字列: IPLGVIHNST LQVSDVDKLV CRDKLSSTNQ LRSVGLNLEG NGVATDVPSA TKRWGFRSGV PPKVVNYEAG EWAENCYNLE IKKPDGSEC LPAAPDGIRG FPRCRYVHKV SGTGPCAGDF AFHKEGAFFL YDRLASTVIY RGTTFAEGVV AFLILPQAKK D FFSSHPLR ...文字列: IPLGVIHNST LQVSDVDKLV CRDKLSSTNQ LRSVGLNLEG NGVATDVPSA TKRWGFRSGV PPKVVNYEAG EWAENCYNLE IKKPDGSEC LPAAPDGIRG FPRCRYVHKV SGTGPCAGDF AFHKEGAFFL YDRLASTVIY RGTTFAEGVV AFLILPQAKK D FFSSHPLR EPVNATEDPS SGYYSTTIRY QATGFGTNET EYLFEVDNLT YVQLESRFTP QFLLQLNETI YTSGKRSNTT GK LIWKVNP EIDTTIGEWA FWETKKNLTR KIRSEELSFT VVSNGAKNIS GQSPARTSSD PGTNTTTEDH KIMASENSSA MVQ VHSQGR EAAVSHLTTL ATISTSPQSL TTKPGPDNST HNTPVYKLDI SEATQVEQHH RRTDNDSTAS DTPSATTAAG PPKA ENTNT SKSTDFLDPA TTTSPQNHSE TAGNNNTHHQ DTGEESASSG KLGLITNTIA GVAGLITGGR RTRR |
-分子 #2: Ebola surface glycoprotein, GP2
| 分子 | 名称: Ebola surface glycoprotein, GP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Zaire ebolavirus (エボラウイルス) / 株: Mayinga-76 Zaire ebolavirus (エボラウイルス) / 株: Mayinga-76 |
| 分子量 | 理論値: 16.137119 KDa |
| 組換発現 | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
| 配列 | 文字列: EAIVNAQPKC NPNLHYWTTQ DEGAAIGLAW IPYFGPAAEG IYTEGLMHNQ DGLICGLRQL ANETTQALQL FLRATTELRT FSILNRKAI DFLLQRWGGT CHILGPDCCI EPHDWTKNIT DKIDQIIHDF VDKTLPDLEV DDDD |
-分子 #3: c2G4 variable Fab domain heavy chain
| 分子 | 名称: c2G4 variable Fab domain heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.573105 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: EVQLQESGGG LMQPGGSMKL SCVASGFTFS NYWMNWVRQS PEKGLEWVAE IRLKSNNYAT HYAESVKGRF TISRDDSKRS VYLQMNTLR AEDTGIYYCT RGNGNYRAMD YWGQGTSVTV S |
-分子 #4: c2G4 variable Fab domain light chain
| 分子 | 名称: c2G4 variable Fab domain light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.641884 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: DIQMTQSPAS LSVSVGETVS ITCRASENIY SSLAWYQQKQ GKSPQLLVYS ATILADGVPS RFSGSGSGTQ YSLKINSLQS EDFGTYYCQ HFWGTPYTFG GGTKLEIK |
-分子 #5: c13C6 variable Fab domain heavy chain
| 分子 | 名称: c13C6 variable Fab domain heavy chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.169679 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: DVKLLESGGG LVQPGGSLKL SCAASGFSLS TSGVGVGWFR QPSGKGLEWL ALIWWDDDKY YNPSLKSQLS ISKDFSRNQV FLKISNVDI ADTATYYCAR RDPFGYDNAM GYWGQGTSVT VS |
-分子 #6: c13C6 variable Fab domain light chain
| 分子 | 名称: c13C6 variable Fab domain light chain / タイプ: protein_or_peptide / ID: 6 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.611877 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: DIVMTQSPLS LSTSVGDRVS LTCKASQNVG TAVAWYQQKP GQSPKLLIYS ASNRYTGVPD RFTGSGSGTD FTLTISNMQS EDLADYFCQ QYSSYPLTFG AGTKLELR |
-分子 #11: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 11 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 名称: 1xTBS |
| グリッド | モデル: C-flat-2/2 4C / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 20.0 nm / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 0.0093 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 77.0 K / 最高: 77.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 2526 / 平均露光時間: 10.0 sec. / 平均電子線量: 57.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Model building and refinement were conducted using a combination of software programs. The refinement target was optimizing using the MolProbity score while maintaining a high (good) EMRinger score. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: EMRinger |
| 得られたモデル |  PDB-5kel: |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X



























