+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8104 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
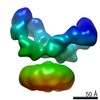
| タイトル | Cryo-EM structure of GluN1/GluN2B NMDA receptor in the DCKA/D-APV-bound conformation, state 5 | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ligand-gated ion channel (リガンド依存性イオンチャネル) / ligand-gated ion channel (リガンド依存性イオンチャネル) /  synaptic transmission / synaptic transmission /  SIGNALING PROTEIN SIGNALING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 NMDA glutamate receptor activity / NMDA selective glutamate receptor complex / response to zinc ion / response to magnesium ion / late endosome / NMDA glutamate receptor activity / NMDA selective glutamate receptor complex / response to zinc ion / response to magnesium ion / late endosome /  postsynaptic membrane / postsynaptic membrane /  リソソーム / リソソーム /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 14.1 Å クライオ電子顕微鏡法 / 解像度: 14.1 Å | |||||||||
 データ登録者 データ登録者 | Zhu S / Stein AR | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2016 ジャーナル: Cell / 年: 2016タイトル: Mechanism of NMDA Receptor Inhibition and Activation. 著者: Shujia Zhu / Richard A Stein / Craig Yoshioka / Chia-Hsueh Lee / April Goehring / Hassane S Mchaourab / Eric Gouaux /  要旨: N-methyl-D-aspartate receptors (NMDARs) are glutamate-gated, calcium-permeable ion channels that mediate synaptic transmission and underpin learning and memory. NMDAR dysfunction is directly ...N-methyl-D-aspartate receptors (NMDARs) are glutamate-gated, calcium-permeable ion channels that mediate synaptic transmission and underpin learning and memory. NMDAR dysfunction is directly implicated in diseases ranging from seizure to ischemia. Despite its fundamental importance, little is known about how the NMDAR transitions between inactive and active states and how small molecules inhibit or activate ion channel gating. Here, we report electron cryo-microscopy structures of the GluN1-GluN2B NMDA receptor in an ensemble of competitive antagonist-bound states, an agonist-bound form, and a state bound with agonists and the allosteric inhibitor Ro25-6981. Together with double electron-electron resonance experiments, we show how competitive antagonists rupture the ligand binding domain (LBD) gating "ring," how agonists retain the ring in a dimer-of-dimers configuration, and how allosteric inhibitors, acting within the amino terminal domain, further stabilize the LBD layer. These studies illuminate how the LBD gating ring is fundamental to signal transduction and gating in NMDARs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8104.map.gz emd_8104.map.gz | 13.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8104-v30.xml emd-8104-v30.xml emd-8104.xml emd-8104.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8104.png emd_8104.png | 71.1 KB | ||
| Filedesc metadata |  emd-8104.cif.gz emd-8104.cif.gz | 6.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8104 http://ftp.pdbj.org/pub/emdb/structures/EMD-8104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8104 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5iptMC  8097C  8098C  8101C  8102C  8103C  8105C  8106C  5iouC  5iovC  5ipqC  5iprC  5ipsC  5ipuC  5ipvC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8104.map.gz / 形式: CCP4 / 大きさ: 32.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8104.map.gz / 形式: CCP4 / 大きさ: 32.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : GluN1-GluN2B receptor in the complex with competitive antagonists...
| 全体 | 名称: GluN1-GluN2B receptor in the complex with competitive antagonists DCKA and D-APV |
|---|---|
| 要素 |
|
-超分子 #1: GluN1-GluN2B receptor in the complex with competitive antagonists...
| 超分子 | 名称: GluN1-GluN2B receptor in the complex with competitive antagonists DCKA and D-APV タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル) |
-分子 #1: N-methyl-D-aspartate receptor subunit NR1-8a
| 分子 | 名称: N-methyl-D-aspartate receptor subunit NR1-8a / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル) |
| 分子量 | 理論値: 92.651234 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DPKIVNIGAV LSTKKHEQIF REAVNQANFF HFTRKIQLNA TSVTHRPNAI QMALSVCEDL ISSQVYAILV SHPPAPTDHL TPTPISYTA GFYRIPVIGL TTRMSIYSDK SIHLSFLRTV PPYSHQALVW FEMMRLFNWN HVILIVSDDH EGRAAQKKLE T LLEEKESK ...文字列: DPKIVNIGAV LSTKKHEQIF REAVNQANFF HFTRKIQLNA TSVTHRPNAI QMALSVCEDL ISSQVYAILV SHPPAPTDHL TPTPISYTA GFYRIPVIGL TTRMSIYSDK SIHLSFLRTV PPYSHQALVW FEMMRLFNWN HVILIVSDDH EGRAAQKKLE T LLEEKESK ADKVLQFEPG TKNLTALLLE AKELEARVII LSASEDDATA VYKSAAMLDM TGAGYVWLVG EREISGSALR YA PDGIIGL QLINGKNESA HISDAVAVVA QAIHELFEME QITDPPRGCV GNTNIWKTGP LFKRVLMSSK YPDGVTGRIE FNE DGDRKF AQYSIMNLQN RKLVQVGIFD GSYIIQNDRK IIWPGGETER PQGYQMSTRL KIVTIHQEPF VYVRPTTSDG TCRE EYTIN GDPIKKVICN GPDETIPGRP TVPQCCYGFC VDLLIKLARE MDFTYEVHLV ADGKFGTQER VNNSNAAAWN GMMGE LLSG QADMIVAPLT INNERAQYIE FSKPFKYQGL TILVKKEIPR STLDSFMQPF QSTLWLLVGL SVHVVAVMLY LLDRFS PFG RFEDALTLSS AMWFSWRVLL NSGLGEGAPR SFSARILGMV WAGFAMIIVA SYTANLAAFL VLRRPEERIT GINDPRL RN PSDKFIYATV KQSSVDIYFR RQVELSTMYR HMEKHNYESA AEAIQAVRDN KLHAFIWDSA VLEFEASQKC DLVTTGEL F FRSGFGIGMR KDSPWKQEVS LNILKSHENG FMEELDKTWV RYQECDSRSN APATLTFENM AGVFMLVAGG IVAGIFLIF IEIAYKSRAE AKRMKGLEVL FQ UniProtKB: Glutamate receptor ionotropic, NMDA 1 |
-分子 #2: Ionotropic glutamate receptor subunit NR2B
| 分子 | 名称: Ionotropic glutamate receptor subunit NR2B / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル) |
| 分子量 | 理論値: 93.234742 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRPTEACCYL KISLIILFYS RAYAQKHPNM DIAVILVGTT EEVAIKDVHE KDDFHHLPVT PRVELVTMQE SDPKSIITRI CDLMSDKKV QGVVFGDDTD QEAIAQILDF ISVQTLTPIL GIHGGSSMIM ADKEEASMFF QFGPSIEQQA SVMLNIMEEY D WYIFSIVT ...文字列: MRPTEACCYL KISLIILFYS RAYAQKHPNM DIAVILVGTT EEVAIKDVHE KDDFHHLPVT PRVELVTMQE SDPKSIITRI CDLMSDKKV QGVVFGDDTD QEAIAQILDF ISVQTLTPIL GIHGGSSMIM ADKEEASMFF QFGPSIEQQA SVMLNIMEEY D WYIFSIVT TYFPGYQDFE NKVRSTIENS FVGWELEEVI HLDMSLDDID SKIQNQLKKL QSPVILLYCT KEEATYIFEV AH SVGLTGY GFTWIVPSLV AGDTDTVPDE FPTGLISVSY DEWDYDLPAR VRDGIAIITT AASTMLSEHN SIPQSKSSCN NIQ ESRVYE AHMLKRYLIN VTFEGRDLSF SEDGYQMHPK LVIILLNQER KWERVGKYKD RSLKMWPVFD LYPNSEEHKD EHLS IVTLE EAPFVIVEDV DPLSGTCMRN TVPCRKQIRP ENRTEEGGNY IKRCCKGFCI DILKKIAKTV KFTYDLYLVT NGKHG KKIN GVWNGMIGEV VTKRAYMAVG SLTINEERSE VVDFSVPFIE TGISVMVSRS NGTVSPSAFL EPFSADVWVM MFVMLL IVS AVAVFVFEYF SPVGYNGPSF TIGKAIWLLW GLVFNNSLPV QNPKGTTSKI MVSVWAFFAV IFLASYTANL AAFMIQR RY VDQVSGLSDK KFQRPNDFSP AFRFGTVPNG STERNIRNNY LEMHSYMVKF NQRSVQDALL SLKSGKLDAF IYDAAVLN Y MAGRDEGCKL VTIGSGKVFA TTGYGIAIQK DSGWKRQVDL AILQLFGDGE MEELEALWLT GICHNEKNEV MSSQLDIDN MAGVFYMLAA AMALSLITFI MEHLF UniProtKB: Glutamate receptor ionotropic, NMDA 2B |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 6.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 18 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 0.01 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / Cs: 0.01 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 10.5 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 393513 |
|---|---|
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 14.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.3) / 使用した粒子像数: 32608 |
 ムービー
ムービー コントローラー
コントローラー