+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7971 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of the mitochondrial calcium uniporter MCU from the fungus Cyphellophora europaea | |||||||||
 マップデータ マップデータ | Cryo-EM structure of the mitochondrial calcium uniporter | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  mitochondria (ミトコンドリア) / mitochondria (ミトコンドリア) /  calcium (カルシウム) / calcium (カルシウム) /  ion channel (イオンチャネル) / ion channel (イオンチャネル) /  eukaryotic (真核生物) / eukaryotic (真核生物) /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 uniporter activity / mitochondrial calcium ion homeostasis / uniporter activity / mitochondrial calcium ion homeostasis /  calcium channel activity / calcium channel activity /  ミトコンドリア内膜 / membrane => GO:0016020 / identical protein binding ミトコンドリア内膜 / membrane => GO:0016020 / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |  Cyphellophora europaea (菌類) / Cyphellophora europaea (菌類) /  Cyphellophora europaea CBS 101466 (菌類) Cyphellophora europaea CBS 101466 (菌類) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.2 Å クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Long SB / Baradaran R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Cryo-EM structures of fungal and metazoan mitochondrial calcium uniporters. 著者: Rozbeh Baradaran / Chongyuan Wang / Andrew Francis Siliciano / Stephen Barstow Long /  要旨: The mitochondrial calcium uniporter (MCU) is a highly selective calcium channel and a major route of calcium entry into mitochondria. How the channel catalyses ion permeation and achieves ...The mitochondrial calcium uniporter (MCU) is a highly selective calcium channel and a major route of calcium entry into mitochondria. How the channel catalyses ion permeation and achieves ion selectivity are not well understood, partly because MCU is thought to have a distinct architecture in comparison to other cellular channels. Here we report cryo-electron microscopy reconstructions of MCU channels from zebrafish and Cyphellophora europaea at 8.5 Å and 3.2 Å resolutions, respectively. In contrast to a previous report of pentameric stoichiometry for MCU, both channels are tetramers. The atomic model of C. europaea MCU shows that a conserved WDXXEP signature sequence forms the selectivity filter, in which calcium ions are arranged in single file. Coiled-coil legs connect the pore to N-terminal domains in the mitochondrial matrix. In C. europaea MCU, the N-terminal domains assemble as a dimer of dimers; in zebrafish MCU, they form an asymmetric crescent. The structures define principles that underlie ion permeation and calcium selectivity in this unusual channel. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7971.map.gz emd_7971.map.gz | 200.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7971-v30.xml emd-7971-v30.xml emd-7971.xml emd-7971.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7971.png emd_7971.png | 115.1 KB | ||
| Filedesc metadata |  emd-7971.cif.gz emd-7971.cif.gz | 6.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7971 http://ftp.pdbj.org/pub/emdb/structures/EMD-7971 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7971 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7971 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7971.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7971.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the mitochondrial calcium uniporter | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.861 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mitochondrial Calcium Uniporter (MCU) from the fungus Cyphellopho...
| 全体 | 名称: Mitochondrial Calcium Uniporter (MCU) from the fungus Cyphellophora europaea. |
|---|---|
| 要素 |
|
-超分子 #1: Mitochondrial Calcium Uniporter (MCU) from the fungus Cyphellopho...
| 超分子 | 名称: Mitochondrial Calcium Uniporter (MCU) from the fungus Cyphellophora europaea. タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Cyphellophora europaea (菌類) Cyphellophora europaea (菌類) |
| 分子量 | 理論値: 158.328 KDa |
-分子 #1: Mitochondrial calcium uniporter MCU
| 分子 | 名称: Mitochondrial calcium uniporter MCU / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Cyphellophora europaea CBS 101466 (菌類) Cyphellophora europaea CBS 101466 (菌類) |
| 分子量 | 理論値: 39.637176 KDa |
| 組換発現 | 生物種:   Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MTKGKLLTTP SRLLKLVLPL STVDHNTDRK DVAPLALLVH PQQPLSYLER LIQAELPPPD PQDSKSTTRS VTFRAMEAKD DEIKPRKKA DTEGGGGSDG SVQSYSGAGR EGEGKDEGEF VRWSPSTEIG DFIRDAARAK EFEVEIEGSP GVIKVAVPSF N DRTYYLRQ ...文字列: MTKGKLLTTP SRLLKLVLPL STVDHNTDRK DVAPLALLVH PQQPLSYLER LIQAELPPPD PQDSKSTTRS VTFRAMEAKD DEIKPRKKA DTEGGGGSDG SVQSYSGAGR EGEGKDEGEF VRWSPSTEIG DFIRDAARAK EFEVEIEGSP GVIKVAVPSF N DRTYYLRQ RLRRTSRKIS KLAAIKEECD KAAHRGAQRI ALAGCGGLIG YWYIVYRLTF ETDLGWDVME PVTYLVGLST LI GGYMWFL WHNREVSYRS ALNITVSARQ NKLYQAKGFS LQDWEGYLEE ANAMRREIKA VASEYDVDWN ETQDEGGDEK VTK ALRDER KNNNGTKNKS KEGEEDDEDD GEGEEF UniProtKB: Calcium uniporter protein |
-分子 #2: 1-[GLYCEROLYLPHOSPHONYL]-2-[8-(2-HEXYL-CYCLOPROPYL)-OCTANAL-1-YL]...
| 分子 | 名称: 1-[GLYCEROLYLPHOSPHONYL]-2-[8-(2-HEXYL-CYCLOPROPYL)-OCTANAL-1-YL]-3-[HEXADECANAL-1-YL]-GLYCEROL タイプ: ligand / ID: 2 / コピー数: 8 / 式: DGG |
|---|---|
| 分子量 | 理論値: 734.981 Da |
| Chemical component information | 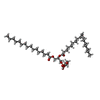 ChemComp-DGG: |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 3 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV / 詳細: 2 second blot, blot force of 0. | ||||||||||||||||||
| 詳細 | Monodisperse sample |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 29000 Bright-field microscopy / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 3 / 実像数: 6040 / 平均露光時間: 0.2 sec. / 平均電子線量: 2.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 547637 | ||||||
|---|---|---|---|---|---|---|---|
| 初期モデル | モデルのタイプ: INSILICO MODEL / In silico モデル: Generated using RELION 2.1 | ||||||
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT ソフトウェア:
| ||||||
| 最終 3次元分類 | クラス数: 1 / 平均メンバー数/クラス: 376541 / ソフトウェア - 名称: FREALIGN (ver. 1.0) / ソフトウェア - 詳細: cisTEM 1.0 | ||||||
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: FREALIGN (ver. 1.0) / ソフトウェア - 詳細: cisTEM 1.0 | ||||||
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C2 (2回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: FREALIGN / ソフトウェア - 詳細: cisTEM 1.0 / 使用した粒子像数: 376541 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: FREALIGN / ソフトウェア - 詳細: cisTEM 1.0 / 使用した粒子像数: 376541 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 136.58 / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6dnf: |
 ムービー
ムービー コントローラー
コントローラー















