+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7944 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Fako virus full particles, icosahedral reconstruction | |||||||||
 マップデータ マップデータ | An icosahedral reconstruction of Fako virus full particles | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  capsid (カプシド) / capsid (カプシド) /  virion (ウイルス) / virion (ウイルス) /  Reoviridae / T=2 / Reoviridae / T=2 /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 | Major capsid protein / Turret protein / Clamp protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Fako virus (ウイルス) Fako virus (ウイルス) | |||||||||
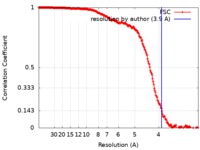
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Kaelber JT / Jiang W / Weaver SC / Auguste AJ / Chiu W | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2020 ジャーナル: Structure / 年: 2020タイトル: Arrangement of the Polymerase Complexes inside a Nine-Segmented dsRNA Virus. 著者: Jason T Kaelber / Wen Jiang / Scott C Weaver / Albert J Auguste / Wah Chiu /  要旨: Members of the family Reoviridae package several copies of the viral polymerase complex into their capsid to carry out replication and transcription within viral particles. Classical single-particle ...Members of the family Reoviridae package several copies of the viral polymerase complex into their capsid to carry out replication and transcription within viral particles. Classical single-particle reconstruction encounters difficulties resolving structures such as the intraparticle polymerase complex because refinement can converge to an incorrect map and because the map could depict a nonrepresentative subset of particles or an average of heterogeneous particles. Using the nine-segmented Fako virus, we tested hypotheses for the arrangement and number of polymerase complexes within the virion by measuring how well each hypothesis describes the set of cryoelectron microscopy images of individual viral particles. We find that the polymerase complex in Fako virus binds at ten possible sites despite having only nine genome segments. A single asymmetric configuration describes the arrangement of these complexes in both virions and genome-free capsids. Similarities between the arrangements of Reoviridae with 9, 10, and 11 segments indicate the generalizability of this architecture. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7944.map.gz emd_7944.map.gz | 685.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7944-v30.xml emd-7944-v30.xml emd-7944.xml emd-7944.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7944_fsc.xml emd_7944_fsc.xml | 20.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7944.png emd_7944.png | 219.9 KB | ||
| Filedesc metadata |  emd-7944.cif.gz emd-7944.cif.gz | 7.3 KB | ||
| その他 |  emd_7944_additional.map.gz emd_7944_additional.map.gz | 1 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7944 http://ftp.pdbj.org/pub/emdb/structures/EMD-7944 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7944 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7944 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7944.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7944.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | An icosahedral reconstruction of Fako virus full particles | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.506 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Another icosahedral reconstruction of Fako virus full particles
| ファイル | emd_7944_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Another icosahedral reconstruction of Fako virus full particles | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Fako virus
| 全体 | 名称:   Fako virus (ウイルス) Fako virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Fako virus
| 超分子 | 名称: Fako virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 1567404 / 生物種: Fako virus / Sci species strain: CSW77 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Culicinae (カ) Culicinae (カ) |
| 分子量 | 理論値: 43 MDa |
| ウイルス殻 | Shell ID: 1 / T番号(三角分割数): 2 |
-分子 #1: Clamp protein
| 分子 | 名称: Clamp protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Fako virus (ウイルス) / 株: CSW77 Fako virus (ウイルス) / 株: CSW77 |
| 分子量 | 理論値: 39.589691 KDa |
| 配列 | 文字列: MTLTYWDKEK RMTLKQMIQQ VAINEQENEL THYVFTTPLS MPTFGKPMLG YVPLNEVATS KFFSNVNDFD RDNQLAMAHF PDTTITQAY NLTNSIKPGD TSLPDAEVAA LKWFWKFFTS INLVRQPPMD NVMYWACQFL SSGTSFLPLE RDVEIVFSGF K GSHICMFS ...文字列: MTLTYWDKEK RMTLKQMIQQ VAINEQENEL THYVFTTPLS MPTFGKPMLG YVPLNEVATS KFFSNVNDFD RDNQLAMAHF PDTTITQAY NLTNSIKPGD TSLPDAEVAA LKWFWKFFTS INLVRQPPMD NVMYWACQFL SSGTSFLPLE RDVEIVFSGF K GSHICMFS NLRQMNLSPI LCPYYDLITN FKTTTEIRAY VDAHEELKSL LTYLCLCTIV GLCDTFTETR NMDTGEYVWK VR DVVSRNH TPAQNVEKFC YTIQNAKYMI QLVHVLLFPL TDNKYADLPN YVAVITQGAI NQSRSHNVIN TTDESNSNTT SDT AASTSG IVSGDTGTVA SLYPDEFKYV QS UniProtKB: Clamp protein |
-分子 #2: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Fako virus (ウイルス) / 株: CSW77 Fako virus (ウイルス) / 株: CSW77 |
| 分子量 | 理論値: 136.689766 KDa |
| 配列 | 文字列: MRPIRMYKNN QERTNLKHQE INEEQQNEQT TSNQGFTRSD NSGKINIERI SSSRNQITDG KTVSSYSKIE TNRSSQDSVQ HGGSSITYT SDTTGNPRIT NARTNNDETH ATGPIEDLNS TSHGREPEIE SFADRAELAM MIQGMTVGAL TVQPMRSIRS T FANLANVL ...文字列: MRPIRMYKNN QERTNLKHQE INEEQQNEQT TSNQGFTRSD NSGKINIERI SSSRNQITDG KTVSSYSKIE TNRSSQDSVQ HGGSSITYT SDTTGNPRIT NARTNNDETH ATGPIEDLNS TSHGREPEIE SFADRAELAM MIQGMTVGAL TVQPMRSIRS T FANLANVL IFHDVFTTED KPSAFIEYHS DEMIVNMPKQ TYNPIDNLAK ILYLPSLEKF KYGTGIVQLN YSPHISKLYQ NT NNIINTI TDGITYANRT EFFIRVMVLM MMDRKILTME FYDVDTSAIS NTAILPTIPT TTGVSPLLRI DTRTEPIWYN DAI KTLITN LTIQYGKIKT VLDANAVKRY SVVGYPIDQY RAYLYNHNLL EYLGKKVKRE DIMSLIKALS YEFDLITISD LEYQ NIPKW FSDNDLSRFI FSICMFPDIV RQFHALNIDY FSQANVFTVK SENAIVKMLN SNQNMEPTII NWFLFRICAI DKTVI DDYF SLEMTPIIMR PKLYDFDMKR GEPVSLLYIL ELILFSIMFP NVTQHMLGQI QARILYISMY AFRQEYLKFI TKFGFY YKI VNGRKEYIQV TNQNERMTEN NDVLTGNLYP SLFTDDPTLS AIAPTLAKIA RLMKPTTSLT PDDRAIAAKF PRFKDSA HL NPYSSLNIGG RTQHSVTYTR MYDAIEEMFN LILRAFASSF AQRPRAGVTQ LKSLLTQLAD PLCLALDGHV YHLYNVMA N MMQNFIPNTD GQFHSFRACS YAVKDGGNIY RVVQNGDELN ESLLIDTAIV WGLLGNTDSS YGNAIGATGT ANVPTKVQP VIPTPDNFIT PTIHLKTSID AICSVEGILL LILSRQTTIP GYEDELNKLR TGISQPKVTE RQYRRARESI KNMLGSGDYN VAPLHFLLH TEHRSTKLSK PLIRRVLDNV VQPYVANLDP AEFENTPQLI ENSNMTRLQI ALKMLTGDMD DIVKGLILHK R ACAKFDVY ETLTIPTDVK TIVLTMQHIS TQTQNNMVYY VFLIDGVKIL AEDIKNVNFQ IDITGIWPEY VITLLLRAIN NG FNTYVSM PNILYKPTIT ADVRQFMNTT KAETLLISNK SIVHEIMFFD NALQPKMSSD TLALSEAVYR TIWNSSIITQ RIS ARGLMN LEDARPPEAK ISHQSELDMG KIDETSGEPI YTSGLQKMQS SKVSMANVVL SAGSDVIRQA AIKYNVVRTQ EIIL FE UniProtKB: Major capsid protein |
-分子 #3: Turret protein
| 分子 | 名称: Turret protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Fako virus (ウイルス) / 株: CSW77 Fako virus (ウイルス) / 株: CSW77 |
| 分子量 | 理論値: 121.23257 KDa |
| 配列 | 文字列: MIDLRLEEDI LTATLPEFLS TRPKYRYAYT NTKQQDIRFQ GPMRHVRLTH LYKQTKLWNL QYIERELAIS EIDDALDEFI QTFSLPYVI EQGTYKYNML LGMHAHNVNY QDDVSELIAN NPQLLNYLDD NPFSAIFELV NVDLQIYQYG QNIFNNEAEH T ILFLKDNT ...文字列: MIDLRLEEDI LTATLPEFLS TRPKYRYAYT NTKQQDIRFQ GPMRHVRLTH LYKQTKLWNL QYIERELAIS EIDDALDEFI QTFSLPYVI EQGTYKYNML LGMHAHNVNY QDDVSELIAN NPQLLNYLDD NPFSAIFELV NVDLQIYQYG QNIFNNEAEH T ILFLKDNT NYGVIQALQK HPFSATHINW HLHKHIFVFH SREQLLNKLL SAGLEDSQLY QRQKTYSTKR GDRPTERMVT YI EDDHIRR IQAVFPLLLD NIFDVKLHKD SSMTWLKSYA DMIYDSVKNS NSTITPEIRK LYLRMYNQYM RIFLPIEQYM LYD NTCWPF SEKITLKINV RLISSRENQP VLWKTPIDTE NLISIVQPDE PINKLNFTAI PSTMIRLNDN ITMYRAVKDM FSAI EYLPD AIENIPTLTM KEQALSRYIS PDSEAQNFFN NQPPYLNSIM NVNRQVFEAV KRGNIQVSTG SMEHLCLCMH VKSGL IVGR TVLIDDKVVL RRNFNASTAK MITCYVKAFA QLYGEGSLIN PGLRMVFFGV ETEPAIDILK LFYGDKSLYI QGFGDR GIG RDKFRTKIED ALTLRIGCDI LISDIDQADY EDPNEEKFDD ITDFVCYVTE LVISNATVGL VKISMPTYYI MNKISST LN NKFSNVAINI VKLSTQKPYT YEAYIMLSHG STLTNKGYLR NPVCDVYLEK ISLQPMDLKI ISTISNEINY DKPTLYRF V VDKNDVTDVS IAMHILSIHC STITTRSVMV RSDNTGAFVT MSGIKDMKRV AIMNRMTDGT SANSYMHEQN GKLYLQKVP YLEDLISAFP NGFGSTYQND YDSSMSVINV NALIRQVVYR VISKSIPVAL LESLSRIRII GGRDLGEMNA VYKLYKTPIE VYDAVGITR EYPHVQISYR AQRYSFTESI PNHTLLLANY VIMNDVDGAP ISSLEQINTI KKIISKISLG SIAYIQVYTD I VARNINVM TKNDSFLISA NADKTVFKVQ VSGYKAVEMC NYEQLLQLVS DNTGVNIIKL TYQDVLESCV LSSGILGDTG SW LLDLVLA STYIIEIRG UniProtKB: Turret protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | 詳細: unspecified | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 4.7 mm Bright-field microscopy / Cs: 4.7 mm |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER / ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) 検出モード: INTEGRATING / 実像数: 2400 / 平均電子線量: 30.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X