+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7515 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram averages of budding-arrested nucleocapsid-like particles (NCLPs) inside CHIKV-infected human cells. | |||||||||
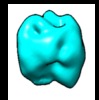
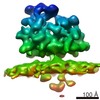
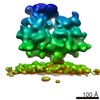
 マップデータ マップデータ | Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside CHIKV-infected human cells. | |||||||||
 試料 試料 | Budding-arrestedChikungunya virus nucleocapsid-like particle != Chikungunya virus Budding-arrestedChikungunya virus nucleocapsid-like particle
| |||||||||
| 生物種 |    Chikungunya virus (チクングニヤウイルス) Chikungunya virus (チクングニヤウイルス) | |||||||||
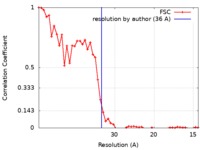
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 36.0 Å クライオ電子顕微鏡法 / 解像度: 36.0 Å | |||||||||
 データ登録者 データ登録者 | Galaz-Montoya JG / Jin J / Sherman MB | |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2018 ジャーナル: Cell Host Microbe / 年: 2018タイトル: Neutralizing Antibodies Inhibit Chikungunya Virus Budding at the Plasma Membrane. 著者: Jing Jin / Jesús G Galaz-Montoya / Michael B Sherman / Stella Y Sun / Cynthia S Goldsmith / Eileen T O'Toole / Larry Ackerman / Lars-Anders Carlson / Scott C Weaver / Wah Chiu / Graham Simmons /   要旨: Neutralizing antibodies (NAbs) are traditionally thought to inhibit virus infection by preventing virion entry into target cells. In addition, antibodies can engage Fc receptors (FcRs) on immune ...Neutralizing antibodies (NAbs) are traditionally thought to inhibit virus infection by preventing virion entry into target cells. In addition, antibodies can engage Fc receptors (FcRs) on immune cells to activate antiviral responses. We describe a mechanism by which NAbs inhibit chikungunya virus (CHIKV), the most common alphavirus infecting humans, by preventing virus budding from infected human cells and activating IgG-specific Fcγ receptors. NAbs bind to CHIKV glycoproteins on the infected cell surface and induce glycoprotein coalescence, preventing budding of nascent virions and leaving structurally heterogeneous nucleocapsids arrested in the cytosol. Furthermore, NAbs induce clustering of CHIKV replication spherules at sites of budding blockage. Functionally, these densely packed glycoprotein-NAb complexes on infected cells activate Fcγ receptors, inducing a strong, antibody-dependent, cell-mediated cytotoxicity response from immune effector cells. Our findings describe a triply functional antiviral pathway for NAbs that might be broadly applicable across virus-host systems, suggesting avenues for therapeutic innovation through antibody design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7515.map.gz emd_7515.map.gz | 599.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7515-v30.xml emd-7515-v30.xml emd-7515.xml emd-7515.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7515_fsc.xml emd_7515_fsc.xml | 4.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7515_1.png emd_7515_1.png emd_7515_2.png emd_7515_2.png emd_7515_3.png emd_7515_3.png emd_7515_4.png emd_7515_4.png emd_7515_5.png emd_7515_5.png | 57.6 KB 58.4 KB 65.2 KB 67.4 KB 65.8 KB | ||
| その他 |  emd_7515_additional_1.map.gz emd_7515_additional_1.map.gz emd_7515_additional_2.map.gz emd_7515_additional_2.map.gz emd_7515_additional_3.map.gz emd_7515_additional_3.map.gz emd_7515_additional_4.map.gz emd_7515_additional_4.map.gz | 615.5 KB 629.1 KB 664 KB 681.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7515 http://ftp.pdbj.org/pub/emdb/structures/EMD-7515 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7515 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7515 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7515.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7515.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside CHIKV-infected human cells. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.16 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside...
| ファイル | emd_7515_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside CHIKV-infected human cells. | ||||||||||||
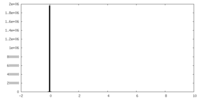
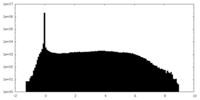
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside...
| ファイル | emd_7515_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside CHIKV-infected human cells. | ||||||||||||
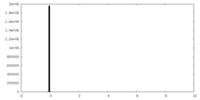
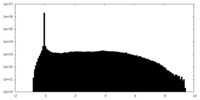
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside...
| ファイル | emd_7515_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside CHIKV-infected human cells. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside...
| ファイル | emd_7515_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of budding-arrested nucleocapsid-like particles (NCLPs) inside CHIKV-infected human cells. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Budding-arrestedChikungunya virus nucleocapsid-like particle
| 全体 | 名称: Budding-arrestedChikungunya virus nucleocapsid-like particle |
|---|---|
| 要素 |
|
-超分子 #1: Chikungunya virus
| 超分子 | 名称: Chikungunya virus / タイプ: virus / ID: 1 / 親要素: 0 / NCBI-ID: 37124 / 生物種: Chikungunya virus / Sci species strain: 181/clone 25 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| ウイルス殻 | Shell ID: 1 / 直径: 698.0 Å / T番号(三角分割数): 4 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Neutralizing antibody treated U2OS cells that were infected with CHIKV vaccine strain 181/clone 25 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) 平均露光時間: 0.6 sec. / 平均電子線量: 1.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X