+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7437 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
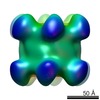
| タイトル | Single-Particle reconstruction of DARP14 - A designed protein scaffold displaying ~17kDa DARPin proteins | |||||||||
 マップデータ マップデータ | Single-Particle reconstruction of DARP14 - A designed protein scaffold displaying ~17kDa DARPin proteins | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Designed Complex (デザイン) / Designed Complex (デザイン) /  DARPin / DARPin /  Scaffold (足場) / Single-Particle Cryo-EM / Scaffold (足場) / Single-Particle Cryo-EM /  DE NOVO PROTEIN (De novo) DE NOVO PROTEIN (De novo) | |||||||||
| 機能・相同性 | 5-carboxymethyl-2-hydroxymuconate isomerase / 5-carboxymethyl-2-hydroxymuconate isomerase /  5-carboxymethyl-2-hydroxymuconate delta-isomerase activity / Tautomerase/MIF superfamily / aromatic compound catabolic process / 5-carboxymethyl-2-hydroxymuconate isomerase 5-carboxymethyl-2-hydroxymuconate delta-isomerase activity / Tautomerase/MIF superfamily / aromatic compound catabolic process / 5-carboxymethyl-2-hydroxymuconate isomerase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 | synthetic construct (人工物) /   Pseudomonas aeruginosa PAO1 (緑膿菌) / synthetic (人工物) / Pseudomonas aeruginosa PAO1 (緑膿菌) / synthetic (人工物) /   Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.49 Å クライオ電子顕微鏡法 / 解像度: 3.49 Å | |||||||||
 データ登録者 データ登録者 | Gonen S / Liu Y / Yeates TO / Gonen T | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Near-atomic cryo-EM imaging of a small protein displayed on a designed scaffolding system. 著者: Yuxi Liu / Shane Gonen / Tamir Gonen / Todd O Yeates /  要旨: Current single-particle cryo-electron microscopy (cryo-EM) techniques can produce images of large protein assemblies and macromolecular complexes at atomic level detail without the need for crystal ...Current single-particle cryo-electron microscopy (cryo-EM) techniques can produce images of large protein assemblies and macromolecular complexes at atomic level detail without the need for crystal growth. However, proteins of smaller size, typical of those found throughout the cell, are not presently amenable to detailed structural elucidation by cryo-EM. Here we use protein design to create a modular, symmetrical scaffolding system to make protein molecules of typical size suitable for cryo-EM. Using a rigid continuous alpha helical linker, we connect a small 17-kDa protein (DARPin) to a protein subunit that was designed to self-assemble into a cage with cubic symmetry. We show that the resulting construct is amenable to structural analysis by single-particle cryo-EM, allowing us to identify and solve the structure of the attached small protein at near-atomic detail, ranging from 3.5- to 5-Å resolution. The result demonstrates that proteins considerably smaller than the theoretical limit of 50 kDa for cryo-EM can be visualized clearly when arrayed in a rigid fashion on a symmetric designed protein scaffold. Furthermore, because the amino acid sequence of a DARPin can be chosen to confer tight binding to various other protein or nucleic acid molecules, the system provides a future route for imaging diverse macromolecules, potentially broadening the application of cryo-EM to proteins of typical size in the cell. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7437.map.gz emd_7437.map.gz | 32.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7437-v30.xml emd-7437-v30.xml emd-7437.xml emd-7437.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7437.png emd_7437.png | 163.9 KB | ||
| Filedesc metadata |  emd-7437.cif.gz emd-7437.cif.gz | 5.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7437 http://ftp.pdbj.org/pub/emdb/structures/EMD-7437 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7437 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7437 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7437.map.gz / 形式: CCP4 / 大きさ: 190.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7437.map.gz / 形式: CCP4 / 大きさ: 190.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single-Particle reconstruction of DARP14 - A designed protein scaffold displaying ~17kDa DARPin proteins | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.655 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : DARP14
| 全体 | 名称: DARP14 |
|---|---|
| 要素 |
|
-超分子 #1: DARP14
| 超分子 | 名称: DARP14 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #2: DARP14 - Subunit A with DARPin
| 超分子 | 名称: DARP14 - Subunit A with DARPin / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #3: DARP14 - Subunit B
| 超分子 | 名称: DARP14 - Subunit B / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
-分子 #1: DARP14 - Subunit A with DARPin
| 分子 | 名称: DARP14 - Subunit A with DARPin / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic (人工物) |
| 分子量 | 理論値: 35.018965 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MRITTKVGDK GSTRLFGGEE VWKDSPIIEA NGTLDELTSF IGEAKHYVDE EMKGILEEIQ NDIYKIMGEI GSKGKIEGIS EERIAWLLK LILRYMEMVN LKSFVLPGGT LESAKLDVCR TIARRALRKV LTVTREFGIG AEAAAYLLAL SDLLFLLARV I EIELGKKL ...文字列: MRITTKVGDK GSTRLFGGEE VWKDSPIIEA NGTLDELTSF IGEAKHYVDE EMKGILEEIQ NDIYKIMGEI GSKGKIEGIS EERIAWLLK LILRYMEMVN LKSFVLPGGT LESAKLDVCR TIARRALRKV LTVTREFGIG AEAAAYLLAL SDLLFLLARV I EIELGKKL LEAARAGQDD EVRILMANGA DVNAHDDQGS TPLHLAAWIG HPEIVEVLLK HGADVNARDT DGWTPLHLAA DN GHLEIVE VLLKYGADVN AQDAYGLTPL HLAADRGHLE IVEVLLKHGA DVNAQDKFGK TAFDISIDNG NEDLAEILQK LN |
-分子 #2: DARP14 - Subunit B
| 分子 | 名称: DARP14 - Subunit B / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 14.346274 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MPHLVIEATA NLRLETSPGE LLEQANKALF ASGQFGEADI KSRFVTLEAY RQGTAAVERA YLHACLSILD GRDIATRTLL GASLCAVLA EAVAGGGEEG VQVSVEVREM ERLSYAKRVV ARQRLEHHHH HH UniProtKB: 5-carboxymethyl-2-hydroxymuconate isomerase |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 想定した対称性 - 点群: T (正4面体型対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.49 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 183753 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.49 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 183753 |
 ムービー
ムービー コントローラー
コントローラー