+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7128 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the mechanically activated ion channel Piezo1 | |||||||||||||||
 マップデータ マップデータ | C3 symmetry refinement mouse Piezo1 core. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mechanosensitive monoatomic cation channel activity / positive regulation of cell-cell adhesion mediated by integrin / detection of mechanical stimulus / mechanosensitive monoatomic ion channel activity / positive regulation of integrin activation / positive regulation of myotube differentiation / lamellipodium membrane / monoatomic cation transport / monoatomic cation channel activity /  regulation of membrane potential ...mechanosensitive monoatomic cation channel activity / positive regulation of cell-cell adhesion mediated by integrin / detection of mechanical stimulus / mechanosensitive monoatomic ion channel activity / positive regulation of integrin activation / positive regulation of myotube differentiation / lamellipodium membrane / monoatomic cation transport / monoatomic cation channel activity / regulation of membrane potential ...mechanosensitive monoatomic cation channel activity / positive regulation of cell-cell adhesion mediated by integrin / detection of mechanical stimulus / mechanosensitive monoatomic ion channel activity / positive regulation of integrin activation / positive regulation of myotube differentiation / lamellipodium membrane / monoatomic cation transport / monoatomic cation channel activity /  regulation of membrane potential / endoplasmic reticulum-Golgi intermediate compartment membrane / cellular response to mechanical stimulus / endoplasmic reticulum membrane / regulation of membrane potential / endoplasmic reticulum-Golgi intermediate compartment membrane / cellular response to mechanical stimulus / endoplasmic reticulum membrane /  小胞体 / identical protein binding / 小胞体 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||||||||
 データ登録者 データ登録者 | Saotome K / Kefauver JM / Patapoutian A / Ward AB | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Structure of the mechanically activated ion channel Piezo1. 著者: Kei Saotome / Swetha E Murthy / Jennifer M Kefauver / Tess Whitwam / Ardem Patapoutian / Andrew B Ward /  要旨: Piezo1 and Piezo2 are mechanically activated ion channels that mediate touch perception, proprioception and vascular development. Piezo proteins are distinct from other ion channels and their ...Piezo1 and Piezo2 are mechanically activated ion channels that mediate touch perception, proprioception and vascular development. Piezo proteins are distinct from other ion channels and their structure remains poorly defined, which impedes detailed study of their gating and ion permeation properties. Here we report a high-resolution cryo-electron microscopy structure of the mouse Piezo1 trimer. The detergent-solubilized complex adopts a three-bladed propeller shape with a curved transmembrane region containing at least 26 transmembrane helices per protomer. The flexible propeller blades can adopt distinct conformations, and consist of a series of four-transmembrane helical bundles that we term Piezo repeats. Carboxy-terminal domains line the central ion pore, and the channel is closed by constrictions in the cytosol. A kinked helical beam and anchor domain link the Piezo repeats to the pore, and are poised to control gating allosterically. The structure provides a foundation to dissect further how Piezo channels are regulated by mechanical force. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
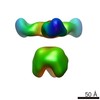
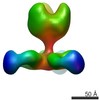
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7128.map.gz emd_7128.map.gz | 226.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7128-v30.xml emd-7128-v30.xml emd-7128.xml emd-7128.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7128.png emd_7128.png | 36.3 KB | ||
| その他 |  emd_7128_additional.map.gz emd_7128_additional.map.gz | 206.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7128 http://ftp.pdbj.org/pub/emdb/structures/EMD-7128 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7128 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7128 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7128.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7128.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C3 symmetry refinement mouse Piezo1 core. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Refinement of symmetry-expanded mouse Piezo1 blade class 1.
| ファイル | emd_7128_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refinement of symmetry-expanded mouse Piezo1 blade class 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
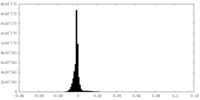
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : mouse Piezo1
| 全体 | 名称: mouse Piezo1 |
|---|---|
| 要素 |
|
-超分子 #1: mouse Piezo1
| 超分子 | 名称: mouse Piezo1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
| 分子量 | 理論値: 876 KDa |
-分子 #1: Piezo-type mechanosensitive ion channel component 1,Piezo-type me...
| 分子 | 名称: Piezo-type mechanosensitive ion channel component 1,Piezo-type mechanosensitive ion channel component 1,mouse Piezo1,Piezo-type mechanosensitive ion channel component 1,Piezo-type ...名称: Piezo-type mechanosensitive ion channel component 1,Piezo-type mechanosensitive ion channel component 1,mouse Piezo1,Piezo-type mechanosensitive ion channel component 1,Piezo-type mechanosensitive ion channel component 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 161.973531 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: PNPIPNFIHC RSYLDMLKVA VFRYLFWLVL VVVFVAGATR ISIFGLGYLL ACFYLLLFGT TLLQKDTRAQ LVLWDCLILY NVTVIISKN MLSLLSCVFV EQMQSNFCWV IQLFSLVCTV KGYYDPKEMM TRDRDCLLPV EEAGIIWDSI CFFFLLLQRR I FLSHYFLH ...文字列: PNPIPNFIHC RSYLDMLKVA VFRYLFWLVL VVVFVAGATR ISIFGLGYLL ACFYLLLFGT TLLQKDTRAQ LVLWDCLILY NVTVIISKN MLSLLSCVFV EQMQSNFCWV IQLFSLVCTV KGYYDPKEMM TRDRDCLLPV EEAGIIWDSI CFFFLLLQRR I FLSHYFLH VSADLKATAL QASRGFALYN AANLKSINFH RQIEEKSLAQ LKRQMKRIRA KQEKYRQSQA SRGQLQSKDP QD PSQEPGP DSPGGSSPPR RQWWRPWLDH ATVIHSGDYF LFESDSEEEE EALPEDPRPA AQSAFQMAYQ AWVTNAQTVL RQR RERARQ ERAEQLASGG DLNPDVEPVD VPEDEMAGRS HMMQRVLSTM QFLWVLGQAT VDGLTRWLRA FTKHHRTMSD VLCA ERYLL TQELLRVGEV RRGVLDQLYV GEDEATLSGP VETRDGPSTA SSGLGAEEPL SSMTDD(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)P ELEEAERFEA QQGRTLRLLR AGYQCVAAHS ELLCYFIII LNHMVTASAA SLVLPVLVFL WAMLTIPRPS KRFWMTAIVF TEVMVVTKYL FQFGFFPWNS YVVLRRYENK PYFPPRILGL EKTDSYIKY DLVQLMALFF HRSQLLCYGL WDHEEDRYPK DHCRSSVKDR EAKEEPEAKL ESQSETGTGH PKEPVLAGTP R DHIQGKGS IRSKDVIQDP PEDLKPRHTR HISIRFRRRK ETPGPKGTAV METEHEEGEG KETTERKRPR HTQEKSKFRE RM KAAGRRL QSFCVSLAQS FYQPLQRFFH DILHTKYRAA TDVYALMFLA DIVDIIIIIF GFWAFGKHSA ATDIASSLSD DQV PQAFLF MLLVQFGTMV IDRALYLRKT VLGKLAFQVV LVVAIHIWMF FILPAVTERM FSQNAVAQLW YFVKCIYFAL SAYQ IRCGY PTRILGNFLT KKYNHLNLFL FQGFRLVPFL VELRAVMDWV WTDTTLSLSN WMCVEDIYAN IFIIKCSRET EKKYP QPKG QKKKKIVKYG MGGLIILFLI AIIWFPLLFM SLIRSVVGVV NQPIDVTVTL KLGGYEPLFT MSAQQPSIVP FTPQAY EEL SQQFDPYPLA MQFISQYSPE DIVTAQIEGS SGALWRISPP SRAQMKQELY NGTADITLRF TWNFQRDLAK GGTVEYT NE KHTLELAPNS TARRQLAQLL EGRPDQSVVI PHLFPKYIRA PNGPEANPVK QLQPDEEEDY LGVRIQLRRE QVGTGASG E QAGTKASDFL EWWVIELQDC KADCNLLPMV IFSDKVSPPS LGFLAGYGIV GLYVSIVLVV GKFVRGFFSE ISHSIMFEE LPCVDRILKL CQDIFLVRET RELELEEELY AKLIFLYRSP ETMIKWTRER EKKLGAPLEV LFQ |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil, UltrAuFoil / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | mouse Piezo1 was purified in glyco-diosgenin. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: Gctf (ver. 0.50) |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION (ver. 2.0) |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION (ver. 2.0) |
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 72627 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 72627 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6bpz: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X