+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7033 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Emptied phiX174 complexed with lipopolysaccharides after DNA ejection | |||||||||
 マップデータ マップデータ | Emptied phiX174 complexed with lipopolysaccharides after DNA ejection, low pass-filtered to 8 Angstrom resolution | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Enterobacteria phage phiX174 isolate Sanger (ファージ) / Enterobacteria phage phiX174 isolate Sanger (ファージ) /   Salmonella enterica (サルモネラ菌) Salmonella enterica (サルモネラ菌) | |||||||||
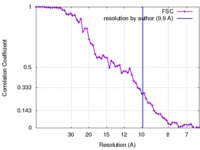
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 9.9 Å クライオ電子顕微鏡法 / 解像度: 9.9 Å | |||||||||
 データ登録者 データ登録者 | Rossmann MG / Sun Y / Klose T / Roznowski A / Fane BA / Pollack L / Tokuda J / Mauney A | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2014 ジャーナル: Nature / 年: 2014タイトル: Icosahedral bacteriophage ΦX174 forms a tail for DNA transport during infection. 著者: Lei Sun / Lindsey N Young / Xinzheng Zhang / Sergei P Boudko / Andrei Fokine / Erica Zbornik / Aaron P Roznowski / Ian J Molineux / Michael G Rossmann / Bentley A Fane /  要旨: Prokaryotic viruses have evolved various mechanisms to transport their genomes across bacterial cell walls. Many bacteriophages use a tail to perform this function, whereas tail-less phages rely on ...Prokaryotic viruses have evolved various mechanisms to transport their genomes across bacterial cell walls. Many bacteriophages use a tail to perform this function, whereas tail-less phages rely on host organelles. However, the tail-less, icosahedral, single-stranded DNA ΦX174-like coliphages do not fall into these well-defined infection processes. For these phages, DNA delivery requires a DNA pilot protein. Here we show that the ΦX174 pilot protein H oligomerizes to form a tube whose function is most probably to deliver the DNA genome across the host's periplasmic space to the cytoplasm. The 2.4 Å resolution crystal structure of the in vitro assembled H protein's central domain consists of a 170 Å-long α-helical barrel. The tube is constructed of ten α-helices with their amino termini arrayed in a right-handed super-helical coiled-coil and their carboxy termini arrayed in a left-handed super-helical coiled-coil. Genetic and biochemical studies demonstrate that the tube is essential for infectivity but does not affect in vivo virus assembly. Cryo-electron tomograms show that tubes span the periplasmic space and are present while the genome is being delivered into the host cell's cytoplasm. Both ends of the H protein contain transmembrane domains, which anchor the assembled tubes into the inner and outer cell membranes. The central channel of the H-protein tube is lined with amide and guanidinium side chains. This may be a general property of viral DNA conduits and is likely to be critical for efficient genome translocation into the host. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7033.map.gz emd_7033.map.gz | 13.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7033-v30.xml emd-7033-v30.xml emd-7033.xml emd-7033.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7033_fsc.xml emd_7033_fsc.xml | 6.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7033.png emd_7033.png | 177 KB | ||
| マスクデータ |  emd_7033_msk_1.map emd_7033_msk_1.map emd_7033_msk_2.map emd_7033_msk_2.map | 15.6 MB 15.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_7033_half_map_1.map.gz emd_7033_half_map_1.map.gz emd_7033_half_map_2.map.gz emd_7033_half_map_2.map.gz | 11.9 MB 11.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7033 http://ftp.pdbj.org/pub/emdb/structures/EMD-7033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7033 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7033.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7033.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Emptied phiX174 complexed with lipopolysaccharides after DNA ejection, low pass-filtered to 8 Angstrom resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
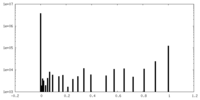
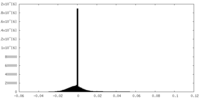
| ファイル |  emd_7033_msk_1.map emd_7033_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #2
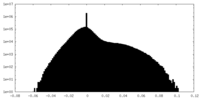
| ファイル |  emd_7033_msk_2.map emd_7033_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Emptied phiX174 complexed with lipopolysaccharides after DNA ejection,...
| ファイル | emd_7033_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Emptied phiX174 complexed with lipopolysaccharides after DNA ejection, half map #1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Emptied phiX174 complexed with lipopolysaccharides after DNA ejection,...
| ファイル | emd_7033_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Emptied phiX174 complexed with lipopolysaccharides after DNA ejection, half map #2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Full phiX174 complexed with lipopolysaccharides before DNA ejection
| 全体 | 名称: Full phiX174 complexed with lipopolysaccharides before DNA ejection |
|---|---|
| 要素 |
|
-超分子 #1: Full phiX174 complexed with lipopolysaccharides before DNA ejection
| 超分子 | 名称: Full phiX174 complexed with lipopolysaccharides before DNA ejection タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage phiX174 isolate Sanger (ファージ) Enterobacteria phage phiX174 isolate Sanger (ファージ) |
| 分子量 | 理論値: 10 KDa |
-超分子 #2: Lipopolysaccharides (rough strains) from Salmonella enterica sero...
| 超分子 | 名称: Lipopolysaccharides (rough strains) from Salmonella enterica serotype typhimurium TV119 (Ra mutant) タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Salmonella enterica (サルモネラ菌) / 株: typhimurium TV119 / 細胞中の位置: outer membrane Salmonella enterica (サルモネラ菌) / 株: typhimurium TV119 / 細胞中の位置: outer membrane |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 |
| 詳細 | Mixed with LPS and incubated at 33 degrees C for 1 minute |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 30.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X