+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6979 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E. coli 50S subunit bound HflX protein in presence of ATP (AMP-PNP) and GTP (GMP-PNP) analogs. | |||||||||
 マップデータ マップデータ | E. coli 50S bound HflX protein in presence of ATP (AMP-PNP) and GTP (GMP-PNP) analogs. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ATPase (ATPアーゼ) / ATPase (ATPアーゼ) /  RNA helicase (ヘリカーゼ) / Heat stress / RNA helicase (ヘリカーゼ) / Heat stress /  RIBOSOME (リボソーム) RIBOSOME (リボソーム) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribosome disassembly /  guanosine tetraphosphate binding / guanosine tetraphosphate binding /  ribosomal large subunit binding / rescue of stalled ribosome / ribosomal large subunit binding / rescue of stalled ribosome /  ribosome binding / response to heat / ribosome binding / response to heat /  rRNA binding / rRNA binding /  GTPase activity / GTP binding / GTPase activity / GTP binding /  ATP hydrolysis activity ...ribosome disassembly / ATP hydrolysis activity ...ribosome disassembly /  guanosine tetraphosphate binding / guanosine tetraphosphate binding /  ribosomal large subunit binding / rescue of stalled ribosome / ribosomal large subunit binding / rescue of stalled ribosome /  ribosome binding / response to heat / ribosome binding / response to heat /  rRNA binding / rRNA binding /  GTPase activity / GTP binding / GTPase activity / GTP binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /   Escherichia coli (strain K12) (大腸菌) Escherichia coli (strain K12) (大腸菌) | |||||||||
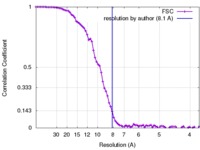
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.1 Å クライオ電子顕微鏡法 / 解像度: 8.1 Å | |||||||||
 データ登録者 データ登録者 | Dey S | |||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2018 ジャーナル: J Cell Biol / 年: 2018タイトル: The universally conserved GTPase HflX is an RNA helicase that restores heat-damaged ribosomes. 著者: Sandip Dey / Chiranjit Biswas / Jayati Sengupta /  要旨: The ribosome-associated GTPase HflX acts as an antiassociation factor upon binding to the 50S ribosomal subunit during heat stress in Although HflX is recognized as a guanosine triphosphatase, ...The ribosome-associated GTPase HflX acts as an antiassociation factor upon binding to the 50S ribosomal subunit during heat stress in Although HflX is recognized as a guanosine triphosphatase, several studies have shown that the N-terminal domain 1 of HflX is capable of hydrolyzing adenosine triphosphate (ATP), but the functional role of its adenosine triphosphatase (ATPase) activity remains unknown. We demonstrate that HflX possesses ATP-dependent RNA helicase activity and is capable of unwinding large subunit ribosomal RNA. A cryo-electron microscopy structure of the 50S-HflX complex in the presence of nonhydrolyzable analogues of ATP and guanosine triphosphate hints at a mode of action for the RNA helicase and suggests the linker helical domain may have a determinant role in RNA unwinding. Heat stress results in inactivation of the ribosome, and we show that HflX can restore heat-damaged ribosomes and improve cell survival. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6979.map.gz emd_6979.map.gz | 33.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6979-v30.xml emd-6979-v30.xml emd-6979.xml emd-6979.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6979_fsc.xml emd_6979_fsc.xml | 8.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6979.png emd_6979.png | 278.3 KB | ||
| Filedesc metadata |  emd-6979.cif.gz emd-6979.cif.gz | 6.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6979 http://ftp.pdbj.org/pub/emdb/structures/EMD-6979 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6979 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6979 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6979.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6979.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli 50S bound HflX protein in presence of ATP (AMP-PNP) and GTP (GMP-PNP) analogs. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.89 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E. coli 50S bound HflX protein in presence of ATP (AMP-PNP) and G...
| 全体 | 名称: E. coli 50S bound HflX protein in presence of ATP (AMP-PNP) and GTP (GMP-PNP) analogs. |
|---|---|
| 要素 |
|
-超分子 #1: E. coli 50S bound HflX protein in presence of ATP (AMP-PNP) and G...
| 超分子 | 名称: E. coli 50S bound HflX protein in presence of ATP (AMP-PNP) and GTP (GMP-PNP) analogs. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: BL21 Escherichia coli (大腸菌) / 株: BL21 |
-分子 #1: GTPase HflX
| 分子 | 名称: GTPase HflX / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (strain K12) (大腸菌) / 株: K12 Escherichia coli (strain K12) (大腸菌) / 株: K12 |
| 分子量 | 理論値: 48.392988 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MFDRYDAGEQ AVLVHIYFTQ DKDMEDLQEF ESLVSSAGVE ALQVITGSRK APHPKYFVGE GKAVEIAEAV KATGASVVLF DHALSPAQE RNLERLCECR VIDRTGLILD IFAQRARTHE GKLQVELAQL RHLATRLVRG WTHLERQKGG IGLRGPGETQ L ETDRRLLR ...文字列: MFDRYDAGEQ AVLVHIYFTQ DKDMEDLQEF ESLVSSAGVE ALQVITGSRK APHPKYFVGE GKAVEIAEAV KATGASVVLF DHALSPAQE RNLERLCECR VIDRTGLILD IFAQRARTHE GKLQVELAQL RHLATRLVRG WTHLERQKGG IGLRGPGETQ L ETDRRLLR NRIVQIQSRL ERVEKQREQG RQSRIKADVP TVSLVGYTNA GKSTLFNRIT EARVYAADQL FATLDPTLRR ID VADVGET VLADTVGFIR HLPHDLVAAF KATLQETRQA TLLLHVIDAA DVRVQENIEA VNTVLEEIDA HEIPTLLVMN KID MLEDFE PRIDRDEENK PNRVWLSAQT GAGIPQLFQA LTERLSGEVA QHTLRLPPQE GRLRSRFYQL QAIEKEWMEE DGSV SLQVR MPIVDWRRLC KQEPALIDYL I UniProtKB: GTPase HflX |
-分子 #2: 5S rRNA
| 分子 | 名称: 5S rRNA / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: MRE 600 Escherichia coli (大腸菌) / 株: MRE 600 |
| 分子量 | 理論値: 38.79009 KDa |
| 配列 | 文字列: UGCCUGGCGG CCGUAGCGCG GUGGUCCCAC CUGACCCCAU GCCGAACUCA GAAGUGAAAC GCCGUAGCGC CGAUGGUAGU GUGGGGUCU CCCCAUGCGA GAGUAGGGAA CUGCCAGGCA U GENBANK: GENBANK: CP027586.1 |
-分子 #3: 23S rRNA
| 分子 | 名称: 23S rRNA / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: MRE 600 Escherichia coli (大腸菌) / 株: MRE 600 |
| 分子量 | 理論値: 941.306188 KDa |
| 配列 | 文字列: GGUUAAGCGA CUAAGCGUAC ACGGUGGAUG CCCUGGCAGU CAGAGGCGAU GAAGGACGUG CUAAUCUGCG AUAAGCGUCG GUAAGGUGA UAUGAACCGU UAUAACCGGC GAUUUCCGAA UGGGGAAACC CAGUGUGUUU CGACACACUA UCAUUAACUG A AUCCAUAG ...文字列: GGUUAAGCGA CUAAGCGUAC ACGGUGGAUG CCCUGGCAGU CAGAGGCGAU GAAGGACGUG CUAAUCUGCG AUAAGCGUCG GUAAGGUGA UAUGAACCGU UAUAACCGGC GAUUUCCGAA UGGGGAAACC CAGUGUGUUU CGACACACUA UCAUUAACUG A AUCCAUAG GUUAAUGAGG CGAACCGGGG GAACUGAAAC AUCUAAGUAC CCCGAGGAAA AGAAAUCAAC CGAGAUUCCC CC AGUAGCG GCGAGCGAAC GGGGAGCAGC CCAGAGCCUG AAUCAGUGUG UGUGUUAGUG GAAGCGUCUG GAAAGGCGCG CGA UACAGG GUGACAGCCC CGUACACAAA AAUGCACAUG CUGUGAGCUC GAUGAGUAGG GCGGGACACG UGGUAUCCUG UCUG AAUAU GGGGGGACCA UCCUCCAAGG CUAAAUACUC CUGACUGACC GAUAGUGAAC CAGUACCGUG AGGGAAAGGC GAAAA GAAC CCCGGCGAGG GGAGUGAAAA AGAACCUGAA ACCGUGUACG UACAAGCAGU GGGAGCACGC UUAGGCGUGU GACUGC GUA CCUUUUGUAU AAUGGGUCAG CGACUUAUAU UCUGUAGCAA GGUUAACCGA AUAGGGGAGC CGAAGGGAAA CCGAGUC UU AACUGGGCGU UAAGUUGCAG GGUAUAGACC CGAAACCCGG UGAUCUAGCC AUGGGCAGGU UGAAGGUUGG GUAACACU A ACUGGAGGAC CGAACCGACU AAUGUUGAAA AAUUAGCGGA UGACUUGUGG CUGGGGGUGA AAGGCCAAUC AAACCGGGA GAUAGCUGGU UCUCCCCGAA AGCUAUUUAG GUAGCGCCUC GUGAAUUCAU CUCCGGGGGU AGAGCACUGU UUCGGCAAGG GGGUCAUCC CGACUUACCA ACCCGAUGCA AACUGCGAAU ACCGGAGAAU GUUAUCACGG GAGACACACG GCGGGUGCUA A CGUCCGUC GUGAAGAGGG AAACAACCCA GACCGCCAGC UAAGGUCCCA AAGUCAUGGU UAAGUGGGAA ACGAUGUGGG AA GGCCCAG ACAGCCAGGA UGUUGGCUUA GAAGCAGCCA UCAUUUAAAG AAAGCGUAAU AGCUCACUGG UCGAGUCGGC CUG CGCGGA AGAUGUAACG GGGCUAAACC AUGCACCGAA GCUGCGGCAG CGACGCUUAU GCGUUGUUGG GUAGGGGAGC GUUC UGUAA GCCUGCGAAG GUGUGCUGUG AGGCAUGCUG GAGGUAUCAG AAGUGCGAAU GCUGACAUAA GUAACGAUAA AGCGG GUGA AAAGCCCGCU CGCCGGAAGA CCAAGGGUUC CUGUCCAACG UUAAUCGGGG CAGGGUGAGU CGACCCCUAA GGCGAG GCC GAAAGGCGUA GUCGAUGGGA AACAGGUUAA UAUUCCUGUA CUUGGUGUUA CUGCGAAGGG GGGACGGAGA AGGCUAU GU UGGCCGGGCG ACGGUUGUCC CGGUUUAAGC GUGUAGGCUG GUUUUCCAGG CAAAUCCGGA AAAUCAAGGC UGAGGCGU G AUGACGAGGC ACUACGGUGC UGAAGCAACA AAUGCCCUGC UUCCAGGAAA AGCCUCUAAG CAUCAGGUAA CAUCAAAUC GUACCCCAAA CCGACACAGG UGGUCAGGUA GAGAAUACCA AGGCGCUUGA GAGAACUCGG GUGAAGGAAC UAGGCAAAAU GGUGCCGUA ACUUCGGGAG AAGGCACGCU GAUAUGUAGG UGAGGUCCCU CGCGGAUGGA GCUGAAAUCA GUCGAAGAUA C CAGCUGGC UGCAACUGUU UAUUAAAAAC ACAGCACUGU GCAAACACGA AAGUGGACGU AUACGGUGUG ACGCCUGCCC GG UGCCGGA AGGUUAAUUG AUGGGGUUAG CGCAAGCGAA GCUCUUGAUC GAAGCCCCGG UAAACGGCGG CCGUAACUAU AAC GGUCCU AAGGUAGCGA AAUUCCUUGU CGGGUAAGUU CCGACCUGCA CGAAUGGCGU AAUGAUGGCC AGGCUGUCUC CACC CGAGA CUCAGUGAAA UUGAACUCGC UGUGAAGAUG CAGUGUACCC GCGGCAAGAC GGAAAGACCC CGUGAACCUU UACUA UAGC UUGACACUGA ACAUUGAGCC UUGAUGUGUA GGAUAGGUGG GAGGCUUUGA AGUGUGGACG CCAGUCUGCA UGGAGC CGA CCUUGAAAUA CCACCCUUUA AUGUUUGAUG UUCUAACGUU GACCCGUAAU CCGGGUUGCG GACAGUGUCU GGUGGGU AG UUUGACUGGG GCGGUCUCCU CCUAAAGAGU AACGGAGGAG CACGAAGGUU GGCUAAUCCU GGUCGGACAU CAGGAGGU U AGUGCAAUGG CAUAAGCCAG CUUGACUGCG AGCGUGACGG CGCGAGCAGG UGCGAAAGCA GGUCAUAGUG AUCCGGUGG UUCUGAAUGG AAGGGCCAUC GCUCAACGGA UAAAAGGUAC UCCGGGGAUA ACAGGCUGAU ACCGCCCAAG AGUUCAUAUC GACGGCGGU GUUUGGCACC UCGAUGUCGG CUCAUCACAU CCUGGGGCUG AAGUAGGUCC CAAGGGUAUG GCUGUUCGCC A UUUAAAGU GGUACGCGAG CUGGGUUUAG AACGUCGUGA GACAGUUCGG UCCCUAUCUG CCGUGGGCGC UGGAGAACUG AG GGGGGCU GCUCCUAGUA CGAGAGGACC GGAGUGGACG CAUCACUGGU GUUCGGGUUG UCAUGCCAAU GGCACUGCCC GGU AGCUAA AUGCGGAAGA GAUAAGUGCU GAAAGCAUCU AAGCACGAAA CUUGCCCCGA GAUGAGUUCU CCCUGACCCU UUAA GGGUC CUGAAGGAAC GUUGAAGACG ACGACGUUGA UAGGCCGGGU GUGUAAGCGC AGCGAUGCGU UGAGCUAACC GGUAC UAAU GAACCGUGAG GCUUAACCU GENBANK: GENBANK: CP016018.1 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION 回折 回折 |
| 試料ステージ | 試料ホルダーモデル: GATAN 910 MULTI-SPECIMEN SINGLE TILT CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 撮影したグリッド数: 4 / 平均露光時間: 2.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-5zzm: |
 ムービー
ムービー コントローラー
コントローラー