+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6729 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Anti-CRISPR proteins AcrF1/2 bound to Csy surveillance complex with a 32nt spacer crRNA backbone region | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  anti-CRISPR protein / Csy complex / Type I-F CRISPR/Cas system / anti-CRISPR protein / Csy complex / Type I-F CRISPR/Cas system /  IMMUNE SYSTEM-RNA complex (免疫系) IMMUNE SYSTEM-RNA complex (免疫系) | |||||||||
| 機能・相同性 |  : / : /  Anti-CRISPR protein Acr30-35/AcrF1 / CRISPR-associated protein Csy3 / CRISPR-associated protein (Cas_Csy3) / defense response to virus / Uncharacterized protein / CRISPR-associated protein Csy3 Anti-CRISPR protein Acr30-35/AcrF1 / CRISPR-associated protein Csy3 / CRISPR-associated protein (Cas_Csy3) / defense response to virus / Uncharacterized protein / CRISPR-associated protein Csy3 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) / Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) /  Pseudomonas phage JBD30 (ファージ) / Pseudomonas phage JBD30 (ファージ) /   Pseudomonas aeruginosa (緑膿菌) Pseudomonas aeruginosa (緑膿菌) | |||||||||
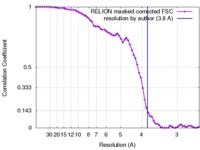
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Peng R / Shi Y / Gao GF | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2017 ジャーナル: Cell Res / 年: 2017タイトル: Alternate binding modes of anti-CRISPR viral suppressors AcrF1/2 to Csy surveillance complex revealed by cryo-EM structures. 著者: Ruchao Peng / Ying Xu / Tengfei Zhu / Ningning Li / Jianxun Qi / Yan Chai / Min Wu / Xinzheng Zhang / Yi Shi / Peiyi Wang / Jiawei Wang / Ning Gao / George Fu Gao /    要旨: Bacteriophages encode anti-CRISPR suppressors to counteract the CRISPR/Cas immunity of their bacterial hosts, thus facilitating their survival and replication. Previous studies have shown that two ...Bacteriophages encode anti-CRISPR suppressors to counteract the CRISPR/Cas immunity of their bacterial hosts, thus facilitating their survival and replication. Previous studies have shown that two phage-encoded anti-CRISPR proteins, AcrF1 and AcrF2, suppress the type I-F CRISPR/Cas system of Pseudomonas aeruginosa by preventing target DNA recognition by the Csy surveillance complex, but the precise underlying mechanism was unknown. Here we present the structure of AcrF1/2 bound to the Csy complex determined by cryo-EM single-particle reconstruction. By structural analysis, we found that AcrF1 inhibits target DNA recognition of the Csy complex by interfering with base pairing between the DNA target strand and crRNA spacer. In addition, multiple copies of AcrF1 bind to the Csy complex with different modes when working individually or cooperating with AcrF2, which might exclude target DNA binding through different mechanisms. Together with previous reports, we provide a comprehensive working scenario for the two anti-CRISPR suppressors, AcrF1 and AcrF2, which silence CRISPR/Cas immunity by targeting the Csy surveillance complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6729.map.gz emd_6729.map.gz | 2.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6729-v30.xml emd-6729-v30.xml emd-6729.xml emd-6729.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6729_fsc.xml emd_6729_fsc.xml | 6.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6729.png emd_6729.png | 180.8 KB | ||
| Filedesc metadata |  emd-6729.cif.gz emd-6729.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6729 http://ftp.pdbj.org/pub/emdb/structures/EMD-6729 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6729 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6729 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6729.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6729.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : anti-CRISPR proteins AcrF1/2 bound to Csy surveillance complex wi...
| 全体 | 名称: anti-CRISPR proteins AcrF1/2 bound to Csy surveillance complex with a 32nt spacer crRNA backbone region |
|---|---|
| 要素 |
|
-超分子 #1: anti-CRISPR proteins AcrF1/2 bound to Csy surveillance complex wi...
| 超分子 | 名称: anti-CRISPR proteins AcrF1/2 bound to Csy surveillance complex with a 32nt spacer crRNA backbone region タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 400 KDa |
-超分子 #2: CRISPR-associated protein Csy3
| 超分子 | 名称: CRISPR-associated protein Csy3 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|
-超分子 #3: crRNA with 20nt spacer sequence
| 超分子 | 名称: crRNA with 20nt spacer sequence / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-超分子 #4: AcrF1
| 超分子 | 名称: AcrF1 / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|
-分子 #1: CRISPR-associated protein Csy3
| 分子 | 名称: CRISPR-associated protein Csy3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 37.579273 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MSKPILSTAS VLAFERKLDP SDALMSAGAW AQRDASQEWP AVTVREKSVR GTISNRLKTK DRDPAKLDAS IQSPNLQTVD VANLPSDAD TLKVRFTLRV LGGAGTPSAC NDAAYRDKLL QTVATYVNDQ GFAELARRYA HNLANARFLW RNRVGAEAVE V RINHIRQG ...文字列: MSKPILSTAS VLAFERKLDP SDALMSAGAW AQRDASQEWP AVTVREKSVR GTISNRLKTK DRDPAKLDAS IQSPNLQTVD VANLPSDAD TLKVRFTLRV LGGAGTPSAC NDAAYRDKLL QTVATYVNDQ GFAELARRYA HNLANARFLW RNRVGAEAVE V RINHIRQG EVARAWRFDA LAIGLRDFKA DAELDALAEL IASGLSGSGH VLLEVVAFAR IGDGQEVFPS QELILDKGDK KG QKSKTLY SVRDAAAIHS QKIGNALRTI DTWYPDEDGL GPIAVEPYGS VTSQGKAYRQ PKQKLDFYTL LDNWVLRDEA PAV EQQHYV IANLIRGGVF GEAEEK UniProtKB: CRISPR-associated protein Csy3 |
-分子 #3: Uncharacterized protein AcrF1
| 分子 | 名称: Uncharacterized protein AcrF1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) |
| 分子量 | 理論値: 8.824931 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MKFIKYLSTA HLNYMNIAVY ENGSKIKARV ENVVNGKSVG ARDFDSTEQL ESWFYGLPGS GLGRIENAMN EISRRENP UniProtKB: Uncharacterized protein |
-分子 #2: crRNA with 32nt spacer sequence
| 分子 | 名称: crRNA with 32nt spacer sequence / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas aeruginosa (緑膿菌) Pseudomonas aeruginosa (緑膿菌) |
| 分子量 | 理論値: 19.265404 KDa |
| 配列 | 文字列: CUAAGAAAUU CACGGCGGGC UUGAUGUCCG CGUCUACCUG GUUCACUGCC GUGUAGGCAG GENBANK: GENBANK: HQ326201.1 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 0.2 sec. / 平均電子線量: 1.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-5xlo: |
 ムービー
ムービー コントローラー
コントローラー