+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6152 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structures of Protective Antibodies Reveal Sites of Vulnerability on Ebola Virus | |||||||||
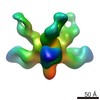
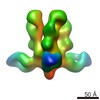
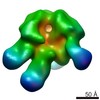
 マップデータ マップデータ | Reconstruction of Ebola virus glycoprotein bound to Fab fragments of c13C6 and c4G7 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Ebola (エボラ出血熱) / Antibody cocktails / mAbs / Ebola (エボラ出血熱) / Antibody cocktails / mAbs /  ZMAb (ZMapp) / ZMAb (ZMapp) /  ZMapp (ZMapp) / ZMapp (ZMapp) /  MB-003 (ZMapp) MB-003 (ZMapp) | |||||||||
| 生物種 |   Ebola virus sp. (エボラウイルス) / Ebola virus sp. (エボラウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 24.0 Å ネガティブ染色法 / 解像度: 24.0 Å | |||||||||
 データ登録者 データ登録者 | Murin CD / Fusco ML / Bornholdt ZA / Qiu X / Olinger GG / Zeitlin L / Kobinger GP / Ward AB / Saphire EO | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2014 ジャーナル: Proc Natl Acad Sci U S A / 年: 2014タイトル: Structures of protective antibodies reveal sites of vulnerability on Ebola virus. 著者: Charles D Murin / Marnie L Fusco / Zachary A Bornholdt / Xiangguo Qiu / Gene G Olinger / Larry Zeitlin / Gary P Kobinger / Andrew B Ward / Erica Ollmann Saphire /   要旨: Ebola virus (EBOV) and related filoviruses cause severe hemorrhagic fever, with up to 90% lethality, and no treatments are approved for human use. Multiple recent outbreaks of EBOV and the likelihood ...Ebola virus (EBOV) and related filoviruses cause severe hemorrhagic fever, with up to 90% lethality, and no treatments are approved for human use. Multiple recent outbreaks of EBOV and the likelihood of future human exposure highlight the need for pre- and postexposure treatments. Monoclonal antibody (mAb) cocktails are particularly attractive candidates due to their proven postexposure efficacy in nonhuman primate models of EBOV infection. Two candidate cocktails, MB-003 and ZMAb, have been extensively evaluated in both in vitro and in vivo studies. Recently, these two therapeutics have been combined into a new cocktail named ZMapp, which showed increased efficacy and has been given compassionately to some human patients. Epitope information and mechanism of action are currently unknown for most of the component mAbs. Here we provide single-particle EM reconstructions of every mAb in the ZMapp cocktail, as well as additional antibodies from MB-003 and ZMAb. Our results illuminate key and recurring sites of vulnerability on the EBOV glycoprotein and provide a structural rationale for the efficacy of ZMapp. Interestingly, two of its components recognize overlapping epitopes and compete with each other for binding. Going forward, this work now provides a basis for strategic selection of next-generation antibody cocktails against Ebola and related viruses and a model for predicting the impact of ZMapp on potential escape mutations in ongoing or future Ebola outbreaks. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6152.map.gz emd_6152.map.gz | 1.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6152-v30.xml emd-6152-v30.xml emd-6152.xml emd-6152.xml | 11.1 KB 11.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6152.jpg emd_6152.jpg | 143.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6152 http://ftp.pdbj.org/pub/emdb/structures/EMD-6152 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6152 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6152 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6152.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6152.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of Ebola virus glycoprotein bound to Fab fragments of c13C6 and c4G7 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Fab fragment of c13C6 and c4G7 chimerized human monoclonal IgG1 a...
| 全体 | 名称: Fab fragment of c13C6 and c4G7 chimerized human monoclonal IgG1 antibody bound to Ebola virus glycoprotein |
|---|---|
| 要素 |
|
-超分子 #1000: Fab fragment of c13C6 and c4G7 chimerized human monoclonal IgG1 a...
| 超分子 | 名称: Fab fragment of c13C6 and c4G7 chimerized human monoclonal IgG1 antibody bound to Ebola virus glycoprotein タイプ: sample / ID: 1000 集合状態: Six Fab fragments bound to a single, trimeric GP (two Fabs per protomer) Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 750 KDa |
-分子 #1: Ebola virus glycoprotein
| 分子 | 名称: Ebola virus glycoprotein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: EBOV GP / コピー数: 1 / 集合状態: trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Ebola virus sp. (エボラウイルス) Ebola virus sp. (エボラウイルス) |
| 分子量 | 理論値: 450 KDa |
| 組換発現 | 生物種:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ)組換細胞: Schneider 2 (S2) / 組換プラスミド: pMT-puro |
-分子 #2: chimerized human IgG1 antigen binding fragment c13C6
| 分子 | 名称: chimerized human IgG1 antigen binding fragment c13C6 タイプ: protein_or_peptide / ID: 2 / Name.synonym: c13C6 Fab / コピー数: 3 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human Homo sapiens (ヒト) / 別称: human |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
-分子 #3: chimerized human IgG1 antigen binding fragment c4G7
| 分子 | 名称: chimerized human IgG1 antigen binding fragment c4G7 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: c4G7 Fab / コピー数: 3 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human Homo sapiens (ヒト) / 別称: human |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 20 mM Tris, 150 mM NaCl |
| 染色 | タイプ: NEGATIVE 詳細: To prepare negative stain grids, a 4 uL aliquot of each complex, which had been diluted to a concentration of ~0.03 ug/mL with TBS buffer, was placed for 15 seconds onto carbon-coated 400 Cu ...詳細: To prepare negative stain grids, a 4 uL aliquot of each complex, which had been diluted to a concentration of ~0.03 ug/mL with TBS buffer, was placed for 15 seconds onto carbon-coated 400 Cu mesh grids that had been plasma cleaned for 20 s (Gatan), blotted off on the edge of the grid, then immediately stained for 30 s with 4 uL of 2% uranyl formate. The stain was blotted off on the edge of the grid and the grid was allowed to dry. |
| グリッド | 詳細: 400 Cu mesh |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 Bright-field microscopy / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
| 日付 | 2014年2月5日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: OTHER / 実像数: 65 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 24.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2, XMipp, IMAGIC / 使用した粒子像数: 10210 |
|---|
 ムービー
ムービー コントローラー
コントローラー