+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5217 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Visualizing the structural changes of bacteriophage epsilon15 and its Salmonella host during infection | |||||||||
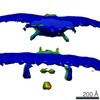
 マップデータ マップデータ | Empty capsid attached to cell | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  virus (ウイルス) / virus (ウイルス) /  infection (感染) / infection (感染) /  Salmonella (サルモネラ) / Salmonella (サルモネラ) /  tomography (トモグラフィー) / tomography (トモグラフィー) /  bacteriophage (ファージ) bacteriophage (ファージ) | |||||||||
| 生物種 |   epsilon15 (ファージ) epsilon15 (ファージ) | |||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Chang JT / Schmid MF / Haase-Pettingell C / Weigele PR / King JA / Chiu W | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2010 ジャーナル: J Mol Biol / 年: 2010タイトル: Visualizing the structural changes of bacteriophage Epsilon15 and its Salmonella host during infection. 著者: Juan T Chang / Michael F Schmid / Cameron Haase-Pettingell / Peter R Weigele / Jonathan A King / Wah Chiu /  要旨: The efficient mechanism by which double-stranded DNA bacteriophages deliver their chromosome across the outer membrane, cell wall, and inner membrane of Gram-negative bacteria remains obscure. ...The efficient mechanism by which double-stranded DNA bacteriophages deliver their chromosome across the outer membrane, cell wall, and inner membrane of Gram-negative bacteria remains obscure. Advances in single-particle electron cryomicroscopy have recently revealed details of the organization of the DNA injection apparatus within the mature virion for various bacteriophages, including epsilon15 (ɛ15) and P-SSP7. We have used electron cryotomography and three-dimensional subvolume averaging to capture snapshots of ɛ15 infecting its host Salmonella anatum. These structures suggest the following stages of infection. In the first stage, the tailspikes of ɛ15 attach to the surface of the host cell. Next, ɛ15's tail hub attaches to a putative cell receptor and establishes a tunnel through which the injection core proteins behind the portal exit the virion. A tube spanning the periplasmic space is formed for viral DNA passage, presumably from the rearrangement of core proteins or from cellular components. This tube would direct the DNA into the cytoplasm and protect it from periplasmic nucleases. Once the DNA has been injected into the cell, the tube and portal seals, and the empty bacteriophage remains at the cell surface. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5217.map.gz emd_5217.map.gz | 25 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5217-v30.xml emd-5217-v30.xml emd-5217.xml emd-5217.xml | 8.9 KB 8.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5217_1.png emd_5217_1.png | 102.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5217 http://ftp.pdbj.org/pub/emdb/structures/EMD-5217 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5217 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5217 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5217.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5217.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Empty capsid attached to cell | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 10.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bacteriophage epsilon15, Salmonella anatum
| 全体 | 名称: Bacteriophage epsilon15, Salmonella anatum |
|---|---|
| 要素 |
|
-超分子 #1000: Bacteriophage epsilon15, Salmonella anatum
| 超分子 | 名称: Bacteriophage epsilon15, Salmonella anatum / タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|
-超分子 #1: epsilon15
| 超分子 | 名称: epsilon15 / タイプ: virus / ID: 1 / Name.synonym: epsilon15 / 生物種: epsilon15 / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: epsilon15 |
|---|---|
| 宿主 | 生物種:   Salmonella enterica subsp. enterica serovar Anatum (サルモネラ菌) Salmonella enterica subsp. enterica serovar Anatum (サルモネラ菌)別称: BACTERIA(EUBACTERIA) |
| ウイルス殻 | Shell ID: 1 / 直径: 700 Å / T番号(三角分割数): 7 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| グリッド | 詳細: 200 mesh copper grid |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 93 K / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 9.0 µm / 最小 デフォーカス(公称値): 6.0 µm / 倍率(公称値): 20000 Bright-field microscopy / 最大 デフォーカス(公称値): 9.0 µm / 最小 デフォーカス(公称値): 6.0 µm / 倍率(公称値): 20000 |
| 特殊光学系 | エネルギーフィルター - 名称: JEM3200FSC エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER |
| 温度 | 最低: 93 K / 最高: 93 K / 平均: 93 K |
| 日付 | 2006年9月16日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC CCD / 平均電子線量: 65 e/Å2 |
- 画像解析
画像解析
| 最終 再構成 | 詳細: The 3D reconstructions were performed with IMOD using gold as fiducial markers. Subvolumes, each containing a single particle, were computationally extracted from the reconstructions, aligned ...詳細: The 3D reconstructions were performed with IMOD using gold as fiducial markers. Subvolumes, each containing a single particle, were computationally extracted from the reconstructions, aligned to a reference model, and averaged together. |
|---|
 ムービー
ムービー コントローラー
コントローラー