+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4612 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of calcium-free mTMEM16F lipid scramblase in digitonin | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報calcium activated phospholipid scrambling / calcium activated phosphatidylserine scrambling / calcium activated phosphatidylcholine scrambling / calcium activated galactosylceramide scrambling / phosphatidylserine exposure on blood platelet / positive regulation of potassium ion export across plasma membrane / purinergic nucleotide receptor signaling pathway / positive regulation of monoatomic ion transmembrane transport /  phospholipid scramblase activity / activation of blood coagulation via clotting cascade ...calcium activated phospholipid scrambling / calcium activated phosphatidylserine scrambling / calcium activated phosphatidylcholine scrambling / calcium activated galactosylceramide scrambling / phosphatidylserine exposure on blood platelet / positive regulation of potassium ion export across plasma membrane / purinergic nucleotide receptor signaling pathway / positive regulation of monoatomic ion transmembrane transport / phospholipid scramblase activity / activation of blood coagulation via clotting cascade ...calcium activated phospholipid scrambling / calcium activated phosphatidylserine scrambling / calcium activated phosphatidylcholine scrambling / calcium activated galactosylceramide scrambling / phosphatidylserine exposure on blood platelet / positive regulation of potassium ion export across plasma membrane / purinergic nucleotide receptor signaling pathway / positive regulation of monoatomic ion transmembrane transport /  phospholipid scramblase activity / activation of blood coagulation via clotting cascade / negative regulation of cell volume / intracellularly calcium-gated chloride channel activity / bone mineralization involved in bone maturation / pore complex assembly / cholinergic synapse / plasma membrane phospholipid scrambling / voltage-gated monoatomic ion channel activity / bleb assembly / positive regulation of phagocytosis, engulfment / Stimuli-sensing channels / voltage-gated chloride channel activity / calcium-activated cation channel activity / positive regulation of endothelial cell apoptotic process / positive regulation of monocyte chemotaxis / dendritic cell chemotaxis / chloride transport / phospholipid translocation / phospholipid scramblase activity / activation of blood coagulation via clotting cascade / negative regulation of cell volume / intracellularly calcium-gated chloride channel activity / bone mineralization involved in bone maturation / pore complex assembly / cholinergic synapse / plasma membrane phospholipid scrambling / voltage-gated monoatomic ion channel activity / bleb assembly / positive regulation of phagocytosis, engulfment / Stimuli-sensing channels / voltage-gated chloride channel activity / calcium-activated cation channel activity / positive regulation of endothelial cell apoptotic process / positive regulation of monocyte chemotaxis / dendritic cell chemotaxis / chloride transport / phospholipid translocation /  chloride channel activity / chloride channel activity /  chloride channel complex / monoatomic cation transport / sodium ion transmembrane transport / regulation of postsynaptic membrane potential / positive regulation of bone mineralization / chloride transmembrane transport / Neutrophil degranulation / chloride channel complex / monoatomic cation transport / sodium ion transmembrane transport / regulation of postsynaptic membrane potential / positive regulation of bone mineralization / chloride transmembrane transport / Neutrophil degranulation /  synaptic membrane / establishment of localization in cell / calcium ion transmembrane transport / synaptic membrane / establishment of localization in cell / calcium ion transmembrane transport /  凝固・線溶系 / positive regulation of apoptotic process / protein homodimerization activity / identical protein binding / 凝固・線溶系 / positive regulation of apoptotic process / protein homodimerization activity / identical protein binding /  metal ion binding / metal ion binding /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Alvadia C / Lim NK / Clerico Mosina V / Oostergetel GT / Dutzler R / Paulino C | |||||||||
| 資金援助 |  スイス, スイス,  オランダ, 2件 オランダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: Cryo-EM structures and functional characterization of the murine lipid scramblase TMEM16F. 著者: Carolina Alvadia / Novandy K Lim / Vanessa Clerico Mosina / Gert T Oostergetel / Raimund Dutzler / Cristina Paulino /   要旨: The lipid scramblase TMEM16F initiates blood coagulation by catalyzing the exposure of phosphatidylserine in platelets. The protein is part of a family of membrane proteins, which encompasses calcium- ...The lipid scramblase TMEM16F initiates blood coagulation by catalyzing the exposure of phosphatidylserine in platelets. The protein is part of a family of membrane proteins, which encompasses calcium-activated channels for ions and lipids. Here, we reveal features of murine TMEM16F (mTMEM16F) that underlie its function as a lipid scramblase and an ion channel. The cryo-EM data of mTMEM16F in absence and presence of Ca define the ligand-free closed conformation of the protein and the structure of a Ca-bound intermediate. Both conformations resemble their counterparts of the scrambling-incompetent anion channel mTMEM16A, yet with distinct differences in the region of ion and lipid permeation. In conjunction with functional data, we demonstrate the relationship between ion conduction and lipid scrambling. Although activated by a common mechanism, both functions appear to be mediated by alternate protein conformations that are at equilibrium in the ligand-bound state. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4612.map.gz emd_4612.map.gz | 38.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4612-v30.xml emd-4612-v30.xml emd-4612.xml emd-4612.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4612_fsc.xml emd_4612_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4612.png emd_4612.png | 201.8 KB | ||
| マスクデータ |  emd_4612_msk_1.map emd_4612_msk_1.map | 40.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_4612_half_map_1.map.gz emd_4612_half_map_1.map.gz emd_4612_half_map_2.map.gz emd_4612_half_map_2.map.gz | 30.3 MB 30.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4612 http://ftp.pdbj.org/pub/emdb/structures/EMD-4612 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4612 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4612 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4612.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4612.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.012 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
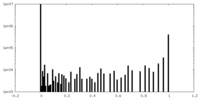
| ファイル |  emd_4612_msk_1.map emd_4612_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 used during refinement and for...
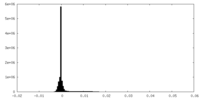
| ファイル | emd_4612_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 used during refinement and for FSC gold-standard resolution calculation mTMEM16F_dig_noCa. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 used during refinement and for...
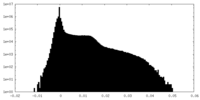
| ファイル | emd_4612_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 used during refinement and for FSC gold-standard resolution calculation mTMEM16F_dig_noCa. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : mTMEM16F
| 全体 | 名称: mTMEM16F |
|---|---|
| 要素 |
|
-超分子 #1: mTMEM16F
| 超分子 | 名称: mTMEM16F / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293T cells Homo sapiens (ヒト) / 組換細胞: HEK293T cells |
| 分子量 | 理論値: 212 KDa |
-分子 #1: Anoctamin-6
| 分子 | 名称: Anoctamin-6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 106.367727 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MQMMTRKVLL NMELEEDDDE DGDIVLENFD QTIVCPTFGS LENQQDFRTP EFEEFNGKPD SLFFTDGQRR IDFILVYEDE SKKENNKKG TNEKQKRKRQ AYESNLICHG LQLEATRSVS DDKLVFVKVH APWEVLCTYA EIMHIKLPLK PNDLKTRSPF G NLNWFTKV ...文字列: MQMMTRKVLL NMELEEDDDE DGDIVLENFD QTIVCPTFGS LENQQDFRTP EFEEFNGKPD SLFFTDGQRR IDFILVYEDE SKKENNKKG TNEKQKRKRQ AYESNLICHG LQLEATRSVS DDKLVFVKVH APWEVLCTYA EIMHIKLPLK PNDLKTRSPF G NLNWFTKV LRVNESVIKP EQEFFTAPFE KSRMNDFYIL DRDSFFNPAT RSRIVYFILS RVKYQVMNNV NKFGINRLVS SG IYKAAFP LHDCRFNYES EDISCPSERY LLYREWAHPR SIYKKQPLDL IRKYYGEKIG IYFAWLGYYT QMLLLAAVVG VAC FLYGYL DQDNCTWSKE VCDPDIGGQI LMCPQCDRLC PFWRLNITCE SSKKLCIFDS FGTLIFAVFM GVWVTLFLEF WKRR QAELE YEWDTVELQQ EEQARPEYEA QCNHVVINEI TQEEERIPFT TCGKCIRVTL CASAVFFWIL LIIASVIGII VYRLS VFIV FSTTLPKNPN GTDPIQKYLT PQMATSITAS IISFIIIMIL NTIYEKVAIM ITNFELPRTQ TDYENSLTMK MFLFQF VNY YSSCFYIAFF KGKFVGYPGD PVYLLGKYRS EECDPGGCLL ELTTQLTIIM GGKAIWNNIQ EVLLPWVMNL IGRYKRV SG SEKITPRWEQ DYHLQPMGKL GLFYEYLEMI IQFGFVTLFV ASFPLAPLLA LVNNILEIRV DAWKLTTQFR RMVPEKAQ D IGAWQPIMQG IAILAVVTNA MIIAFTSDMI PRLVYYWSFS IPPYGDHTYY TMDGYINNTL SVFNITDFKN TDKENPYIG LGNYTLCRYR DFRNPPGHPQ EYKHNIYYWH VIAAKLAFII VMEHIIYSVK FFISYAIPDV SKITKSKIKR EKYLTQKLLH ESHLKDLTK NMGIIAERIG GTVDNSVRPK LE |
-分子 #2: 1,2-DIDECANOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIDECANOYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: P1O |
|---|---|
| 分子量 | 理論値: 566.728 Da |
| Chemical component information |  ChemComp-P1O: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 0.1% digitonin, 150 mM NaCl, 20 mM HEPES, pH 7.5 and 2 mM EGTA |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 詳細: at 5 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 288 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 0.3 µm / 倍率(補正後): 49407 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 49407 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 49407 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 90.0 K / 最高: 105.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-60 / 撮影したグリッド数: 7 / 実像数: 5657 / 平均露光時間: 9.0 sec. / 平均電子線量: 52.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-6qpb: |
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X