+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4470 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Spiral structure of E. coli RavA in the RavA-LdcI cage-like complex | |||||||||||||||
 マップデータ マップデータ | Cryo-EM map of Class 2 of the E. coli RavA-LdcI cage-like complex after symmetry expansion and masked 3D classification. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 リシンデカルボキシラーゼ / リシンデカルボキシラーゼ /  lysine decarboxylase activity / lysine decarboxylase activity /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する / amino acid metabolic process / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する / amino acid metabolic process /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||||||||
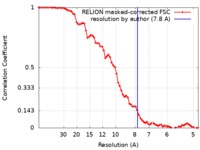
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.8 Å クライオ電子顕微鏡法 / 解像度: 7.8 Å | |||||||||||||||
 データ登録者 データ登録者 | Arragain B / Felix J / Malet H / Gutsche I / Jessop M | |||||||||||||||
| 資金援助 |  フランス, 4件 フランス, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2020 ジャーナル: Commun Biol / 年: 2020タイトル: Structural insights into ATP hydrolysis by the MoxR ATPase RavA and the LdcI-RavA cage-like complex. 著者: Matthew Jessop / Benoit Arragain / Roger Miras / Angélique Fraudeau / Karine Huard / Maria Bacia-Verloop / Patrice Catty / Jan Felix / Hélène Malet / Irina Gutsche /  要旨: The hexameric MoxR AAA+ ATPase RavA and the decameric lysine decarboxylase LdcI form a 3.3 MDa cage, proposed to assist assembly of specific respiratory complexes in E. coli. Here, we show that ...The hexameric MoxR AAA+ ATPase RavA and the decameric lysine decarboxylase LdcI form a 3.3 MDa cage, proposed to assist assembly of specific respiratory complexes in E. coli. Here, we show that inside the LdcI-RavA cage, RavA hexamers adopt an asymmetric spiral conformation in which the nucleotide-free seam is constrained to two opposite orientations. Cryo-EM reconstructions of free RavA reveal two co-existing structural states: an asymmetric spiral, and a flat C2-symmetric closed ring characterised by two nucleotide-free seams. The closed ring RavA state bears close structural similarity to the pseudo two-fold symmetric crystal structure of the AAA+ unfoldase ClpX, suggesting a common ATPase mechanism. Based on these structures, and in light of the current knowledge regarding AAA+ ATPases, we propose different scenarios for the ATP hydrolysis cycle of free RavA and the LdcI-RavA cage-like complex, and extend the comparison to other AAA+ ATPases of clade 7. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4470.map.gz emd_4470.map.gz | 2.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4470-v30.xml emd-4470-v30.xml emd-4470.xml emd-4470.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4470_fsc.xml emd_4470_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4470.png emd_4470.png | 261.5 KB | ||
| マスクデータ |  emd_4470_msk_1.map emd_4470_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_4470_half_map_1.map.gz emd_4470_half_map_1.map.gz emd_4470_half_map_2.map.gz emd_4470_half_map_2.map.gz | 23.5 MB 23.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4470 http://ftp.pdbj.org/pub/emdb/structures/EMD-4470 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4470 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4470 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4470.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4470.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of Class 2 of the E. coli RavA-LdcI cage-like complex after symmetry expansion and masked 3D classification. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 2.42 Å / Y: 2.42 Å / Z: 2.42001 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4470_msk_1.map emd_4470_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half-map 1.
| ファイル | emd_4470_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half-map 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half-map 1.
| ファイル | emd_4470_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half-map 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of the hexameric MoxR AAA+ ATPase RavA and the decameric ...
| 全体 | 名称: Complex of the hexameric MoxR AAA+ ATPase RavA and the decameric lysine decarboxylase LdcI. |
|---|---|
| 要素 |
|
-超分子 #1: Complex of the hexameric MoxR AAA+ ATPase RavA and the decameric ...
| 超分子 | 名称: Complex of the hexameric MoxR AAA+ ATPase RavA and the decameric lysine decarboxylase LdcI. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: MG1655 delta_relA delta_spoT Escherichia coli (大腸菌) / 組換株: MG1655 delta_relA delta_spoT |
| 分子量 | 理論値: 3.3 MDa |
-分子 #1: Inducible lysine decarboxylase
| 分子 | 名称: Inducible lysine decarboxylase / タイプ: protein_or_peptide / ID: 1 / コピー数: 20 / 光学異性体: LEVO / EC番号:  リシンデカルボキシラーゼ リシンデカルボキシラーゼ |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 81.357008 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MNVIAILNHM GVYFKEEPIR ELHRALERLN FQIVYPNDRD DLLKLIENNA RLCGVIFDWD KYNLELCEEI SKMNENLPLY AFANTYSTL DVSLNDLRLQ ISFFEYALGA AEDIANKIKQ TTDEYINTIL PPLTKALFKY VREGKYTFCT PGHMGGTAFQ K SPVGSLFY ...文字列: MNVIAILNHM GVYFKEEPIR ELHRALERLN FQIVYPNDRD DLLKLIENNA RLCGVIFDWD KYNLELCEEI SKMNENLPLY AFANTYSTL DVSLNDLRLQ ISFFEYALGA AEDIANKIKQ TTDEYINTIL PPLTKALFKY VREGKYTFCT PGHMGGTAFQ K SPVGSLFY DFFGPNTMKS DISISVSELG SLLDHSGPHK EAEQYIARVF NADRSYMVTN GTSTANKIVG MYSAPAGSTI LI DRNCHKS LTHLMMMSDV TPIYFRPTRN AYGILGGIPQ SEFQHATIAK RVKETPNATW PVHAVITNST YDGLLYNTDF IKK TLDVKS IHFDSAWVPY TNFSPIYEGK CGMSGGRVEG KVIYETQSTH KLLAAFSQAS MIHVKGDVNE ETFNEAYMMH TTTS PHYGI VASTETAAAM MKGNAGKRLI NGSIERAIKF RKEIKRLRTE SDGWFFDVWQ PDHIDTTECW PLRSDSTWHG FKNID NEHM YLDPIKVTLL TPGMEKDGTM SDFGIPASIV AKYLDEHGIV VEKTGPYNLL FLFSIGIDKT KALSLLRALT DFKRAF DLN LRVKNMLPSL YREDPEFYEN MRIQELAQNI HKLIVHHNLP DLMYRAFEVL PTMVMTPYAA FQKELHGMTE EVYLDEM VG RINANMILPY PPGVPLVMPG EMITEESRPV LEFLQMLCEI GAHYPGFETD IHGAYRQADG RYTVKVLKEE SKK |
-分子 #2: ATPase RavA
| 分子 | 名称: ATPase RavA / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO EC番号:  加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する 加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 56.351445 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAHPHLLAER ISRLSSSLEK GLYERSHAIR LCLLAALSGE SVFLLGPPGI AKSLIARRLK FAFQNARAFE YLMTRFSTPE EVFGPLSIQ ALKDEGRYER LTSGYLPEAE IVFLDEIWKA GPAILNTLLT AINERQFRNG AHVEKIPMRL LVAASNELPE A DSSLEALY ...文字列: MAHPHLLAER ISRLSSSLEK GLYERSHAIR LCLLAALSGE SVFLLGPPGI AKSLIARRLK FAFQNARAFE YLMTRFSTPE EVFGPLSIQ ALKDEGRYER LTSGYLPEAE IVFLDEIWKA GPAILNTLLT AINERQFRNG AHVEKIPMRL LVAASNELPE A DSSLEALY DRMLIRLWLD KVQDKANFRS MLTSQQDEND NPVPDALQVT DEEYERWQKE IGEITLPDHV FELIFMLRQQ LD KLPDAPY VSDRRWKKAI RLLQASAFFS GRSAVAPVDL ILLKDCLWYD AQSLNLIQQQ IDVLMTGHAW QQQGMLTRLG AIV QRHLQL QQQQSDKTAL TVIRLGGIFS RRQQYQLPVN VTASTLTLLL QKPLKLHDME VVHISFERSA LEQWLSKGGE IRGK LNGIG FAQKLNLEVD SAQHLVVRDV SLQGSTLALP GSSAEGLPGE IKQQLEELES DWRKQHALFS EQQKCLFIPG DWLGR IEAS LQDVGAQIRQ AQQ |
-分子 #3: PYRIDOXAL-5'-PHOSPHATE
| 分子 | 名称: PYRIDOXAL-5'-PHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 20 / 式: PLP |
|---|---|
| 分子量 | 理論値: 247.142 Da |
| Chemical component information |  ChemComp-PLP: |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 5 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.98 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 詳細: 20 mM Tris pH 7.9, 300 mM NaCl, 2 mM ADP, 10 mM MgCl 2 , 0.1 mM PLP and 1 mM DTT |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Concentrations of LdcI and RavA were 0.38 mg/ml (4.67 microM) and 0.6 mg/ml (10.64 microM) respectively. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 41270 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 詳細 | Data collection was performed on an FEI Polara microscope operated at 300 kV. Movies of 40 frames were collected with a total exposure time of 8s and a total dose of 40e-/Angstrom^2 on a K2 summit direct electron detector (Gatan) at a magnification of 41270x, corresponding to 1.21 Angstrom/pixel at the specimen level. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 3-40 / 実像数: 1819 / 平均露光時間: 8.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | (PDB ID: , ) |
|---|---|
| 詳細 | Local resolution estimation and subsequent filtering of maps were performed in Relion3.0. For fitting of atomic models in the resulting filtered maps, we used the previously-determined X-ray structures of LdcI (PDB ID: 3N75) (Kanjee et al., 2011) and RavA (PDB ID: 3NBX) (El Bakkouri et al., 2010). In each map, two decameric LdcI molecules extracted from PDB 3N75 and one spiral RavA hexamer extracted from a continuous RavA helix generated from PDB 3NBX were fitted separately using iMODFIT (Lopez-Blanco and Chacon, 2013), followed by a single round of B-factor (ADP) refinement in Phenix. |
| 精密化 | プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-6q7m: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X