+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4319 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | uL23 beta hairpin loop deletion of E.coli ribosome | |||||||||
 マップデータ マップデータ | Sharpened map of E.coli ribosome with the beta-hairpin of uL23 deleted. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 ribosomal large subunit assembly / cytosolic large ribosomal subunit / cytoplasmic translation / ribosomal large subunit assembly / cytosolic large ribosomal subunit / cytoplasmic translation /  rRNA binding / rRNA binding /  リボソーム / structural constituent of ribosome / リボソーム / structural constituent of ribosome /  翻訳 (生物学) / 翻訳 (生物学) /  ribonucleoprotein complex / ribonucleoprotein complex /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /   Escherichia coli O157:H7 (大腸菌) Escherichia coli O157:H7 (大腸菌) | |||||||||
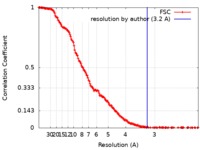
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.2 Å クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Kudva R / von Heijne G / Carroni M | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2018 ジャーナル: Elife / 年: 2018タイトル: The shape of the bacterial ribosome exit tunnel affects cotranslational protein folding. 著者: Renuka Kudva / Pengfei Tian / Fátima Pardo-Avila / Marta Carroni / Robert B Best / Harris D Bernstein / Gunnar von Heijne /   要旨: The ribosome exit tunnel can accommodate small folded proteins, while larger ones fold outside. It remains unclear, however, to what extent the geometry of the tunnel influences protein folding. ...The ribosome exit tunnel can accommodate small folded proteins, while larger ones fold outside. It remains unclear, however, to what extent the geometry of the tunnel influences protein folding. Here, using ribosomes with deletions in loops in proteins uL23 and uL24 that protrude into the tunnel, we investigate how tunnel geometry determines where proteins of different sizes fold. We find that a 29-residue zinc-finger domain normally folding close to the uL23 loop folds deeper in the tunnel in uL23 Δloop ribosomes, while two ~ 100 residue proteins normally folding close to the uL24 loop near the tunnel exit port fold at deeper locations in uL24 Δloop ribosomes, in good agreement with results obtained by coarse-grained molecular dynamics simulations. This supports the idea that cotranslational folding commences once a protein domain reaches a location in the exit tunnel where there is sufficient space to house the folded structure. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4319.map.gz emd_4319.map.gz | 347.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4319-v30.xml emd-4319-v30.xml emd-4319.xml emd-4319.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4319_fsc.xml emd_4319_fsc.xml | 19.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4319.png emd_4319.png | 172.3 KB | ||
| マスクデータ |  emd_4319_msk_1.map emd_4319_msk_1.map | 371.3 MB |  マスクマップ マスクマップ | |
| その他 |  emd_4319_half_map_1.map.gz emd_4319_half_map_1.map.gz emd_4319_half_map_2.map.gz emd_4319_half_map_2.map.gz | 344.3 MB 344.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4319 http://ftp.pdbj.org/pub/emdb/structures/EMD-4319 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4319 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4319 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4319.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4319.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of E.coli ribosome with the beta-hairpin of uL23 deleted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4319_msk_1.map emd_4319_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
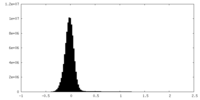
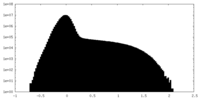
| 密度ヒストグラム |
-ハーフマップ: Half map A of E.coli ribosome with the beta-hairpin of uL23 deleted.
| ファイル | emd_4319_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of E.coli ribosome with the beta-hairpin of uL23 deleted. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B of E.coli ribosome with the beta-hairpin of uL23 deleted.
| ファイル | emd_4319_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of E.coli ribosome with the beta-hairpin of uL23 deleted. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : E.coli ribosome with beta-hairpin of uL23 deleted
| 全体 | 名称: E.coli ribosome with beta-hairpin of uL23 deleted |
|---|---|
| 要素 |
|
-超分子 #1: E.coli ribosome with beta-hairpin of uL23 deleted
| 超分子 | 名称: E.coli ribosome with beta-hairpin of uL23 deleted / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Deposited PDB is of the uL23 with the beta-hairpin loop deleted. |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 2.6 MDa |
-分子 #1: 50S ribosomal protein L23
| 分子 | 名称: 50S ribosomal protein L23 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli O157:H7 (大腸菌) Escherichia coli O157:H7 (大腸菌) |
| 分子量 | 理論値: 9.324003 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MIREERLLKV LRAPHVSEKA STAMEKSNTI VLKVAKDATK AEIKAAVQKL FEVEVEVVNT LVVKRRSDWK KAYVTLKEGQ NL |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 支持フィルム - Film thickness: 0.02 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.0003 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 3500 / 平均電子線量: 1.17 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
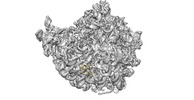
| 得られたモデル |  PDB-6fu8: |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X