+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4174 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Ageratum Yellow Vein virus (AYVV) | ||||||||||||||||||
 マップデータ マップデータ | Ageratum Yellow Vein virus cryoEM density map | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=1 icosahedral viral capsid / viral penetration into host nucleus / symbiont entry into host cell / host cell nucleus / structural molecule activity /  DNA binding / DNA binding /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Ageratum yellow vein virus (ウイルス) Ageratum yellow vein virus (ウイルス) | ||||||||||||||||||
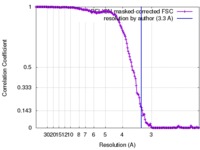
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Hesketh EL / Saunders K / Fisher C / Potze J / Stanley J / Lomonossoff GP / Ranson NA | ||||||||||||||||||
| 資金援助 |  英国, 5件 英国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: The 3.3 Å structure of a plant geminivirus using cryo-EM. 著者: Emma L Hesketh / Keith Saunders / Chloe Fisher / Joran Potze / John Stanley / George P Lomonossoff / Neil A Ranson /  要旨: Geminiviruses are major plant pathogens that threaten food security globally. They have a unique architecture built from two incomplete icosahedral particles, fused to form a geminate capsid. ...Geminiviruses are major plant pathogens that threaten food security globally. They have a unique architecture built from two incomplete icosahedral particles, fused to form a geminate capsid. However, despite their importance to agricultural economies and fundamental biological interest, the details of how this is realized in 3D remain unknown. Here we report the structure of Ageratum yellow vein virus at 3.3 Å resolution, using single-particle cryo-electron microscopy, together with an atomic model that shows that the N-terminus of the single capsid protein (CP) adopts three different conformations essential for building the interface between geminate halves. Our map also contains density for ~7 bases of single-stranded DNA bound to each CP, and we show that the interactions between the genome and CPs are different at the interface than in the rest of the capsid. With additional mutagenesis data, this suggests a central role for DNA binding-induced conformational change in directing the assembly of geminate capsids. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4174.map.gz emd_4174.map.gz | 197.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4174-v30.xml emd-4174-v30.xml emd-4174.xml emd-4174.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4174_fsc.xml emd_4174_fsc.xml | 15.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4174.png emd_4174.png | 194.4 KB | ||
| その他 |  emd_4174_additional.map.gz emd_4174_additional.map.gz | 273.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4174 http://ftp.pdbj.org/pub/emdb/structures/EMD-4174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4174 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4174.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4174.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Ageratum Yellow Vein virus cryoEM density map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.065 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Ageratum Yellow Vein virus unsharpened
| ファイル | emd_4174_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Ageratum Yellow Vein virus unsharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ageratum yellow vein virus
| 全体 | 名称:   Ageratum yellow vein virus (ウイルス) Ageratum yellow vein virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Ageratum yellow vein virus
| 超分子 | 名称: Ageratum yellow vein virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 / NCBI-ID: 44560 / 生物種: Ageratum yellow vein virus / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Ageratum (カッコウアザミ属) Ageratum (カッコウアザミ属) |
| 分子量 | 理論値: 251 KDa |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Ageratum yellow vein virus (ウイルス) Ageratum yellow vein virus (ウイルス) |
| 分子量 | 理論値: 22.500703 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: PDVPKGCEGP CKVQSYEQRH DISHVGKVLC VSDVTRGNGL THRVGKRFCV KSVYVLGKIW MDENIKTKNH TNTVMFYLVR DRRPFGTAM DFGQVFNMYD NEPSTATIKN DLRDRYQVLR KFTSTVTGGQ YASKEQALVK KFMKINNYVV YNHQEAAKYD N HTENALLL ...文字列: PDVPKGCEGP CKVQSYEQRH DISHVGKVLC VSDVTRGNGL THRVGKRFCV KSVYVLGKIW MDENIKTKNH TNTVMFYLVR DRRPFGTAM DFGQVFNMYD NEPSTATIKN DLRDRYQVLR KFTSTVTGGQ YASKEQALVK KFMKINNYVV YNHQEAAKYD N HTENALLL YMACTHASNP VYATLKIRIY FYDSVQN |
-分子 #3: coat protein subunit I
| 分子 | 名称: coat protein subunit I / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Ageratum yellow vein virus (ウイルス) Ageratum yellow vein virus (ウイルス) |
| 分子量 | 理論値: 23.644094 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: RLYRMYRTPD VPKGCEGPCK VQSYEQRHDI SHVGKVLCVS DVTRGNGLTH RVGKRFCVKS VYVLGKIWMD ENIKTKNHTN TVMFYLVRD RRPFGTAMDF GQVFNMYDNE PSTATIKNDL RDRYQVLRKF TSTVTGGQYA SKEQALVKKF MKINNYVVYN H QEAAKYDN ...文字列: RLYRMYRTPD VPKGCEGPCK VQSYEQRHDI SHVGKVLCVS DVTRGNGLTH RVGKRFCVKS VYVLGKIWMD ENIKTKNHTN TVMFYLVRD RRPFGTAMDF GQVFNMYDNE PSTATIKNDL RDRYQVLRKF TSTVTGGQYA SKEQALVKKF MKINNYVVYN H QEAAKYDN HTENALLLYM ACTHASNPVY ATLKIRIYFY DSVQN |
-分子 #4: coat protein subunit H
| 分子 | 名称: coat protein subunit H / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Ageratum yellow vein virus (ウイルス) Ageratum yellow vein virus (ウイルス) |
| 分子量 | 理論値: 25.664473 KDa |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
| 配列 | 文字列: NRRRTWTNRP MYRKPRLYRM YRTPDVPKGC EGPCKVQSYE QRHDISHVGK VLCVSDVTRG NGLTHRVGKR FCVKSVYVLG KIWMDENIK TKNHTNTVMF YLVRDRRPFG TAMDFGQVFN MYDNEPSTAT IKNDLRDRYQ VLRKFTSTVT GGQYASKEQA L VKKFMKIN ...文字列: NRRRTWTNRP MYRKPRLYRM YRTPDVPKGC EGPCKVQSYE QRHDISHVGK VLCVSDVTRG NGLTHRVGKR FCVKSVYVLG KIWMDENIK TKNHTNTVMF YLVRDRRPFG TAMDFGQVFN MYDNEPSTAT IKNDLRDRYQ VLRKFTSTVT GGQYASKEQA L VKKFMKIN NYVVYNHQEA AKYDNHTENA LLLYMACTHA SNPVYATLKI RIYFYDSVQN |
-分子 #2: ssDNA loop
| 分子 | 名称: ssDNA loop / タイプ: dna / ID: 2 / コピー数: 10 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Ageratum yellow vein virus (ウイルス) Ageratum yellow vein virus (ウイルス) |
| 分子量 | 理論値: 2.05139 KDa |
| 配列 | 文字列: (DC)(DA)(DA)(DC)(DC)(DA)(DC) |
-分子 #5: ssDNA loop associated with subunit H
| 分子 | 名称: ssDNA loop associated with subunit H / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Ageratum yellow vein virus (ウイルス) Ageratum yellow vein virus (ウイルス) |
| 分子量 | 理論値: 1.762208 KDa |
| 配列 | 文字列: (DC)(DA)(DA)(DC)(DC)(DA) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 構成要素 - 濃度: 100.0 mM / 構成要素 - 式: NaPo4 / 構成要素 - 名称: Sodium phosphate |
|---|---|
| グリッド | 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 支持フィルム - Film thickness: 2000.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.037 kPa / 詳細: PELCO easiglow |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 5.0 µm / 最小 デフォーカス(補正後): 0.3 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 75000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 78.0 K / 最高: 78.0 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 12028 / 平均露光時間: 2.0 sec. / 平均電子線量: 110.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-6f2s: |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X