+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4102 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of F-ATPase from Pichia angusta, state1 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 angiostatin binding / ATPase inhibitor activity / negative regulation of hydrolase activity / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / heme biosynthetic process / mitochondrial proton-transporting ATP synthase complex / negative regulation of endothelial cell proliferation / mitochondrial nucleoid ... angiostatin binding / ATPase inhibitor activity / negative regulation of hydrolase activity / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / heme biosynthetic process / mitochondrial proton-transporting ATP synthase complex / negative regulation of endothelial cell proliferation / mitochondrial nucleoid ... angiostatin binding / ATPase inhibitor activity / negative regulation of hydrolase activity / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / heme biosynthetic process / mitochondrial proton-transporting ATP synthase complex / negative regulation of endothelial cell proliferation / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton-transporting ATP synthase complex, coupling factor F(o) / proton motive force-driven ATP synthesis / proton transmembrane transporter activity / proton-transporting ATP synthase complex, catalytic core F(1) / angiostatin binding / ATPase inhibitor activity / negative regulation of hydrolase activity / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / heme biosynthetic process / mitochondrial proton-transporting ATP synthase complex / negative regulation of endothelial cell proliferation / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton-transporting ATP synthase complex, coupling factor F(o) / proton motive force-driven ATP synthesis / proton transmembrane transporter activity / proton-transporting ATP synthase complex, catalytic core F(1) /  ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism /  赤血球形成 / 赤血球形成 /  ADP binding / ADP binding /  ミトコンドリア / ミトコンドリア /  ATPase binding / protein homotetramerization / ATPase binding / protein homotetramerization /  ミトコンドリア内膜 / membrane => GO:0016020 / ミトコンドリア内膜 / membrane => GO:0016020 /  calmodulin binding / calmodulin binding /  hydrolase activity / hydrolase activity /  lipid binding / structural molecule activity / lipid binding / structural molecule activity /  細胞膜 / 細胞膜 /  ATP hydrolysis activity / protein homodimerization activity / protein-containing complex / ATP hydrolysis activity / protein homodimerization activity / protein-containing complex /  ミトコンドリア / ミトコンドリア /  ATP binding / identical protein binding ATP binding / identical protein binding類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Ogataea angusta (菌類) Ogataea angusta (菌類) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.0 Å クライオ電子顕微鏡法 / 解像度: 7.0 Å | ||||||||||||
 データ登録者 データ登録者 | Vinothkumar KR / Montgomery MG / Liu S / Walker JE | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2016 ジャーナル: Proc Natl Acad Sci U S A / 年: 2016タイトル: Structure of the mitochondrial ATP synthase from determined by electron cryo-microscopy. 著者: Kutti R Vinothkumar / Martin G Montgomery / Sidong Liu / John E Walker /  要旨: The structure of the intact monomeric ATP synthase from the fungus, , has been solved by electron cryo-microscopy. The structure provides insights into the mechanical coupling of the transmembrane ...The structure of the intact monomeric ATP synthase from the fungus, , has been solved by electron cryo-microscopy. The structure provides insights into the mechanical coupling of the transmembrane proton motive force across mitochondrial membranes in the synthesis of ATP. This mechanism requires a strong and integral stator, consisting of the catalytic αβ-domain, peripheral stalk, and, in the membrane domain, subunit a and associated supernumerary subunits, kept in contact with the rotor turning at speeds up to 350 Hz. The stator's integrity is ensured by robust attachment of both the oligomycin sensitivity conferral protein (OSCP) to the catalytic domain and the membrane domain of subunit b to subunit a. The ATP8 subunit provides an additional brace between the peripheral stalk and subunit a. At the junction between the OSCP and the apparently stiff, elongated α-helical b-subunit and associated d- and h-subunits, an elbow or joint allows the stator to bend to accommodate lateral movements during the activity of the catalytic domain. The stator may also apply lateral force to help keep the static a-subunit and rotating c-ring together. The interface between the c-ring and the a-subunit contains the transmembrane pathway for protons, and their passage across the membrane generates the turning of the rotor. The pathway has two half-channels containing conserved polar residues provided by a bundle of four α-helices inclined at ∼30° to the plane of the membrane, similar to those described in other species. The structure provides more insights into the workings of this amazing machine. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4102.map.gz emd_4102.map.gz | 91.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4102-v30.xml emd-4102-v30.xml emd-4102.xml emd-4102.xml | 32.2 KB 32.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
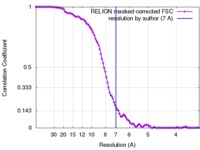
| FSC (解像度算出) |  emd_4102_fsc.xml emd_4102_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4102.png emd_4102.png | 52.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4102 http://ftp.pdbj.org/pub/emdb/structures/EMD-4102 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4102 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4102 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4102.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4102.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.75 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Yeast F1FO ATP Synthase
+超分子 #1: Yeast F1FO ATP Synthase
+分子 #1: ATP synthase subunit f
+分子 #2: ATP synthase subunit AAP1
+分子 #3: ATP synthase subunit a
+分子 #4: ATP synthase subunit b
+分子 #5: ATP synthase alpha subunit
+分子 #6: ATP synthase beta subunit
+分子 #7: ATP synthase gamma subunit
+分子 #8: ATP synthase delta subunit
+分子 #9: ATP synthase epsilon subunit
+分子 #10: ATP synthase inhibitor protein IF1
+分子 #11: ATP synthase subunit c
+分子 #12: ATP synthase OSCP subunit
+分子 #13: ATP synthase subunit b
+分子 #14: ATP synthase subunit d
+分子 #15: ATP synthase subunit h
+分子 #16: ATP synthase subunit a
+分子 #17: ATP synthase subunit a
+分子 #18: ADENOSINE-5'-TRIPHOSPHATE
+分子 #19: MAGNESIUM ION
+分子 #20: ADENOSINE-5'-DIPHOSPHATE
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER 詳細: Grids were blotted for 12-14 seconds before plunging.. | ||||||||||||
| 詳細 | The enzyme with bound inhibitor protein extracted and purified in Cymal-7. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 81395 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 47000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 87.5 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / デジタル化 - 画像ごとのフレーム数: 1-32 / 平均露光時間: 4.0 sec. / 平均電子線量: 64.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | The refinement of whole data was done at 1.72 A sampling. The map was scaled to 1.75 A after comparison with the model. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-5lqz: |
 ムービー
ムービー コントローラー
コントローラー