+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3980 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Post-catalytic P complex spliceosome with 3' splice site undocked | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
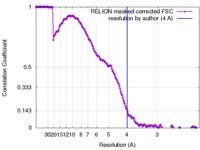
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.0 Å クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Wilkinson ME / Fica SM / Galej WP / Norman CM / Newman AJ / Nagai K | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2017 ジャーナル: Science / 年: 2017タイトル: Postcatalytic spliceosome structure reveals mechanism of 3'-splice site selection. 著者: Max E Wilkinson / Sebastian M Fica / Wojciech P Galej / Christine M Norman / Andrew J Newman / Kiyoshi Nagai /  要旨: Introns are removed from eukaryotic messenger RNA precursors by the spliceosome in two transesterification reactions-branching and exon ligation. The mechanism of 3'-splice site recognition during ...Introns are removed from eukaryotic messenger RNA precursors by the spliceosome in two transesterification reactions-branching and exon ligation. The mechanism of 3'-splice site recognition during exon ligation has remained unclear. Here we present the 3.7-angstrom cryo-electron microscopy structure of the yeast P-complex spliceosome immediately after exon ligation. The 3'-splice site AG dinucleotide is recognized through non-Watson-Crick pairing with the 5' splice site and the branch-point adenosine. After the branching reaction, protein factors work together to remodel the spliceosome and stabilize a conformation competent for 3'-splice site docking, thereby promoting exon ligation. The structure accounts for the strict conservation of the GU and AG dinucleotides at the 5' and 3' ends of introns and provides insight into the catalytic mechanism of exon ligation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3980.map.gz emd_3980.map.gz | 392.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3980-v30.xml emd-3980-v30.xml emd-3980.xml emd-3980.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3980_fsc.xml emd_3980_fsc.xml | 17 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3980.png emd_3980.png | 68.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3980 http://ftp.pdbj.org/pub/emdb/structures/EMD-3980 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3980 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3980 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3980.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3980.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : P complex spliceosome with 3' splice site undocked
| 全体 | 名称: P complex spliceosome with 3' splice site undocked |
|---|---|
| 要素 |
|
-超分子 #1: P complex spliceosome with 3' splice site undocked
| 超分子 | 名称: P complex spliceosome with 3' splice site undocked / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#30 詳細: P complex was assembled in vitro in splicing extract supplemented with dominant negative mutant Prp22 protein. P complex was purified via MS2-MBP fusion protein bound to 3 MS2 hairpin loops on the mRNA. |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 2.2 MDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.4 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.9 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 7.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III 詳細: 3 microlitres sample were applied to the grid, left for 15 seconds and then blotted for 3.0 seconds before plunging.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-20 / 撮影したグリッド数: 1 / 実像数: 2295 / 平均露光時間: 12.0 sec. / 平均電子線量: 47.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー