+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3877 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Inner membrane Bcs complex (E. coli) | |||||||||||||||
 マップデータ マップデータ | An inner membrane subcomplex of the E. coli cellulose secretion system (Bcs). The complex was purified from solubilised membranes of Bl21(DE3) cells over-expressing the BcsRQAB subunits. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||||||||
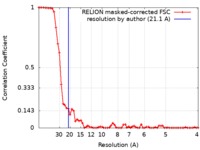
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 21.1 Å ネガティブ染色法 / 解像度: 21.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Krasteva PV / Fronzes R | |||||||||||||||
| 資金援助 |  フランス, 4件 フランス, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Insights into the structure and assembly of a bacterial cellulose secretion system. 著者: Petya Violinova Krasteva / Joaquin Bernal-Bayard / Laetitia Travier / Fernando Ariel Martin / Pierre-Alexandre Kaminski / Gouzel Karimova / Rémi Fronzes / Jean-Marc Ghigo /   要旨: Secreted exopolysaccharides present important determinants for bacterial biofilm formation, survival, and virulence. Cellulose secretion typically requires the concerted action of a c-di-GMP- ...Secreted exopolysaccharides present important determinants for bacterial biofilm formation, survival, and virulence. Cellulose secretion typically requires the concerted action of a c-di-GMP-responsive inner membrane synthase (BcsA), an accessory membrane-anchored protein (BcsB), and several additional Bcs components. Although the BcsAB catalytic duo has been studied in great detail, its interplay with co-expressed subunits remains enigmatic. Here we show that E. coli Bcs proteins partake in a complex protein interaction network. Electron microscopy reveals a stable, megadalton-sized macromolecular assembly, which encompasses most of the inner membrane and cytosolic Bcs components and features a previously unobserved asymmetric architecture. Heterologous reconstitution and mutational analyses point toward a structure-function model, where accessory proteins regulate secretion by affecting both the assembly and stability of the system. Altogether, these results lay the foundation for more comprehensive models of synthase-dependent exopolysaccharide secretion in biofilms and add a sophisticated secretory nanomachine to the diverse bacterial arsenal for virulence and adaptation. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3877.map.gz emd_3877.map.gz | 8.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3877-v30.xml emd-3877-v30.xml emd-3877.xml emd-3877.xml | 13.3 KB 13.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3877_fsc.xml emd_3877_fsc.xml | 6.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3877.png emd_3877.png | 31.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3877 http://ftp.pdbj.org/pub/emdb/structures/EMD-3877 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3877 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3877 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3877.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3877.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | An inner membrane subcomplex of the E. coli cellulose secretion system (Bcs). The complex was purified from solubilised membranes of Bl21(DE3) cells over-expressing the BcsRQAB subunits. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.9 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bcs inner membrane sub-complex.
| 全体 | 名称: Bcs inner membrane sub-complex. |
|---|---|
| 要素 |
|
-超分子 #1: Bcs inner membrane sub-complex.
| 超分子 | 名称: Bcs inner membrane sub-complex. / タイプ: complex / ID: 1 / 親要素: 0 詳細: Sample purified using BcsA as bait from solubilised E. coli Bl21(DE3) cells over-expressing the BcsRQAB sub-units of cellulose-secreting E. coli 1094. |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: 1094 / 細胞中の位置: inner membrane Escherichia coli (大腸菌) / 株: 1094 / 細胞中の位置: inner membrane |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: Bl21(DE3) / 組換プラスミド: pCDF-Duet Escherichia coli (大腸菌) / 組換株: Bl21(DE3) / 組換プラスミド: pCDF-Duet |
| 分子量 | 理論値: 1 MDa |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM HEPES 8.0 120 mM NaCl 10 glycerol 5 mM MgCl2 10 uM AppCp 2 uM c-di-GMP 0.008% LM-NPG cOmplete protease inhibitors |
| 染色 | タイプ: NEGATIVE / 材質: uranyl acetate 詳細: 5 ul of sample were spotted on glow-discharged carbon-coated copper grids and incubated for 1 minute. Liquid was blotted off and the sample was passed through 3 drops of 10 ul 2% uranyl ...詳細: 5 ul of sample were spotted on glow-discharged carbon-coated copper grids and incubated for 1 minute. Liquid was blotted off and the sample was passed through 3 drops of 10 ul 2% uranyl acetate with a 30-second incubation over the third. After that the stain was blotted off and the grid was air-dried. |
| グリッド | 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER |
| 詳細 | Samples were purified using anti-FLAG affinity gel from detergent-solubilised membranes of E. coli(DE3) cells over-expressing the BcsRQAB subunits of an E. coli 1094-derived strain (pCDF vector, IPTG-inducible overnight expression in TB, affinity tag on BcsA). Eluted samples were further purified by centrifugation over a 10-40% glycerol density gradient. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.5 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 Bright-field microscopy / 最大 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 撮影したグリッド数: 2 / 平均電子線量: 12.0 e/Å2 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー