+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3824 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the SAGA and NuA4 coactivator subunit Tra1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SLIK (SAGA-like) complex / SAGA complex /  NuA4 histone acetyltransferase complex / NuA4 histone acetyltransferase complex /  クロマチンリモデリング / クロマチンリモデリング /  DNA修復 / DNA-templated transcription / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / DNA修復 / DNA-templated transcription / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) | |||||||||
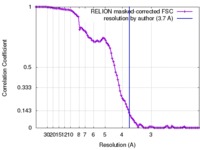
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Diaz-Santin LM / Lukoyanova N / Aciyan E / Cheung ACM | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2017 ジャーナル: Elife / 年: 2017タイトル: Cryo-EM structure of the SAGA and NuA4 coactivator subunit Tra1 at 3.7 angstrom resolution. 著者: Luis Miguel Díaz-Santín / Natasha Lukoyanova / Emir Aciyan / Alan Cm Cheung /  要旨: Coactivator complexes SAGA and NuA4 stimulate transcription by post-translationally modifying chromatin. Both complexes contain the Tra1 subunit, a highly conserved 3744-residue protein from the ...Coactivator complexes SAGA and NuA4 stimulate transcription by post-translationally modifying chromatin. Both complexes contain the Tra1 subunit, a highly conserved 3744-residue protein from the Phosphoinositide 3-Kinase-related kinase (PIKK) family and a direct target for multiple sequence-specific activators. We present the Cryo-EM structure of Tra1 to 3.7 Å resolution, revealing an extensive network of alpha-helical solenoids organized into a diamond ring conformation and is strikingly reminiscent of DNA-PKcs, suggesting a direct role for Tra1 in DNA repair. The structure was fitted into an existing SAGA EM reconstruction and reveals limited contact surfaces to Tra1, hence it does not act as a molecular scaffold within SAGA. Mutations that affect activator targeting are distributed across the Tra1 structure, but also cluster within the N-terminal Finger region, indicating the presence of an activator interaction site. The structure of Tra1 is a key milestone in deciphering the mechanism of multiple coactivator complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3824.map.gz emd_3824.map.gz | 7.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3824-v30.xml emd-3824-v30.xml emd-3824.xml emd-3824.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3824_fsc.xml emd_3824_fsc.xml | 10.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3824.png emd_3824.png | 156.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3824 http://ftp.pdbj.org/pub/emdb/structures/EMD-3824 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3824 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3824 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3824.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3824.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tra1 - Transcription-associated protein 1
| 全体 | 名称: Tra1 - Transcription-associated protein 1 |
|---|---|
| 要素 |
|
-超分子 #1: Tra1 - Transcription-associated protein 1
| 超分子 | 名称: Tra1 - Transcription-associated protein 1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 433 KDa |
-分子 #1: Transcription-associated protein 1
| 分子 | 名称: Transcription-associated protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) |
| 分子量 | 理論値: 436.527281 KDa |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: MDYKDHDGDY KDHDIDYKDD DDKMSLTEQI EQFASRFRDD DATLQSRYST LSELYDIMEL LNSPEDYHFF LQAVIPLLLN QLKEVPISY DAHSPEQKLR NSMLDIFNRC LMNQTFQPYA MEVLEFLLSV LPKENEENGI LCMKVLTTLF KSFKSILQDK L DSFIRIII ...文字列: MDYKDHDGDY KDHDIDYKDD DDKMSLTEQI EQFASRFRDD DATLQSRYST LSELYDIMEL LNSPEDYHFF LQAVIPLLLN QLKEVPISY DAHSPEQKLR NSMLDIFNRC LMNQTFQPYA MEVLEFLLSV LPKENEENGI LCMKVLTTLF KSFKSILQDK L DSFIRIII QIYKNTPNLI NQTFYEAGKA EQGDLDSPKE PQADELLDEF SKNDEEKDFP SKQSSTEPRF ENSTSSNGLR SS MFSFKIL SECPITMVTL YSSYKQLTST SLPEFTPLIM NLLNIQIKQQ QEAREQAESR GEHFTSISTE IINRPAYCDF ILA QIKATS FLAYVFIRGY APEFLQDYVN FVPDLIIRLL QDCPSELSSA RKELLHATRH ILSTNYKKLF LPKLDYLFDE RILI GNGFT MHETLRPLAY STVADFIHNI RSELQLSEIE KTIKIYTGYL LDESLALTVQ IMSAKLLLNL VERILKLGKE NPQEA PRAK KLLMIIIDSY MNRFKTLNRQ YDTIMKYYGR YETHKKEKAE KLKNSIQDND KESEEFMRKV LEPSDDDHLM PQPKKE DIN DSPDVEMTES DKVVKNDVEM FDIKNYAPIL LLPTPTNDPI KDAFYLYRTL MSFLKTIIHD LKVFNPPPNE YTVANPK LW ASVSRVFSYE EVIVFKDLFH ECIIGLKFFK DHNEKLSPET TKKHFDISMP SLPVSATKDA RELMDYLAFM FMQMDNAT F NEIIEQELPF VYERMLEDSG LLHVAQSFLT SEITSPNFAG ILLRFLKGKL KDLGNVDFNT SNVLIRLFKL SFMSVNLFP NINEVVLLPH LNDLILNSLK YSTTAEEPLV YFYLIRTLFR SIGGGRFENL YRSIKPILQV LLQSLNQMIL TARLPHEREL YVELCITVP VRLSVLAPYL PFLMKPLVFA LQQYPDLVSQ GLRTLELCID NLTAEYFDPI IEPVIDDVSK ALFNLLQPQP F NHAISHNV VRILGKLGGR NRQFLKPPTD LTEKTELDID AIADFKINGM PEDVPLSVTP GIQSALNILQ SYKSDIHYRK SA YKYLTCV LLLMTKSSAE FPTNYTELLK TAVNSIKLER IGIEKNFDLE PTVNKRDYSN QENLFLRLLE SVFYATSIKE LKD DAMDLL NNLLDHFCLL QVNTTLLNKR NYNGTFNIDL KNPNFMLDSS LILDAIPFAL SYYIPEVREV GVLAYKRIYE KSCL IYGEE LALSHSFIPE LAKQFIHLCY DETYYNKRGG VLGIKVLIDN VKSSSVFLKK YQYNLANGLL FVLKDTQSEA PSAIT DSAE KLLIDLLSIT FADVKEEDLG NKVLENTLTD IVCELSNANP KVRNACQKSL HTISNLTGIP IVKLMDHSKQ FLLSPI FAK PLRALPFTMQ IGNVDAITFC LSLPNTFLTF NEELFRLLQE SIVLADAEDE SLSTNIQKTT EYSTSEQLVQ LRIACIK LL AIALKNEEFA TAQQGNIRIR ILAVFFKTML KTSPEIINTT YEALKGSLAE NSKLPKELLQ NGLKPLLMNL SDHQKLTV P GLDALSKLLE LLIAYFKVEI GRKLLDHLTA WCRVEVLDTL FGQDLAEQMP TKIIVSIINI FHLLPPQADM FLNDLLLKV MLLERKLRLQ LDSPFRTPLA RYLNRFHNPV TEYFKKNMTL RQLVLFMCNI VQRPEAKELA EDFEKELDNF YDFYISNIPK NQVRVVSFF TNMVDLFNTM VITNGDEWLK KKGNMILKLK DMLNLTLKTI KENSFYIDHL QLNQSIAKFQ ALYLRFTELS E RDQNPLLL DFIDFSFSNG IKASYSLKKF IFHNIIASSN KEKQNNFIND ATLFVLSDKC LDARIFVLKN VINSTLIYEV AT SGSLKSY LVEDKKPKWL ELLHNKIWKN SNAILAYDVL DHHDLFRFEL LQLSAIFIKA DPEIIAEIKK DIIKFCWNFI KLE DTLIKQ SAYLVTSYFI SKFDFPIKVV TQVFVALLRS SHVEARYLVK QSLDVLTPVL HERMNAAGTP DTWINWVKRV MVEN SSSQN NILYQFLISH PDLFFNSRDL FISNIIHHMN KITFMSNSNS DSHTLAIDLA SLILYWENKT LEITNVNNTK TDSDG DVVM SDSKSDINPV EADTTAIIVD ANNNSPISLH LREACTAFLI RYVCASNHRA IETELGLRAI NILSELISDK HWTNVN VKL VYFEKFLIFQ DLDSENILYY CMNALDVLYV FFKNKTKEWI MENLPTIQNL LEKCIKSDHH DVQEALQKVL QVIMKAI KA QGVSVIIEEE SPGKTFIQML TSVITQDLQE TSSVTAGVTL AWVLFMNFPD NIVPLLTPLM KTFSKLCKDH LSISQPKD A MALEEARITT KLLEKVLYIL SLKVSLLGDS RRPFLSTVAL LIDHSMDQNF LRKIVNMSRS WIFNTEIFPT VKEKAAILT KMLAFEIRGE PSLSKLFYEI VLKLFDQEHF NNTEITVRME QPFLVGTRVE DIGIRKRFMT ILDNSLERDI KERLYYVIRD QNWEFIADY PWLNQALQLL YGSFNREKEL SLKNIYCLSP PSILQEYLPE NAEMVTEVND LELSNFVKGH IASMQGLCRI I SSDFIDSL IEIFYQDPKA IHRAWVTLFP QVYKSIPKNE KYGFVRSIIT LLSKPYHTRQ ISSRTNVINM LLDSISKIES LE LPPHLVK YLAISYNAWY QSINILESIQ SNTSIDNTKI IEANEDALLE LYVNLQEEDM FYGLWRRRAK YTETNIGLSY EQI GLWDKA QQLYEVAQVK ARSGALPYSQ SEYALWEDNW IQCAEKLQHW DVLTELAKHE GFTDLLLECG WRVADWNSDR DALE QSVKS VMDVPTPRRQ MFKTFLALQN FAESRKGDQE VRKLCDEGIQ LSLIKWVSLP IRYTPAHKWL LHGFQQYMEF LEATQ IYAN LHTTTVQNLD SKAQEIKRIL QAWRDRLPNT WDDVNMWNDL VTWRQHAFQV INNAYLPLIP ALQQSNSNSN INTHAY RGY HEIAWVINRF AHVARKHNMP DVCISQLARI YTLPNIEIQE AFLKLREQAK CHYQNMNELT TGLDVISNTN LVYFGTV QK AEFFTLKGMF LSKLRAYEEA NQAFATAVQI DLNLAKAWAQ WGFFNDRRLS EEPNNISFAS NAISCYLQAA GLYKNSKI R ELLCRILWLI SIDDASGMLT NAFDSFRGEI PVWYWITFIP QLLTSLSHKE ANMVRHILIR IAKSYPQALH FQLRTTKED FAVIQRQTMA VMGDKPDTND RNGRRQPWEY LQELNNILKT AYPLLALSLE SLVAQINDRF KSTTDEDLFR LINVLLIDGT LNYNRLPFP RKNPKLPENT EKNLVKFSTT LLAPYIRPKF NADFIDNKPD YETYIKRLRY WRRRLENKLD RASKKENLEV L CPHLSNFH HQKFEDIEIP GQYLLNKDNN VHFIKIARFL PTVDFVRGTH SSYRRLMIRG HDGSVHSFAV QYPAVRHSRR EE RMFQLYR LFNKSLSKNV ETRRRSIQFN LPIAIPLSPQ VRIMNDSVSF TTLHEIHNEF CKKKGFDPDD IQDFMADKLN AAH DDALPA PDMTILKVEI FNSIQTMFVP SNVLKDHFTS LFTQFEDFWL FRKQFASQYS SFVFMSYMMM INNRTPHKIH VDKT SGNVF TLEMLPSRFP YERVKPLLKN HDLSLPPDSP IFHNNEPVPF RLTPNIQSLI GDSALEGIFA VNLFTISRAL IEPDN ELNT YLALFIRDEI ISWFSNLHRP IIENPQLREM VQTNVDLIIR KVAQLGHLNS TPTVTTQFIL DCIGSAVSPR NLARTD VNF MPWF |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
| グリッド | モデル: Agar Scientific / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 94 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Two subsequent applications of protein were required to achieve the desired particle density on grids. Each application was followed by 20 sec waiting time, with a short 0.5 sec blotting ...詳細: Two subsequent applications of protein were required to achieve the desired particle density on grids. Each application was followed by 20 sec waiting time, with a short 0.5 sec blotting after first application and 5 sec blotting after the second.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 1.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-5ojs: |
 ムービー
ムービー コントローラー
コントローラー