+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3819 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of an RNA polymerase II-DSIF transcription elongation complex, DSIF-EC5 | ||||||||||||
 マップデータ マップデータ | Locally filtered map, B-factor 0 | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |   Bos taurus (ウシ) / Bos taurus (ウシ) /   Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | ||||||||||||
 データ登録者 データ登録者 | Bernecky C / Plitzko JM / Cramer P | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2017 ジャーナル: Nat Struct Mol Biol / 年: 2017タイトル: Structure of a transcribing RNA polymerase II-DSIF complex reveals a multidentate DNA-RNA clamp. 著者: Carrie Bernecky / Jürgen M Plitzko / Patrick Cramer /  要旨: During transcription, RNA polymerase II (Pol II) associates with the conserved elongation factor DSIF. DSIF renders the elongation complex stable and functions during Pol II pausing and RNA ...During transcription, RNA polymerase II (Pol II) associates with the conserved elongation factor DSIF. DSIF renders the elongation complex stable and functions during Pol II pausing and RNA processing. We combined cryo-EM and X-ray crystallography to determine the structure of the mammalian Pol II-DSIF elongation complex at a nominal resolution of 3.4 Å. Human DSIF has a modular structure with two domains forming a DNA clamp, two domains forming an RNA clamp, and one domain buttressing the RNA clamp. The clamps maintain the transcription bubble, position upstream DNA, and retain the RNA transcript in the exit tunnel. The mobile C-terminal region of DSIF is located near exiting RNA, where it can recruit factors for RNA processing. The structure provides insight into the roles of DSIF during mRNA synthesis. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3819.map.gz emd_3819.map.gz | 58.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3819-v30.xml emd-3819-v30.xml emd-3819.xml emd-3819.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3819.png emd_3819.png | 103.1 KB | ||
| マスクデータ |  emd_3819_msk_1.map emd_3819_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_3819_additional_1.map.gz emd_3819_additional_1.map.gz emd_3819_additional_2.map.gz emd_3819_additional_2.map.gz emd_3819_half_map_1.map.gz emd_3819_half_map_1.map.gz emd_3819_half_map_2.map.gz emd_3819_half_map_2.map.gz | 59.9 MB 59.7 MB 49.6 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3819 http://ftp.pdbj.org/pub/emdb/structures/EMD-3819 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3819 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3819 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3819.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3819.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally filtered map, B-factor 0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_3819_msk_1.map emd_3819_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
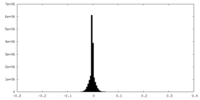
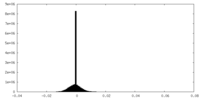
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: None
| ファイル | emd_3819_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: None
| ファイル | emd_3819_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Halfmap 1
| ファイル | emd_3819_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Halfmap 2
| ファイル | emd_3819_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNA polymerase II-DSIF elongation complex
| 全体 | 名称: RNA polymerase II-DSIF elongation complex |
|---|---|
| 要素 |
|
-超分子 #1: RNA polymerase II-DSIF elongation complex
| 超分子 | 名称: RNA polymerase II-DSIF elongation complex / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 分子量 | 理論値: 690 KDa |
-超分子 #2: RNA polymerase II
| 超分子 | 名称: RNA polymerase II / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
| 分子量 | 理論値: 520 KDa |
-超分子 #3: DSIF
| 超分子 | 名称: DSIF / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 分子量 | 理論値: 130 KDa |
-超分子 #4: DNA-RNA synthetic construct
| 超分子 | 名称: DNA-RNA synthetic construct / タイプ: complex / ID: 4 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 40 KDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.25 構成要素:
詳細: pH was adjusted at 25 degrees Celsius | |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Sample (four microliters) was applied to glow-discharged Quantifoil R 2/1 holey carbon grids, which were then blotted for 8.5s and plunge-frozen in liquid ethane.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.6 µm / 最小 デフォーカス(補正後): 0.6 µm / 倍率(補正後): 37037 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 37000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 37000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-33 / 撮影したグリッド数: 1 / 実像数: 2549 / 平均露光時間: 9.9 sec. / 平均電子線量: 33.0 e/Å2 詳細: Movies were aligned and binned to the physical pixel size of 1.35 angstroms. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 687928 |
|---|---|
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4.0.16) |
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.4) / 使用した粒子像数: 67969 |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X