+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3812 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tomogram of e. coli carrying the ple7 plasmid carrying YFP-MreB hyper-overexpressed by induction with 1uM IPTG | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  電子線トモグラフィー法 電子線トモグラフィー法 | |||||||||
 データ登録者 データ登録者 | Swulius MT / Jensen GJ | |||||||||
 引用 引用 |  ジャーナル: J Bacteriol / 年: 2012 ジャーナル: J Bacteriol / 年: 2012タイトル: The helical MreB cytoskeleton in Escherichia coli MC1000/pLE7 is an artifact of the N-Terminal yellow fluorescent protein tag. 著者: Matthew T Swulius / Grant J Jensen /  要旨: Based on fluorescence microscopy, the actin homolog MreB has been thought to form extended helices surrounding the cytoplasm of rod-shaped bacterial cells. The presence of these and other putative ...Based on fluorescence microscopy, the actin homolog MreB has been thought to form extended helices surrounding the cytoplasm of rod-shaped bacterial cells. The presence of these and other putative helices has come to dominate models of bacterial cell shape regulation, chromosome segregation, polarity, and motility. Here we use electron cryotomography to show that MreB does in fact form extended helices and filaments in Escherichia coli when yellow fluorescent protein (YFP) is fused to its N terminus but native (untagged) MreB expressed to the same levels does not. In contrast, mCherry fused to an internal loop (MreB-RFP(SW)) does not induce helices. The helices are therefore an artifact of the placement of the fluorescent protein tag. YFP-MreB helices were also clearly distinguishable from the punctate, "patchy" localization patterns of MreB-RFP(SW), even by standard light microscopy. The many interpretations in the literature of such punctate patterns as helices should therefore be reconsidered. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3812.map.gz emd_3812.map.gz | 417.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3812-v30.xml emd-3812-v30.xml emd-3812.xml emd-3812.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3812.png emd_3812.png | 119.6 KB | ||
| その他 |  emd_3812_additional_1.map.gz emd_3812_additional_1.map.gz emd_3812_additional_2.map.gz emd_3812_additional_2.map.gz emd_3812_additional_3.map.gz emd_3812_additional_3.map.gz emd_3812_additional_4.map.gz emd_3812_additional_4.map.gz | 555.3 MB 517.2 MB 462.4 MB 630.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3812 http://ftp.pdbj.org/pub/emdb/structures/EMD-3812 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3812 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3812 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3810C  3811C C: 同じ文献を引用 ( |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10115 (タイトル: Tilt-series of e. coli carrying the ple7 plasmid carrying YFP-MreB hyper-overexpressed by induction with 1uM IPTG EMPIAR-10115 (タイトル: Tilt-series of e. coli carrying the ple7 plasmid carrying YFP-MreB hyper-overexpressed by induction with 1uM IPTGData size: 5.0 Data #1: Tilt-series for e. coli carrying the ple7 plasmid carrying YFP-MreB hyper-overexpressed by induction with 1uM IPTG [tilt series]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3812.map.gz / 形式: CCP4 / 大きさ: 984.4 MB / タイプ: IMAGE STORED AS SIGNED BYTE ダウンロード / ファイル: emd_3812.map.gz / 形式: CCP4 / 大きさ: 984.4 MB / タイプ: IMAGE STORED AS SIGNED BYTE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 9.46 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_3812_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
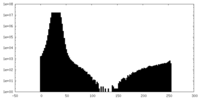
| 密度ヒストグラム |
-追加マップ: #2
| ファイル | emd_3812_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #3
| ファイル | emd_3812_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #4
| ファイル | emd_3812_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia Coli
| 全体 | 名称:   Escherichia Coli (大腸菌) Escherichia Coli (大腸菌) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia Coli
| 超分子 | 名称: Escherichia Coli / タイプ: cell / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #2: Helical MreB Cytoskeleton
| 超分子 | 名称: Helical MreB Cytoskeleton / タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
 解析 解析 |  電子線トモグラフィー法 電子線トモグラフィー法 |
|---|---|
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 構成要素 - 濃度: 50.0 ug/ml / 構成要素 - 式: C16H19N3O4S / 構成要素 - 名称: Ampicillin アンピシリン アンピシリン詳細: MC1000, MC1000/pLE6, and MC1000/pLE7 were grown in LB at 37C with 50ug/ml ampicillin when appropriate. |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 特殊光学系 | エネルギーフィルター - 名称: FEI |
| 温度 | 最高: 123.15 K |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サイズ - 横: 1016 pixel / デジタル化 - サイズ - 縦: 1016 pixel / 平均電子線量: 0.4 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | ソフトウェア - 名称:  IMOD IMODソフトウェア - 詳細: Tomograms were reconstructed and modelled 使用した粒子像数: 501 |
|---|
 ムービー
ムービー コントローラー
コントローラー



 Z
Z Y
Y X
X