+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3785 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Watermelon mosaic virus potyvirus. | |||||||||
 マップデータ マップデータ | Cryoelectron microscopy map of Watermelon mosaic virus filament. Resulting map from Relion postprocessing. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Potyvirus coat protein / Potyvirus coat protein /  カプシド / Coat protein カプシド / Coat protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Watermelon mosaic virus (ウイルス) Watermelon mosaic virus (ウイルス) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.0 Å クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Zamora M / Mendez-Lopez E / Agirrezabala X / Cuesta R / Lavin JL / Sanchez-Pina MA / Aranda M / Valle M | |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2017 ジャーナル: Sci Adv / 年: 2017タイトル: Potyvirus virion structure shows conserved protein fold and RNA binding site in ssRNA viruses. 著者: Miguel Zamora / Eduardo Méndez-López / Xabier Agirrezabala / Rebeca Cuesta / José L Lavín / M Amelia Sánchez-Pina / Miguel A Aranda / Mikel Valle /  要旨: Potyviruses constitute the second largest genus of plant viruses and cause important economic losses in a large variety of crops; however, the atomic structure of their particles remains unknown. ...Potyviruses constitute the second largest genus of plant viruses and cause important economic losses in a large variety of crops; however, the atomic structure of their particles remains unknown. Infective potyvirus virions are long flexuous filaments where coat protein (CP) subunits assemble in helical mode bound to a monopartite positive-sense single-stranded RNA [(+)ssRNA] genome. We present the cryo-electron microscopy (cryoEM) structure of the potyvirus watermelon mosaic virus at a resolution of 4.0 Å. The atomic model shows a conserved fold for the CPs of flexible filamentous plant viruses, including a universally conserved RNA binding pocket, which is a potential target for antiviral compounds. This conserved fold of the CP is widely distributed in eukaryotic viruses and is also shared by nucleoproteins of enveloped viruses with segmented (-)ssRNA (negative-sense ssRNA) genomes, including influenza viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3785.map.gz emd_3785.map.gz | 42.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3785-v30.xml emd-3785-v30.xml emd-3785.xml emd-3785.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
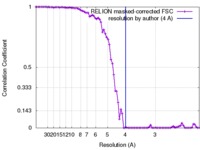
| FSC (解像度算出) |  emd_3785_fsc.xml emd_3785_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3785.png emd_3785.png | 245.2 KB | ||
| その他 |  emd_3785_additional.map.gz emd_3785_additional.map.gz emd_3785_half_map_1.map.gz emd_3785_half_map_1.map.gz emd_3785_half_map_2.map.gz emd_3785_half_map_2.map.gz | 17.4 MB 35.5 MB 35.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3785 http://ftp.pdbj.org/pub/emdb/structures/EMD-3785 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3785 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3785 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3785.map.gz / 形式: CCP4 / 大きさ: 46.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3785.map.gz / 形式: CCP4 / 大きさ: 46.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryoelectron microscopy map of Watermelon mosaic virus filament. Resulting map from Relion postprocessing. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Cryoelectron microscopy map of Watermelon mosaic virus filament....
| ファイル | emd_3785_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryoelectron microscopy map of Watermelon mosaic virus filament. Map generated by applying helical symmetry parameters to Relion postprocessing resulting map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
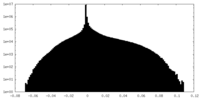
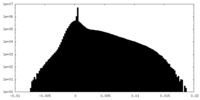
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half 1 map of Watermelon mosaic virus...
| ファイル | emd_3785_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half 1 map of Watermelon mosaic virus filament resulting from Relion 3D Refine. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
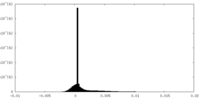
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half 2 map of Watermelon mosaic virus...
| ファイル | emd_3785_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half 2 map of Watermelon mosaic virus filament resulting from Relion 3D Refine. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
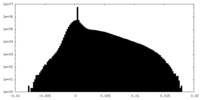
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Watermelon mosaic virus
| 全体 | 名称:   Watermelon mosaic virus (ウイルス) Watermelon mosaic virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Watermelon mosaic virus
| 超分子 | 名称: Watermelon mosaic virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 146500 / 生物種: Watermelon mosaic virus / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: coat protein
| 分子 | 名称: coat protein / タイプ: protein_or_peptide / ID: 1 詳細: 57 residues from N-terminus and 17 residues from C-terminus are not present in our pdb model due to the absence of densities for them in the cryo-electron microscopy map as a consequence of ...詳細: 57 residues from N-terminus and 17 residues from C-terminus are not present in our pdb model due to the absence of densities for them in the cryo-electron microscopy map as a consequence of being high flexible regions in the protein. コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Watermelon mosaic virus (ウイルス) Watermelon mosaic virus (ウイルス) |
| 分子量 | 理論値: 31.463412 KDa |
| 配列 | 文字列: SGKEAVENLD AGKESKKDTS GKGDKPQNSQ TGQGSKEQTK TGTVSKDVNV GSKGKEVPRL QKITKKMNLP TVGGKIILSL DHLLEYKPN QVDLFNTRAT KTQFESWYSA VKVEYDLNDE QMGVIMNGFM VWCIDNGTSP DVNGVWVMMD GEEQVEYPLK P IVENAKPT ...文字列: SGKEAVENLD AGKESKKDTS GKGDKPQNSQ TGQGSKEQTK TGTVSKDVNV GSKGKEVPRL QKITKKMNLP TVGGKIILSL DHLLEYKPN QVDLFNTRAT KTQFESWYSA VKVEYDLNDE QMGVIMNGFM VWCIDNGTSP DVNGVWVMMD GEEQVEYPLK P IVENAKPT LRQIMHHFSD AAEAYIEMRN SESPYMPRYG LLRNLRDREL ARYAFDFYEV TSKTPNRARE AIAQMKAAAL AG INSRLFG LDGNISTNSE NTERHTARDV NQNMHTLLGM GPPQ |
-分子 #2: RNA (5'-R(P*UP*UP*UP*UP*U)-3')
| 分子 | 名称: RNA (5'-R(P*UP*UP*UP*UP*U)-3') / タイプ: rna / ID: 2 / コピー数: 24 |
|---|---|
| 由来(天然) | 生物種:   Watermelon mosaic virus (ウイルス) Watermelon mosaic virus (ウイルス) |
| 分子量 | 理論値: 1.485872 KDa |
| 配列 | 文字列: UUUUU |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) デジタル化 - 画像ごとのフレーム数: 2-27 / 平均電子線量: 1.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X