+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3694 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ subtomogram average of Rubisco within the Chlamydomonas pyrenoid | |||||||||
 マップデータ マップデータ | In situ subtomogram average of Rubisco holoenzymes within the pyrenoid of Chlamydomonas reinhardtii. Filtered to 16%u212B. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Chlamydomonas reinhardtii (クラミドモナス) Chlamydomonas reinhardtii (クラミドモナス) | |||||||||
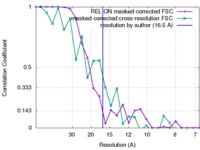
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 16.5 Å クライオ電子顕微鏡法 / 解像度: 16.5 Å | |||||||||
 データ登録者 データ登録者 | Cuellar LK / Schaffer M / Strauss M / Martinez-Sanchez A / Plitzko JM / Foerster F / Engel BD | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: The Eukaryotic CO-Concentrating Organelle Is Liquid-like and Exhibits Dynamic Reorganization. 著者: Elizabeth S Freeman Rosenzweig / Bin Xu / Luis Kuhn Cuellar / Antonio Martinez-Sanchez / Miroslava Schaffer / Mike Strauss / Heather N Cartwright / Pierre Ronceray / Jürgen M Plitzko / ...著者: Elizabeth S Freeman Rosenzweig / Bin Xu / Luis Kuhn Cuellar / Antonio Martinez-Sanchez / Miroslava Schaffer / Mike Strauss / Heather N Cartwright / Pierre Ronceray / Jürgen M Plitzko / Friedrich Förster / Ned S Wingreen / Benjamin D Engel / Luke C M Mackinder / Martin C Jonikas /   要旨: Approximately 30%-40% of global CO fixation occurs inside a non-membrane-bound organelle called the pyrenoid, which is found within the chloroplasts of most eukaryotic algae. The pyrenoid matrix is ...Approximately 30%-40% of global CO fixation occurs inside a non-membrane-bound organelle called the pyrenoid, which is found within the chloroplasts of most eukaryotic algae. The pyrenoid matrix is densely packed with the CO-fixing enzyme Rubisco and is thought to be a crystalline or amorphous solid. Here, we show that the pyrenoid matrix of the unicellular alga Chlamydomonas reinhardtii is not crystalline but behaves as a liquid that dissolves and condenses during cell division. Furthermore, we show that new pyrenoids are formed both by fission and de novo assembly. Our modeling predicts the existence of a "magic number" effect associated with special, highly stable heterocomplexes that influences phase separation in liquid-like organelles. This view of the pyrenoid matrix as a phase-separated compartment provides a paradigm for understanding its structure, biogenesis, and regulation. More broadly, our findings expand our understanding of the principles that govern the architecture and inheritance of liquid-like organelles. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3694.map.gz emd_3694.map.gz | 638.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3694-v30.xml emd-3694-v30.xml emd-3694.xml emd-3694.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3694_fsc_1.xml emd_3694_fsc_1.xml emd_3694_fsc_2.xml emd_3694_fsc_2.xml | 2.1 KB 2.4 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3694.png emd_3694.png | 66.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3694 http://ftp.pdbj.org/pub/emdb/structures/EMD-3694 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3694 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3694 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3694.map.gz / 形式: CCP4 / 大きさ: 686.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3694.map.gz / 形式: CCP4 / 大きさ: 686.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | In situ subtomogram average of Rubisco holoenzymes within the pyrenoid of Chlamydomonas reinhardtii. Filtered to 16%u212B. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.42 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : In situ Rubisco holoenzyme
| 全体 | 名称: In situ Rubisco holoenzyme |
|---|---|
| 要素 |
|
-超分子 #1: In situ Rubisco holoenzyme
| 超分子 | 名称: In situ Rubisco holoenzyme / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: In situ subtomogram average generated from Rubisco holoenzymes imaged within the native Chlamydomonas pyrenoid. Cells were thinned by focused ion beam milling. |
|---|---|
| 由来(天然) | 生物種:   Chlamydomonas reinhardtii (クラミドモナス) Chlamydomonas reinhardtii (クラミドモナス)株: mat3-4 |
| 分子量 | 理論値: 540 KDa |
-分子 #1: Rubisco holoenzyme
| 分子 | 名称: Rubisco holoenzyme / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO EC番号:  リブロース1,5-ビスリン酸カルボキシラーゼ/オキシゲナーゼ リブロース1,5-ビスリン酸カルボキシラーゼ/オキシゲナーゼ |
|---|---|
| 配列 | 文字列: MVPQTETKAG AGFKAGVKDY RLTYYTPDYV VRDTDILAAF RMTPQPGVPP EECGAAVAAE SSTGTWTTVW TDGLTSLDRY KGRCYDIEPV PGEDNQYIAY VAYPIDLFEE GSVTNMFTSI VGNVFGFKAL RALRLEDLRI PPAYVKTFVG PPHGIQVERD KLNKYGRGLL ...文字列: MVPQTETKAG AGFKAGVKDY RLTYYTPDYV VRDTDILAAF RMTPQPGVPP EECGAAVAAE SSTGTWTTVW TDGLTSLDRY KGRCYDIEPV PGEDNQYIAY VAYPIDLFEE GSVTNMFTSI VGNVFGFKAL RALRLEDLRI PPAYVKTFVG PPHGIQVERD KLNKYGRGLL GCTIKPKLGL SAKNYGRAVY ECLRGGLDFT KDDENVNSQP FMRWRDRFLF VAEAIYKAQA ETGEVKGHYL NATAGTCEEM MKRAVCAKEL GVPIIMHDYL TGGFTANTSL AIYCRDNGLL LHIHRAMHAV IDRQRNHGIH FRVLAKALRM SGGDHLHSGT VVGKLEGERE VTLGFVDLMR DDYVEKDRSR GIYFTQDWCS MPGVMPVASG GIHVWHMPAL VEIFGDDACL QFGGGTLGHP WGNAPGAAAN RVALEACTQA RNEGRDLARE GGDVIRSACK WSPELAAACE VWKEIKFEFD TIDKL |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV 詳細: Blotted for 10 seconds with 10 blot force before plunging.. |
| 詳細 | Rubisco holoenzymes within the native Chlamydomonas pyrenoid. Whole cells were plunge-frozen onto EM grids and then thinned with a focused ion beam instrument. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 6.457 µm / 最小 デフォーカス(補正後): 5.077 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 42000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 42000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 1 / 平均露光時間: 1.5 sec. / 平均電子線量: 1.5 e/Å2 詳細: Images were collected in movie mode at 17 frames per second |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー