+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3603 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | KaiCB circadian clock backbone model based on a Cryo-EM density | |||||||||
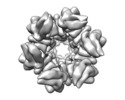
 マップデータ マップデータ | Circadian Clock Complex KaiBC | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of phosphorelay signal transduction system / negative regulation of circadian rhythm / entrainment of circadian clock / negative regulation of phosphorylation / protein serine/threonine/tyrosine kinase activity /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 /  regulation of circadian rhythm / regulation of circadian rhythm /  概日リズム / 概日リズム /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  リン酸化 ...regulation of phosphorelay signal transduction system / negative regulation of circadian rhythm / entrainment of circadian clock / negative regulation of phosphorylation / protein serine/threonine/tyrosine kinase activity / リン酸化 ...regulation of phosphorelay signal transduction system / negative regulation of circadian rhythm / entrainment of circadian clock / negative regulation of phosphorylation / protein serine/threonine/tyrosine kinase activity /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 /  regulation of circadian rhythm / regulation of circadian rhythm /  概日リズム / 概日リズム /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  リン酸化 / protein serine kinase activity / protein serine/threonine kinase activity / regulation of DNA-templated transcription / magnesium ion binding / リン酸化 / protein serine kinase activity / protein serine/threonine kinase activity / regulation of DNA-templated transcription / magnesium ion binding /  ATP hydrolysis activity / ATP hydrolysis activity /  DNA binding / DNA binding /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Synechococcus elongatus (バクテリア) Synechococcus elongatus (バクテリア) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.0 Å クライオ電子顕微鏡法 / 解像度: 7.0 Å | |||||||||
 データ登録者 データ登録者 | Schuller JM / Snijder J / Loessl P / Heck AJR / Foerster F | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2017 ジャーナル: Science / 年: 2017タイトル: Structures of the cyanobacterial circadian oscillator frozen in a fully assembled state. 著者: Joost Snijder / Jan M Schuller / Anika Wiegard / Philip Lössl / Nicolas Schmelling / Ilka M Axmann / Jürgen M Plitzko / Friedrich Förster / Albert J R Heck /   要旨: Cyanobacteria have a robust circadian oscillator, known as the Kai system. Reconstituted from the purified protein components KaiC, KaiB, and KaiA, it can tick autonomously in the presence of ...Cyanobacteria have a robust circadian oscillator, known as the Kai system. Reconstituted from the purified protein components KaiC, KaiB, and KaiA, it can tick autonomously in the presence of adenosine 5'-triphosphate (ATP). The KaiC hexamers enter a natural 24-hour reaction cycle of autophosphorylation and assembly with KaiB and KaiA in numerous diverse forms. We describe the preparation of stoichiometrically well-defined assemblies of KaiCB and KaiCBA, as monitored by native mass spectrometry, allowing for a structural characterization by single-particle cryo-electron microscopy and mass spectrometry. Our data reveal details of the interactions between the Kai proteins and provide a structural basis to understand periodic assembly of the protein oscillator. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3603.map.gz emd_3603.map.gz | 2.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3603-v30.xml emd-3603-v30.xml emd-3603.xml emd-3603.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3603.png emd_3603.png | 73.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3603 http://ftp.pdbj.org/pub/emdb/structures/EMD-3603 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3603 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3603 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3603.map.gz / 形式: CCP4 / 大きさ: 28.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3603.map.gz / 形式: CCP4 / 大きさ: 28.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Circadian Clock Complex KaiBC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : KaiCB circadian clock complex
| 全体 | 名称: KaiCB circadian clock complex |
|---|---|
| 要素 |
|
-超分子 #1: KaiCB circadian clock complex
| 超分子 | 名称: KaiCB circadian clock complex / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Synechococcus elongatus (バクテリア) Synechococcus elongatus (バクテリア) |
| 組換発現 | 生物種:   Synechococcus elongatus (バクテリア) Synechococcus elongatus (バクテリア) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 15.2 sec. / 平均電子線量: 45.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4) |
|---|---|
| 初期モデル | モデルのタイプ: INSILICO MODEL In silico モデル: 3D Shape with the approximate demensions of the 2D Classes |
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 7.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 5137 |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー